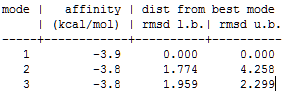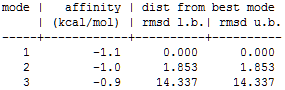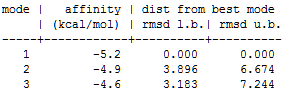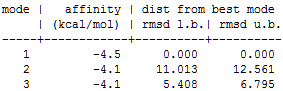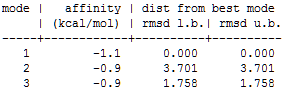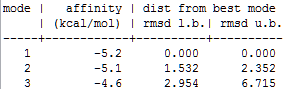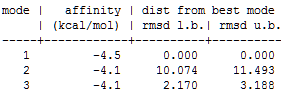Докинг низкомолекулярных лигандов в структуру белка
1) В банке pdb находится SMILES для NAG: nag.smi.
2) C помощью obgen построена 3D структура этого сахара в pdb формате: nag.pdb.
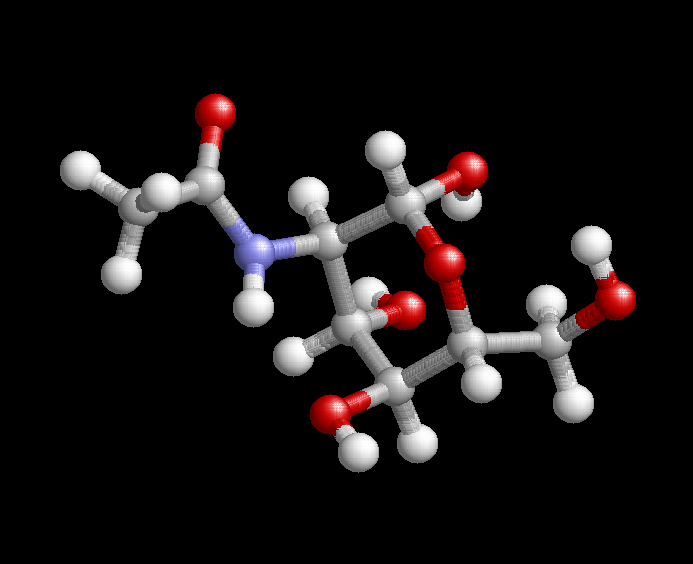
3) Cоздаем pdbqt файл лиганда: nag.pdbqt.
4) Скриптом prepare_receptor4.py создадим pdbqt файл белка: seq4.pdbqt.
5) Теперь надо создать файл с параметрами докинга vina.cfg.
6) Проведем первый докинг с фиксированными аминокислотами белка и подвижным лигандом. Запустим команду vina --config vina.cfg --receptor seq4.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
На выходе имеем два файла nag_prot.pdbqt, содержащий информацию о положениях лиганда, и nag_prot.log, содержащий
информацию о энергиях каждой модели.
Ниже на рисунке представлены все состояния лиганда (картинка получена с помощью Pymol).
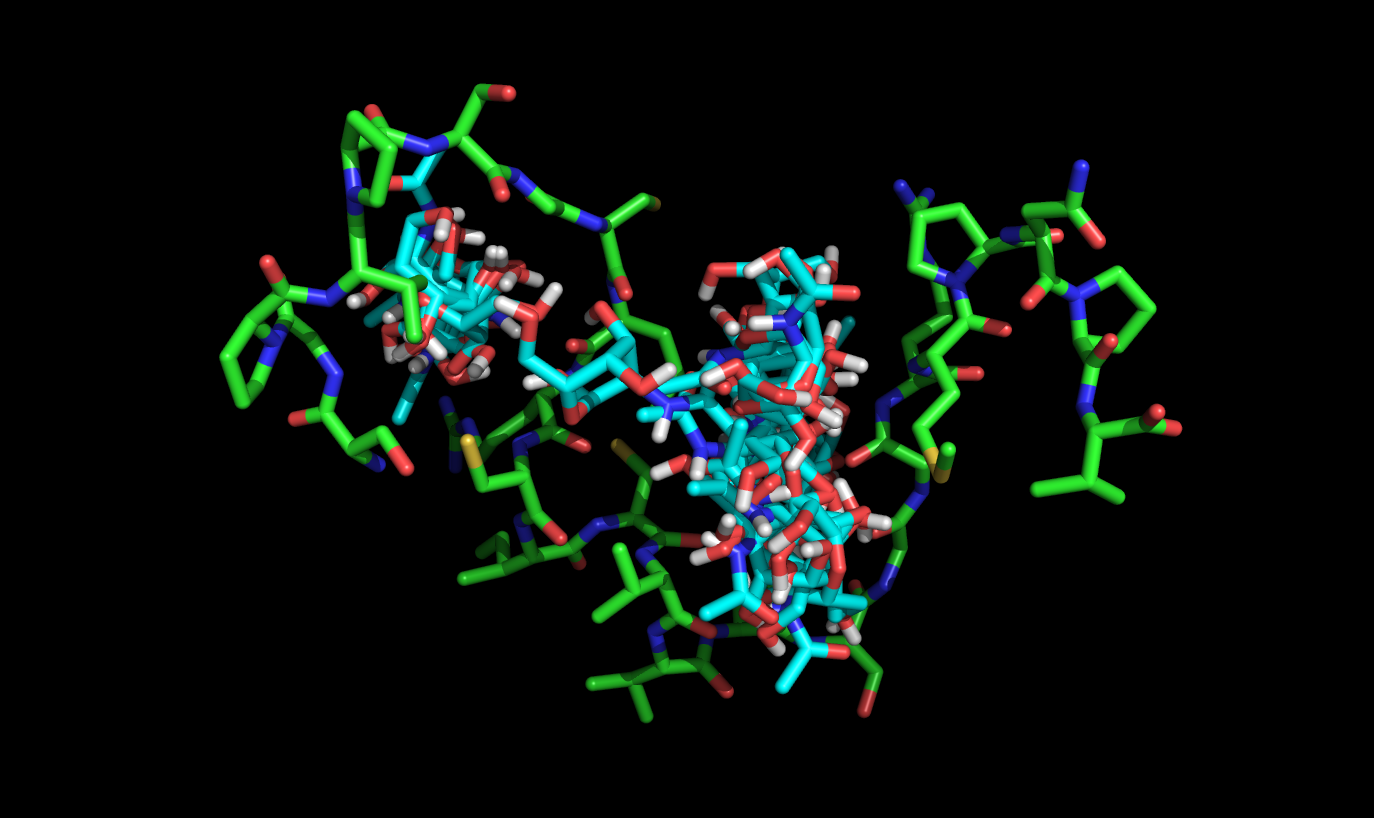
Теперь выберем 3 наиболее выгодных энергетически состояния:
Как видно, даже вдали от нативного центра энергия все равно высокая, это еще один результат того, что мой белок представлен лишь 25-ю а.к.
7) Теперь давайте проведём докинг, рассматривая подвижность некоторых боковых радикалов белка. Сначала разобьем белок на две части, подвижную и неподвижную.
Задаем подвижную и константную часть при помощи команды python /usr/share/pyshared/AutoDockTools/Utilities24/prepare_flexreceptor4.py -r seq4.pdbqt -s ASN23 (т.к. мой белок состоит лишь из 25 аминоксилот, поэтому и в прошлом и в этом занятии была выбрана лишь одна аминокислота - ASN23).
Теперь моделируем. Получаем такие состояния:
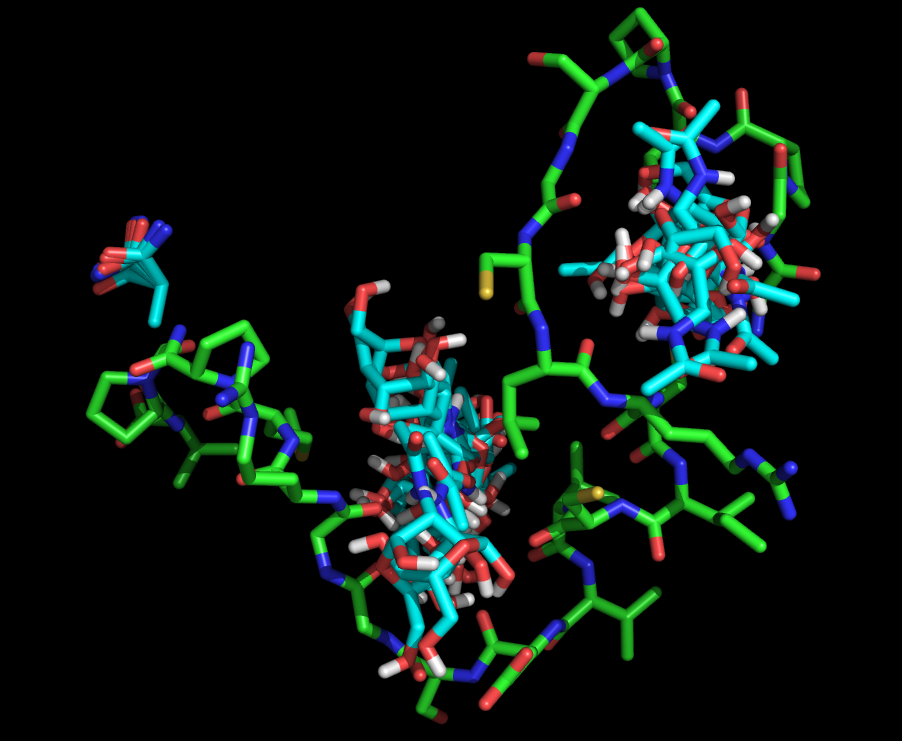
В результате докинга были получены файлы vina_prot_flex.pdbqt и vina_prot_flex.log.
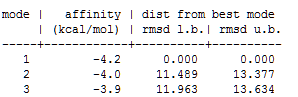
8) Просмотрим файл vina_prot_flex.log. Энергии трех лучших расположений и геометрическая разница между ними представлена в таблице:

9) Можно сделать вывод, что докинг не смог расположить лиганд наиболее близким образом к тому, что мы получили в моделировании.
10) Вся работа была проведена для лигандов где метильный радикал СH3C(=O)NH группы заменён на:
Все файлы представлены в директории Term6/11.
a) Для каждого из этих лигандов был проведен обыкновенный докинг.Результаты в виде таблицы из трёх лучших расположений для каждого лиганда:
-H
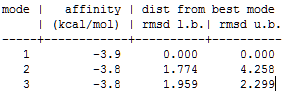
-OH
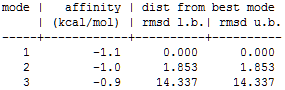
-PH
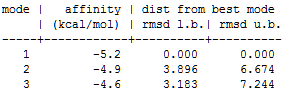
-NH2
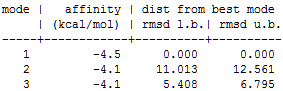
б) Докинг с подвижными радикалами для новых 3 лигандов (кроме Н):
Получили такие энергии:
-OH
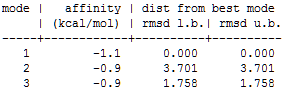
-PH
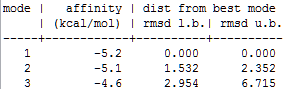
-NH2
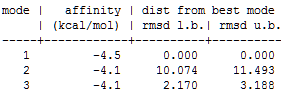
Как видно, наибольшую афинность имеет лиганд с Ph, он и помещается наиболее близко к белку.
© Шерстюк Александра, MSU 2009