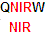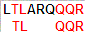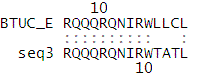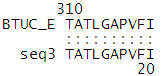|
|
Факультет Биоинженерии и биоинформатики |

Глобальное
и локальное выравнивание аминокислотных последовательностей
Работа с программами GeneDoc, Excel, пакетом
EMBOSS: Needle, Water и Matcher |

Матрица
переходов.
Параметры для построения матрицы переходов:
вес совпадения = 2, вес замены = -1, штраф за делецию = -2.

Seq1:
QNIRW
Seq2: NIR |
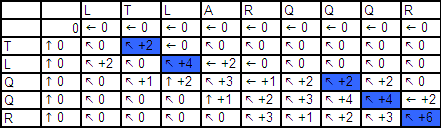
Построение
матрицы глобального выравнивания
Цветом
отмечен путь оптимального пути перехода.
Вес выранивания: 2
Выравнивание: 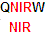
|
Seq1:
LTLARQQQR
Seq2: TLQQR |
Построение матрицы локального выравнивания.
Оранжевым цветом отмечен путь оптимального пути перехода.
Вес
выранивания: 6
Вес субоптимального выравнивания: 4
Выравнивание: 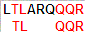
|

Поиск участков
локальной гомологии
|
| Последовательности,
для которых при помощи программы Matcher из пакета Emboss
было построено локальное выравнивание в формате Fasta:
|
>BtuC_Ecoli
MLTLARQQQRQNIRWLLCLSVLMLLALLLSLCAGEQWISPGDWFTPRGELFVWQIRLPRT
LAVLLVGAALAISGAVMQALFENPLAEPGLLGVSNGAGVGLIAAVLLGQGQLPNWALGLC
AIAGALIITLILLRFARRHLSTSRLLLAGVALGIICSALMTWAIYFSTSVDLRQLMYWMM
GGFGGVDWRQSWLMLALIPVLLWICCQSRPMNMLALGEISARQLGLPLWFWRNVLVAATG
WMVGVSVALAGAIGFIGLVIPHILRLCGLTDHRVLLPGCALAGASALLLADIVARLALAA
AELPIGVVTATLGAPVFIWLLLKAGR
>Seq3
RQQQRQNIRWTATLGAPVFI

Координаты
участков выравниваний в белке BtuC: 6-19
Координаты
участков выравниваний в белке BtuC: 309-318 |
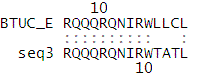
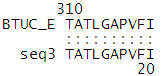
|
Влияние параметров на глобальное выравнивание
С помощью программы needle были построены глобальные выравнивания для тех же последовательностей, что и в части 2, но в одном случае использовалась матрица BLOSUM62 и штраф заоткрытие делеции 10, а во втором случае - BLOSUM80 и штраф - 1.
Выравнивание для которого использовалась матрица BLOSUM62 
|
Выравнивание для которого использовалась матрица BLOSUM80 
|
Видно, что выравнивания довольно сильно различаются. В первом случае имеет место только одна делеция, а во втором - 7. Это связана со стоимостью штрафа за открытие делеции.
На главную
Второй семестр

© 2004. ФББ МГУ им. М.В.Ломоносова
|
|