Buchnera aphidicola - представитель грамм-отрицательных бактерий, ведущий эндосимбионтный образ жизни. Их хозяевами являются гороховые тли. Подобный образ жизни привёл к утрате некоторых генов. В этом практикуме будет решаться задача сборки de novo генома этой бактерии из одноконцевых чтений, полученных по технологии Illumina.
| Команда | Комментарий |
| wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/009/SRR4240359/SRR4240359.fastq.gz | Скачиваем чтения для работы |
| cat ../../adapters/*-SE.fa >> adapters.fasta | Создаём файл с адаптерам Illumina для одноконцевых чтений |
| java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240359.fastq.gz SRR4240359_noadapt.fastq ILLUMINACLIP:adapters.fasta:2:7:7 | Выполняем чистку чтений от возможных остатков адаптеров |
| java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240359_noadapt.fastq SRR4240359_trimmed.fastq TRAILING:20 MINLEN:32 | Удаляем с концов чтений нуклеотиды качества ниже 20 и оставляем чтения длины не менее 32 нуклеотидов |
| velveth ./velv 31 -fastq -short SRR4240359_trimmed.fastq | Подготавливаем к-меры для построения графов |
| velvetg ./velv | Построение графов де Брёйна и сборка контигов |
Поток ошибок команд сохранялся в качестве лог-файлов.
Чтения были проверены на качество с помощью fastqc. Сравнение результатов анализа исходных чтений, чтений после очистки от остатков адаптеров и удаления нуклеотидов плохого качества говорит о том, что данные процедуры привели к значительному улучшению качества наших чтений. Остатками адаптеров оказалась 0,41% исходных чтений. По причинам плохого качества или небольшой длины было удалено 1308515 (9.69%) чтений, оставшихся после триммирования остатков адаптеров. До этой очистки вес файла составлял 1.4G, после — 1.2G.
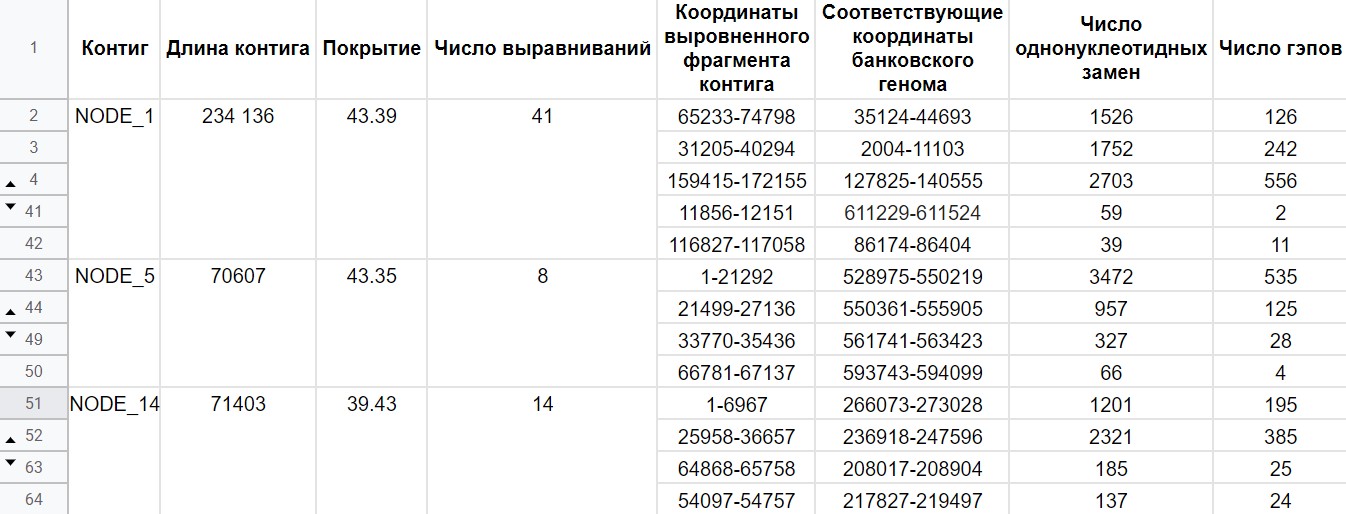
После окончания работы velvetg узнаём, что для нашей сборки N50 составляет 70607. Самыми длинными контигами оказались NODE_1 (234136) с покрытием 43.4, NODE_5 (70607) с покрытием 43.35 и NODE_14 (71403) с покрытием 39.4. Среди контигов попались два, имеющие очень высокое покрытие: один из них имееют длину 47 и покрытие 139.5, другой — длину 40 и покрытие 109.5.
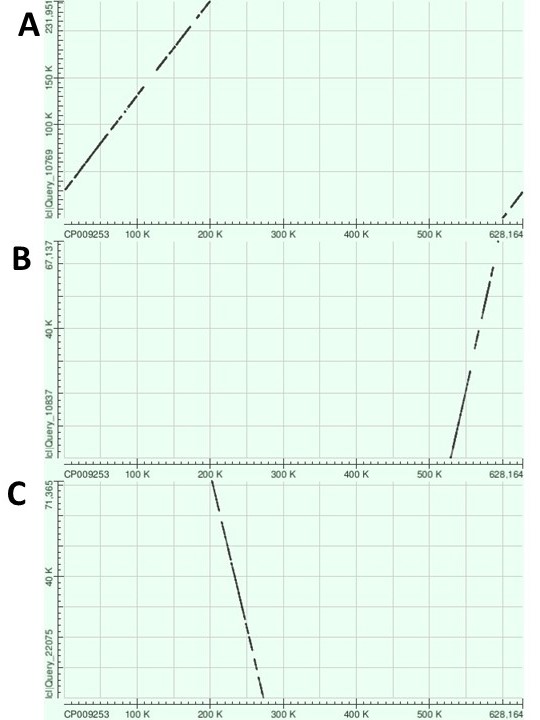
Полученные самые длинные контиги были проанализированны с помощью megablast (Рис.1). Контиг NODE_1 ложится на начало записи из банка, но претерпевает разрыв, так как за начало кольцевой хромосомы бактерии авторы выбрали другой регион. Контиг NODE_5 выравнивается с концом геномной записи. Контиг NODE_14 инвертирован по отношению к геномной записи из банка и располагается ближе к её середине. Контиги выранивались не целиком, а в виде фрагментов (Табл.1). В целом, выравнивания получились очень хорошими, однако присутствует некоторое количество разрывов на графиках, что может говорить о наличии неконсервативных регионов в геноме.