
В этом задании был проведён поиск консервативного мотива в выранивании последовательностей гомологичных белков. Для этого в база данных Pfam был найден домен Rcd1. Белки этого семейства участвуют в процессах клеточной дифференцировки в качестве транскрипционных кофакторов. Далее было скачано и открыто в Jalview выравнивание seed, которое включало 52 последовательности.
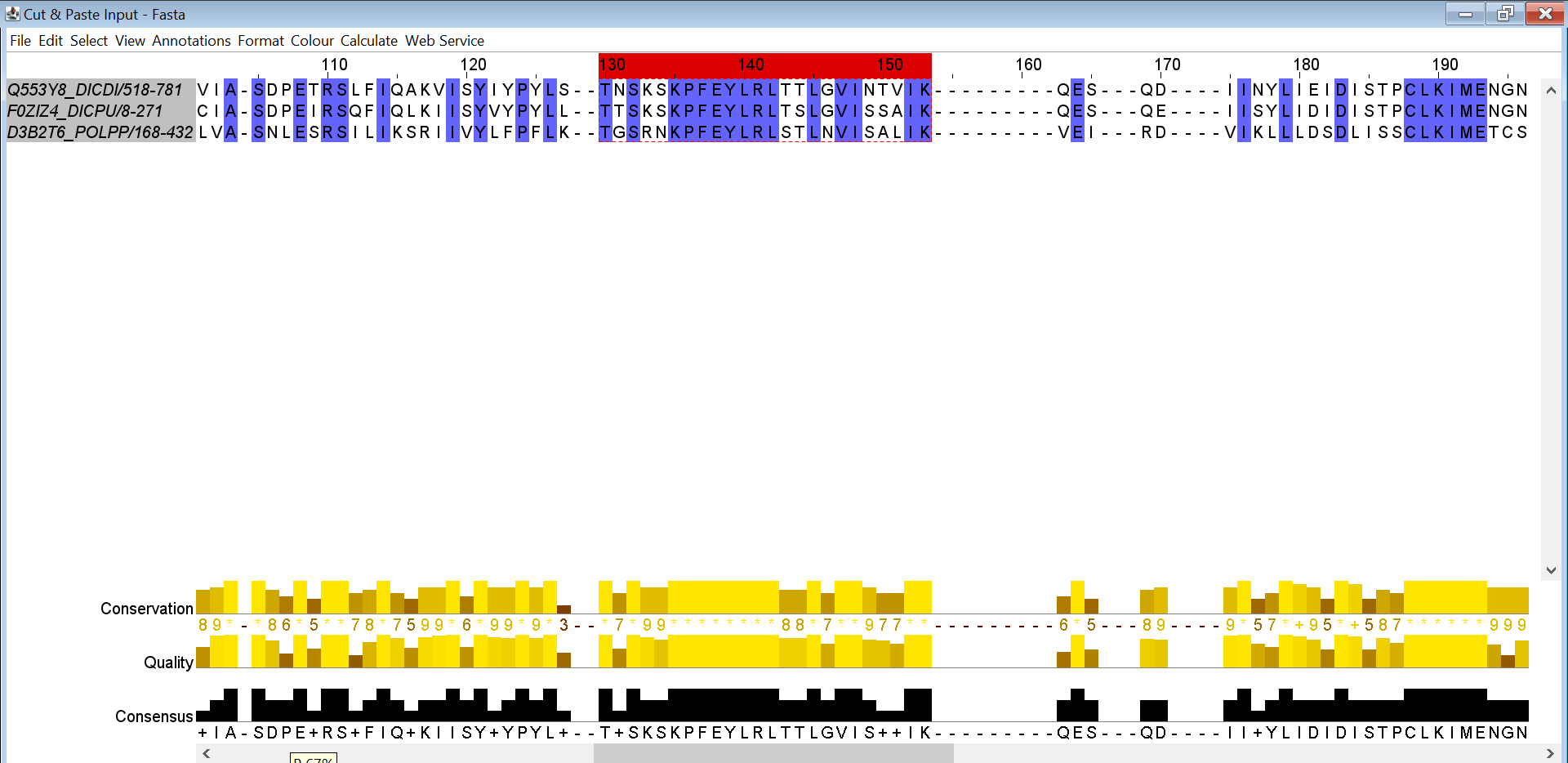
При покраске с порогом идентичности в 100% оказались выделены только отдельные столбцы. Поэтому порог был понижен до 80%, и между двумя полностью идентичными столбцами можно было увидеть мотив (Рис.1). По этому мотиву можно составить паттерн: E.L[RK]LTSLGVIGALVK. Несмотря на то, что мотив был записан достаточно строго и имел большую длину, во всём выравнивании нашлось по нему 28 последовательностей. То есть у этого мотива действительно высокое информационное содержание, и поиск с ним был продолжен на сервисе MyHits по базе SwissProt. Нашлось 11 совпадений, причём все они были из взятого семейства. Находки были в основном среди хордовых (данио рерио, пара земноводных, млекопитающие, в том числе и человек), но было и две находки среди грибов.

На следующем этапе было построено филогенетическое дерево по алгоритму NJ и выбрана клада, объединяющая трёх организмов. Выравнивание последовательностей из этих организмов было выделено в отдельной окно, и выполнена раскраска по проценту идентичночти (Рис.2). Так как последовальтельностей всего три, порог в 100% кажется лучшим вариантом. Здесь рассматриеваемый ранее мотив можно расширить: T.S[KR].KPFEYLRL[TS]{2}L.VI.{3}IK. Поиск по нему среди всех последовательностей выравнивания даёт ожидаемые три последовательности. Думаю, можно сделать вывод, что удалось найти признак, подтверждающий правильность выбранной ветви.
Результаты выполнения задания также можно увидеть в файле.
В этом задании был опробован PSI-BLAST. Был выбран белок с AC: Q7VDL2. Он принадлежит бактерии Prochlorococcus marinus (strain SARG / CCMP1375 / SS120) и является ингибитором клеточного деления, нарушающий сборку колец белка FtsZ, предшествующую делению.
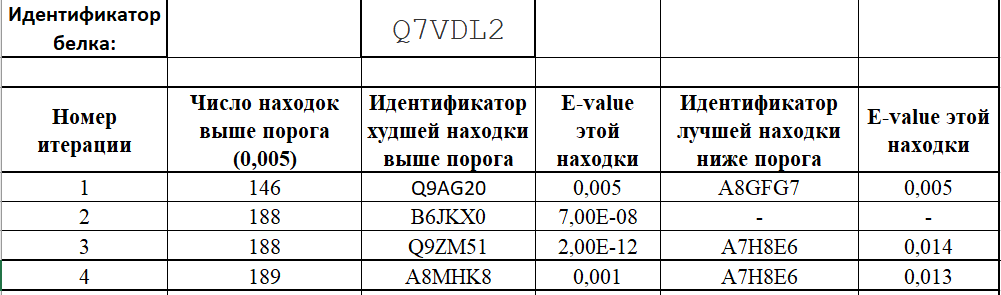
Можно заметить, что уже на второй итерации оформилось семейство, а вот на 4 итерации уже добавился посторонний белок, обладающий совершенно другой функцией. В пользу того, что это "ложноположительный результат", говорит и малое различие E-value между ним и находкой ниже порога.
Узнаем, отличается ли число TA от ожидаемого по статистике. Ожидаемое число ТА составляет 99020. В хромосоме Rubrobacter marinus strain SCSIO 52915, которая имеет длину 4156452 п.н., ТА обнаружено 69584. Это составляет только 70% от ожидаемого.