Практиум 12
1. Множественное выравнивание с использованием разных программ.
При выполнении этого задания я использовала белки из парктикума 9: PBPA_CLOPE, PBPA_NEIGO, PBPA_MYCTU, PBPA_AQUAE. Выравнивание было выполнено в Jalview с помощью Clustl и Muscle.
Выравнивание с помощью Clustl Выравнивание с помощью MuscleДля сравнения этих двух выравниваний я использовала программу Матвея Киселёва, Егора Малахова, Артема Тюкаева и Якоба Коробицина, тк мне очень понравился визуально понятный и компактный вывод.
Со временем программа усовершенствовалась, но я приведу и старый, и новый вывод.
Первоначальный вывод программы:
1-16 17-603 ------ 604-642 643-650 ------- 651-682 683-686 ------- 687-689 690-691 ------- 692-704 705-712 ------- 713-733 734-742 ------- 743-785 786-821 ------- 822-853
1-16 ------ 17-644 645-683 ------- 684-692 693-724 ------- 725-728 729-731 ------- 732-733 734-746 ------- 747-754 755-775 ------- 776-784 785-827 ------- 828-863 864-895
| Clusstl | Muscle |
|---|---|
| 1-16 | 1-16 |
| 604-642 | 645-683 |
| 651-682 | 693-724 |
| 687-689 | 729-731 |
| 692-704 | 734-746 |
| 713-733 | 755-775 |
| 743-785 | 785-827 |
| 822-853 | 864-895 |
Исходя из выводных данных можно сказать, что выравнивание этих двух программ хоть и не совпадают на 90 или 100%, но результаты достаточно близки.
2. Множественное выравнивсание 3D структур.
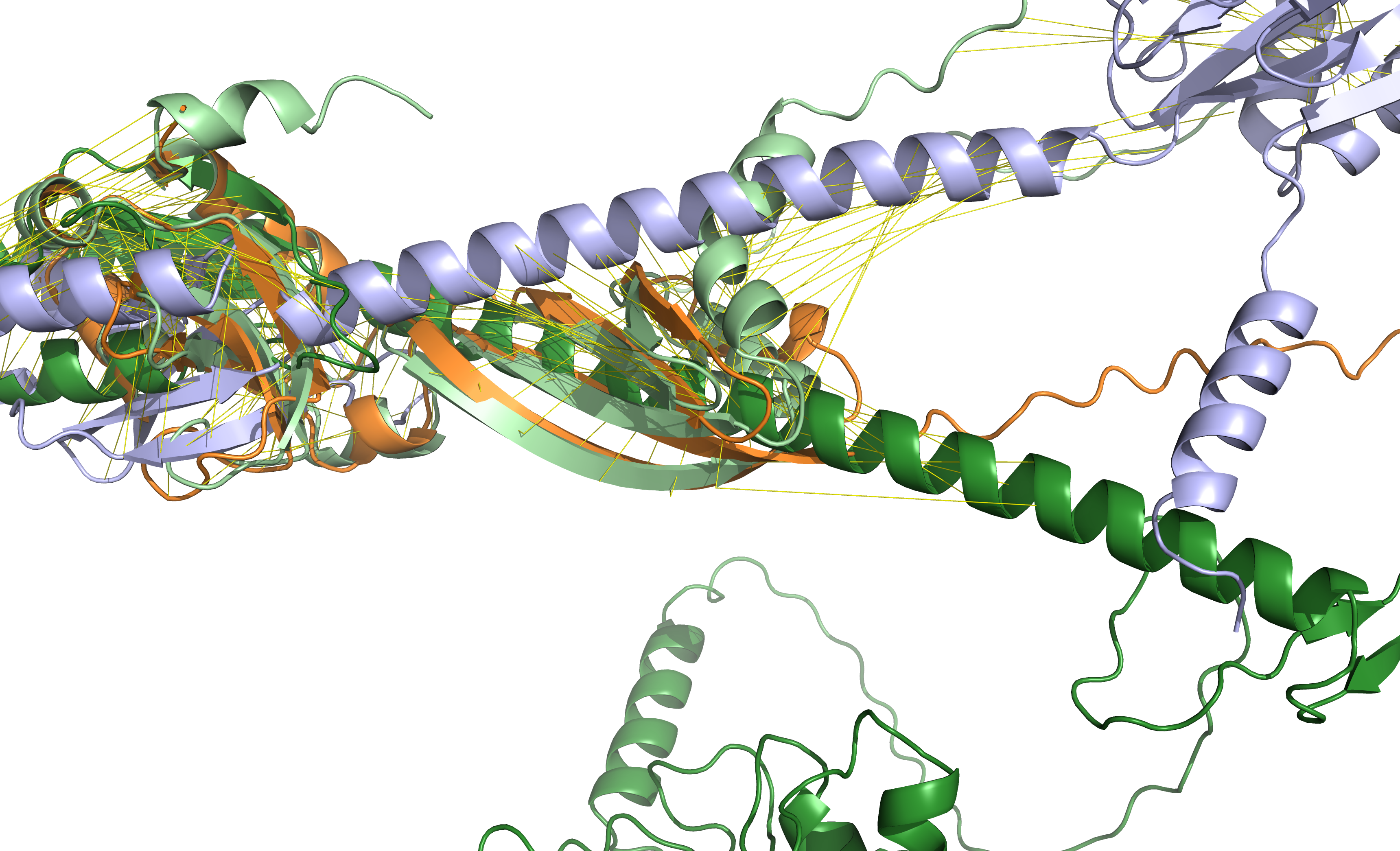
При выполенении этого задания была использованна программа PyMol с предварительным скачиванием 3D структур с UniProt ( на этом сайте они взяты с сайта Alpha-fold). Для выравнивания я выбрала семейство из практикума 11: Имя: BofC C-terminal domain; Архитектуры: 1-L0EG14_THECK; 2-C5D5F0_GEOSW; 3-R6H908_9ACTN; 4-W5IHW0_SCAIO.
Хотя из предыдущего практикума было видно, что выранвнивание этих белков не очень хорошее, было все же инетренсо посмотреть на результат. Ниже я представела блок выравнивание для 1 белка: 46-81; 2: 23-87; 3: 253-293; 4-382-420. Понимаю, что это не самый гомологичный блок, но по моему мнению он наиболее наглядный для понимания работы PyMol (action: align).
3. Описание программы MUSCLE.
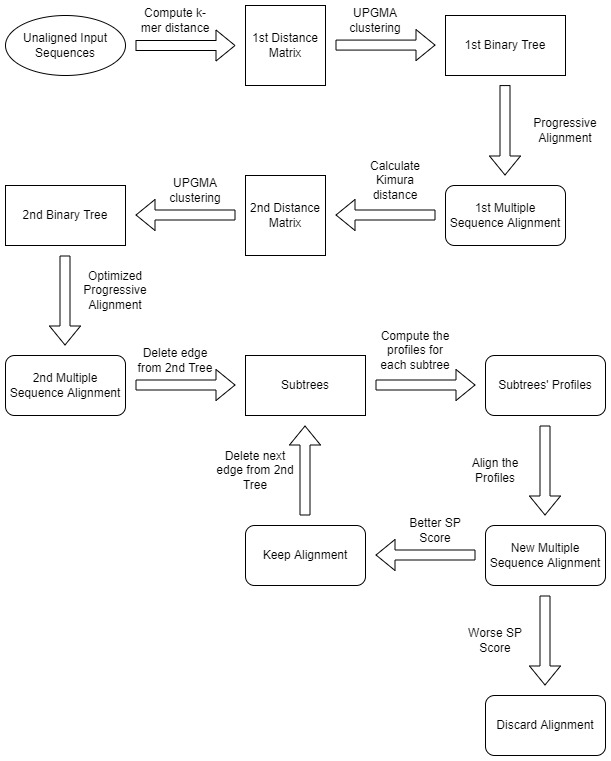
MUSCLE (MUltiple Sequence Comparison by Log- Expectation)
Это компьютерная программа для выравнивания множественных последовательностей белковых и нуклеотидных последовательностейэто компьютерная программа для выравнивания множественных последовательностей белковых и нуклеотидных последовательностей, которая использует cравнение множественных последовательностей с помощью логарифмического ожидания.
Метод был опубликован Робертом К. Эдгаром в двух статьях в 2004 году.
Алгоритм MUSCLE выполняется в три этапа:
- Этап 1: Прогрессивный проект (скоростное множественное выравнивнаия, но точноть страдает при этом).
- Этап 2: Усовершенствованный прогрессивный проект (более точное выравнивание благодаря проведению опреций в поддеревьях, порядок ветвления которых изменился по сравнению с первым двоичным деревом).
- Этап 3: Совершенствование(Процесс удаления ребра и выравнивания повторяется до сближения или пока не будет достигнут определенный пользователем предел). Muscle, Wokipedia