Карты локального сходства
Задание 1. Выбор короткого контига и различные варианты BLAST.
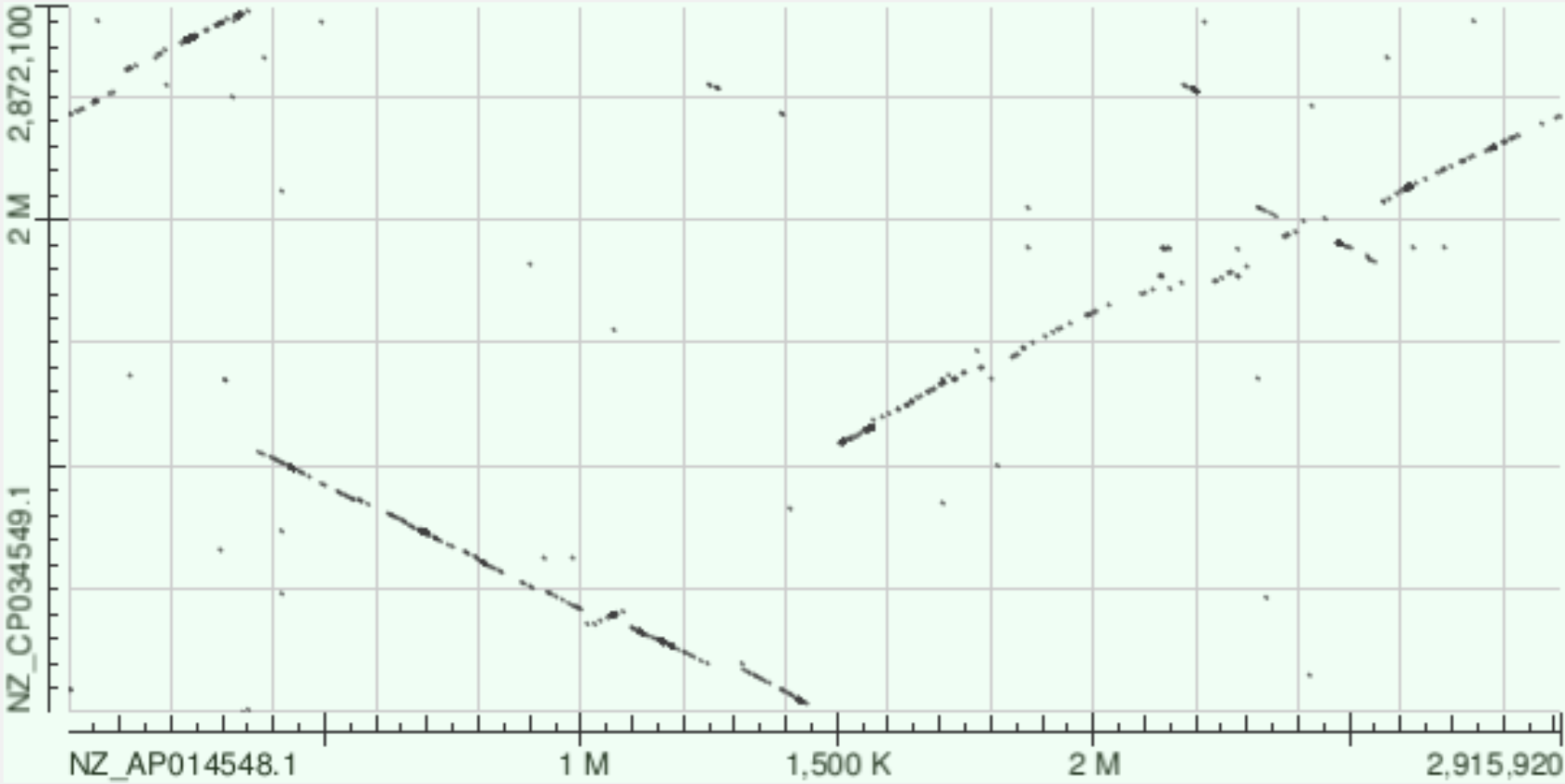
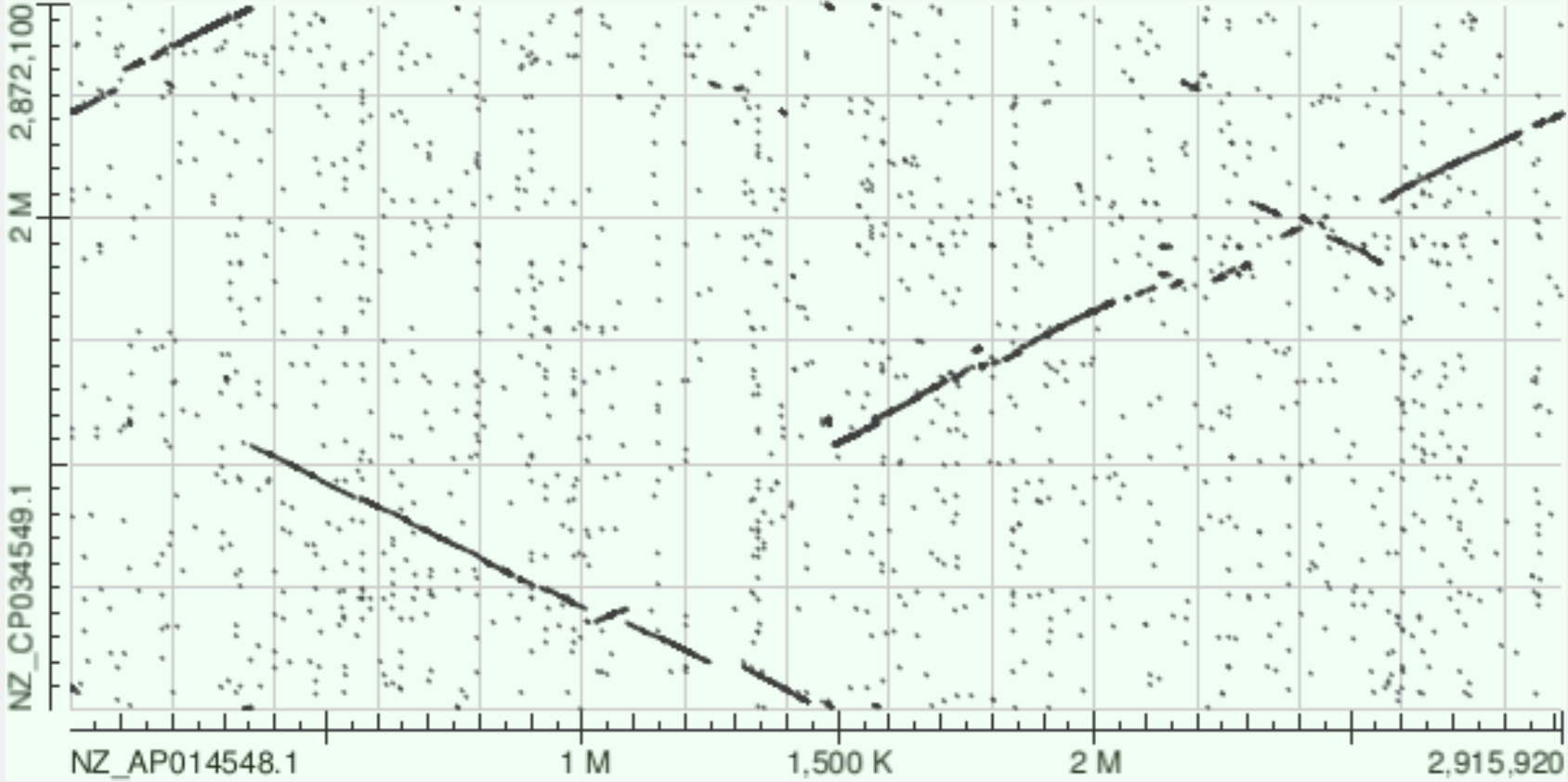
Для выполнения задания я выбрала двух фотогетеротрофных грамотрицательных бактерий семейства Flavobacteriaceae - Nonlabens marinus (NZ_AP014548.1) и Nonlabens ponticola (NZ_CP034549.1). Поиск производился в базе данных NCBI Nucleotide по запросу "Nonlabens" с указанием "chromosome" в "Title". Анализируются хромосомы. Ниже приведены два Dot Plot, построенные разными алгоритмами BLAST:
Как можно заметить, blastn оказался чувствительнее megablast.
Участок [0-400.000]bp указывает на разные ориджины - точки начала репликации - хромосом: они не совпадают.
Участки [400.000-1.500.000]bp и [2.400.000-2.600.000]bp - инверсии (можно заметить, что внутри есть инвертированные участки
поменьше - они либо были подвержены повторной инверсии, либо оставались в такой ориентации всё время и первичная инверсия
их не затронула). На [1.250.000-1.350.000]bp есть делеция (разрыв в прямой).