Практиум 8
Задание 1. Выбор короткого контига и различные варианты BLAST.
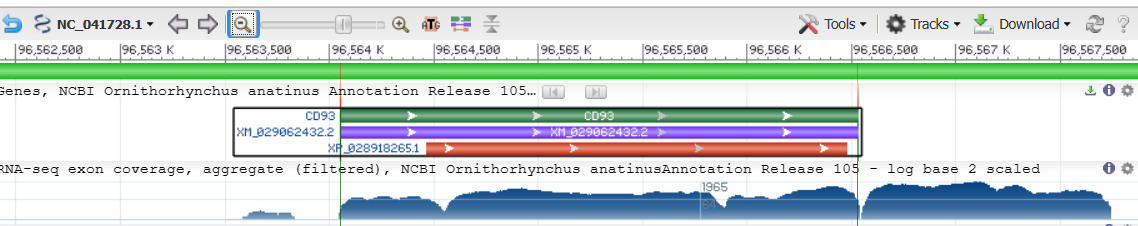
Для работы была выбран контиг хромосомы 1 Ornithorhynchus anatinus с индентификатором NW_041728.1 с длиной 5549 bp. На этом участке расположен ген CD93, он помочен зеленым, красным цветом обозначен CDS, а фиолеовым - соответствующая мРНК. Длина кодирующей последовательности, те красного участка - 2022 нуклеотида.
1. Blastn
Для чего нужен: чтобы определить принадлежность орагнизма к таксону, а также, чтобы сравнивать геномы не близкородственных организмов./p
Работа с Blastn: я решила сравнить, выбранный мой ген утконос с генами млекопитающих, для этого я поставила фильтр mammals (taxid:40674). Находок было 95/100, были представлено много различных представителей.
2. Megablast
Для чего нужен: Осуществляет быстрый поиск среди очень похожих последовательностей.
Работа с Megablast: Запрос и цель были такими же, как и в первом случае, а вот результат отличался : 99/100 и преобладали очень сильно 2 оргнаизма - утконос и ехидна.
3. Blastx
Для чего нужен: Определение кодирующих последовательностей.
Работа с Blastx: Запрос и цель были такими же, как и в первом случае, а вот результат отличался : 100/100 ( я решила изменить количество выдаваемых результатов до 500 и получила 304/500) очень разнообразные находки, соеди которых только 1 утконос.
4. Tblastx
Для чего нужен: Можно использовать для предсказания генов в последовательностях.
Работа с Blastx: Все попытки смягчить параметры или изменить запрос увенчались неудачей.
Задание 2. Поиск в геноме эукариота генов основных рибосомальных РНК по далекому гомологу.
Сначала при помощи команды makeblastdb -in GCF_004115215.2_mOrnAna1.pri.v4_genomic.fna -dbtype nucl была создана база данных на основе генома моего организма.
Нам были даны зЗРЛ 16S и 23S E.coli (16S рРНК входит в состав малой субъединицы, 23S рРНК в состав большой субъединицы прокариотической рибосомы)
Для выполнения задачи был использован blastn, так как исходная последовательность не является белок-кодирующей, а виды не близкие.
Использованные команды:
blastn -task blastn -evalue 0.05 -query 16S_rRNA.txt -db rawr.fna -out blast_rRNA166.txt -outfmt 7
blastn -task blastn -evalue 0.05 -query 28S_rRNA.txt -db rawr.fna -out blast_rRNA233.txt -outfmt 7
Параметр -evalue установлен, чтобы убрать плохие находки, параметр -outfmt для удобной выдачи в виде таблицы.
В результате первого бласта, при сравнение с 16S рРНК не было найдено совпадений. В результате сравнения с 23S рРНК было найдено 13 совпадений.
NC_041732.1 (86533792-86533963) входит в некодирующую длиную рнк.
NC_000891.1 (2330-2513) входит в митохондриальный геном в 16S рРНК.
NC_041735.1 (53594235-53594052) входит в некодирующую длиную рнк.
Все остальные находки являются неаннотированными.