Практиум 1
Задание 1.
Отобранные бактерии
| Название | Мнемоника |
|---|---|
| Rhizobium meliloti | RHIME |
| Roseobacter denitrificans | ROSDO |
| Saccharophagus degradans | SACD2 |
| Serratia proteamaculans | SERP5 |
| Shewanella denitrificans | SHEDO |
| Thiobacillus denitrificans | THIDA |
| Yersinia pestis | YERPE |
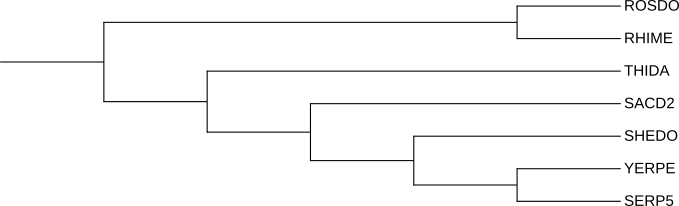
Скобочная формула
(((((SERP5,YERPE), SHEDO), SACD2), THIDA), (RHIME, ROSDO));
Изображение дерева
Ветви дерева
Дерево содержит 4 нейтральные ветви:
{SERP5, YERPE} против {SHEDO, SACD2, THIDA, RHIME, ROSDO}
{SERP5, YERPE, SHEDO} против {SACD2, THIDA, RHIME, ROSDO}
{SERP5, YERPE, SHEDO, SACD2} против {THIDA, RHIME, ROSDO}
{SERP5, YERPE, SHEDO, SACD2, THIDA} против {RHIME, ROSDO}
Практикум 2
Задание 1.
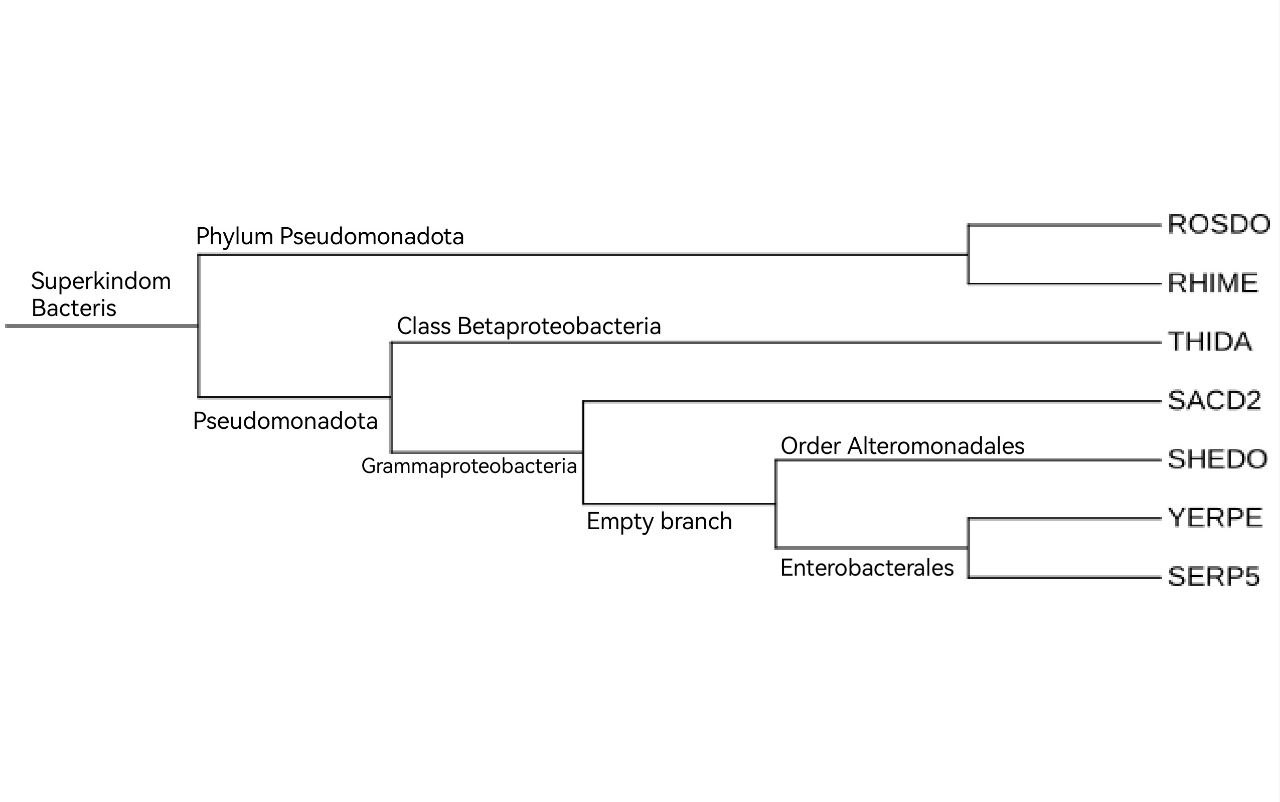
Построим таблицу с полной таксономии всех выбранных бактерий, а затем подпишим все нетривиальные ветви нашего дерева (Дерево было построено в практикуме 1).
| Название | Таксономия |
|---|---|
| Rhizobium meliloti (RHIME) | cellular organisms; Bacteria; Pseudomonadota; Alphaproteobacteria; Hyphomicrobiales; Rhizobiaceae; Sinorhizobium/Ensifer group; Sinorhizobium |
| Roseobacter denitrificans (ROSDO) | cellular organisms; Bacteria; Pseudomonadota; Alphaproteobacteria; Rhodobacterales; Roseobacteraceae; Roseobacter |
| Saccharophagus degradans (SACD2) | cellular organisms; Bacteria; Pseudomonadota; Gammaproteobacteria; Cellvibrionales; Cellvibrionaceae; Saccharophagus |
| Serratia proteamaculans (SERP5) | cellular organisms; Bacteria; Pseudomonadota; Gammaproteobacteria; Enterobacterales; Yersiniaceae; Serratia |
| Shewanella denitrificans (SHEDO) | cellular organisms; Bacteria; Pseudomonadota; Gammaproteobacteria; Alteromonadales; Shewanellaceae; Shewanella |
| Thiobacillus denitrificans (THIDA) | cellular organisms; Bacteria; Pseudomonadota; Betaproteobacteria; Nitrosomonadales; Thiobacillaceae; Thiobacillus |
| Yersinia pestis (YERPE) | cellular organisms; Bacteria; Pseudomonadota; Gammaproteobacteria; Enterobacterales; Yersiniaceae; Yersinia; Yersinia pseudotuberculosis complex |
В дереве присутствует пустая ветвь, SHEDO, YERPE и SERP5 имеют разные порядки.
Задание 2.
Для реконструкции филогенетического дерева была взята функция белка RL21 (один из белков большой субъединицы рибосомы). Из Uniprot были получены белковые последовательности
На сайте NGPhylogeny.fr была проведена реконструкция деревьем тремя алгоритмами: FastME (минимальная эволюция), TNT (максимал>
Практикум 2
Задание 1. Укоренение с использованием внешней группы
Задание 2. Бутсреп
Задание 3. Построение дерева по нуклеотидным последовательностям