Практиум 4
Задание 1. Составление списка гомологичных белков, включающих паралоги.
Из данной дериктории /P/y22/term4/Proteomes, я скачала все фаста файлы моих 7 белков. Далее среди них мы искали достоверные гомологи белка CLPX_ECOLI. В результате работы BLAST мы получили 26 гомологов, которые я сохранила сюда. Выдача бласта туть
Задание 2. Реконструкция и визуализация.
Файл с ноходками гомологов, которым мы будем пользоваться далее.
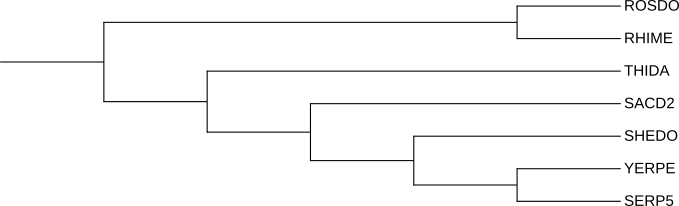
Дерево найденных гомологов было реконструированно программой FastME c параметрами: 'Gamma distributed rates across sites' — No, 'Starting tree' — BIONJ, 'No refinement', 100 бутстреп реплик.
Полученная форма Newick.
По реконструированному дереву находим три пары ортологов(а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования) и три пары парологов (Два гомологичных белка из одного организма).
Ортологи: CLPX_SERP5 и CLPX_YYERPPE, CLPX_RHIME и CLPX_ROSDO, HSLU_RHIME и HSLU_ROSDO.
Парологи: A0A5P8YB42 YERPE и A0A5P8YCE6 YERPE, Q12QI8 SHEDO и HSLU_SHEDO, A8G901_SERP5 и CLPX_SERP5.
Сравненивая реконструированные филогенией ортологические группы с филогенией бактерий можно заметить одиннаковыую тенденцию во всех ортологических группах, а именно: YERPE SERP5 против SHEDO против SACD2 против THIDA против ROSDO и RHIME, что сведетельствует о правильной филогении.