Практиум 5
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке.
1.1 Выбор белка
Название: Интимин (Intimin)
Индификатор PDB: 4e1s
Индификатор Uniprot: отсутствует
Организм из которого был взят белок: Escherichia coli
Локализация: внешняя мембрана грам отрицательной бактреии
Функции: Intimin является фактором вирулентности (адгезином) EPEC (Энтеропатогенная кишечная палочка) и EHEC (Энтерогеморрагическая Кишечная палочка) штаммов E. co> Это прикрепляющий и стирающий (A/ E) белок, который вместе с другими факторами вирулентности необходим и ответственен за энтеропатогенную (водянистая диарея, используют интимин для связывания с клетками кишечника хозяина) и энтерогеморрагическую диарею (кровянистая или бескровная диарея).
1.2 Координаты трансмембранных участков белка.
9 - TM segments: 1( 211- 219), 2( 226- 234), 3( 242- 251), 4( 256- 264), 5( 272- 279), 6( 287- 296), 7( 300- 308), 8( 329- 336), 9( 347- 353),10( 372- 380),11( 387- 392),12( 401- 408)
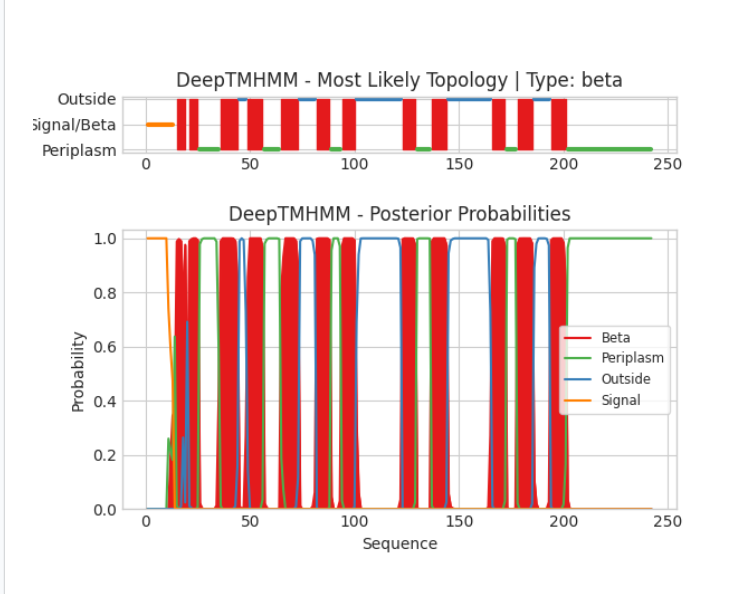
1.3 DeepTMHMM.
Со страницы PDB этого белка была получена его последовательность, которую я подала на вход DeepTMHMM.
Выдача DEEPTMHMM в формате gff
1(15, 19)
2(21, 25)
3(36, 44)
4(49, 56)
5(65, 73)
6(82, 88)
7(94, 100)
8(123, 129)
9(137, 144)
10(166, 172)
11(178, 185)
12(194, 201)
Исходя из вывода программы, можно сказать, что количество трансмембранных β-листов DEEPTMHMM предсказал верно, а вот координаты очень отличаются.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке.
2.1 Выбор белка
Название: комплекс переносчиков спермидина abc (Spermidine/putrescine transport system permease protein PotB homolog)
Индификатор SwissProt: POTB_MYCPN
Организм: Mycoplasma pneumoniae
Функции: Необходимы для деятельности бактериальной транспортной системы путресцина и спермидина
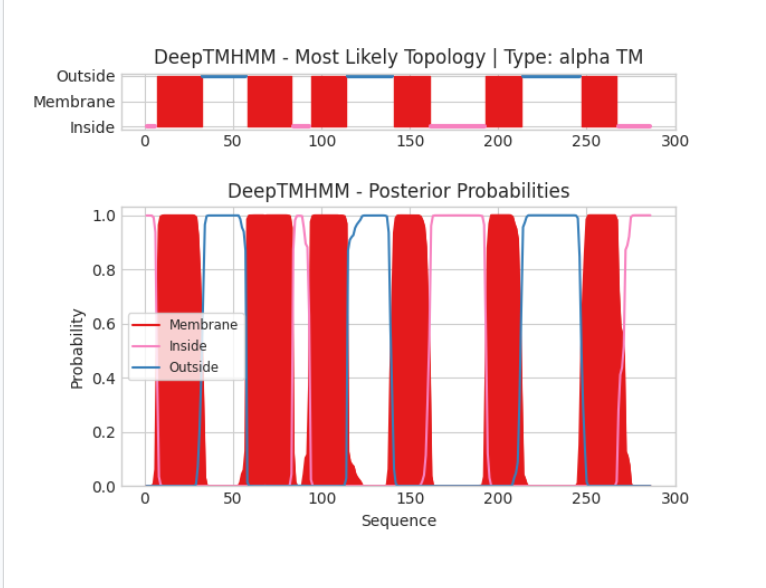
Выдача текстовая в формате gff.
1(7, 32)
2(58, 83)
3(94, 114)
4(141, 161)
5(193, 213)
6(247, 267)
Программа DEEPTMHHM нашла 6 альфа-спиралей.
Чтобы получить описание трансмембранных участков из AlphaFold, нашла в UniProte раздел про структуры и скачала оттуда файл с предсказанной структорой. Параметры для работы с программой PMM 3.0.:
тип: трансмембранные белки
количество мемюран: 1
тип мембраны: внещняя мембрана грам-положительной бактерии (исходя из информации в Uniprot и википедии)
разрешаемый изгиб: нет (этот параметр решила никак ре менять)
топология N-конца: in (исходя из рисунков и инофрмации на википедия и UniProt, N-конец распложен в цитоплазме)
Описание трасмембранных участков:
1(7, 33)
2(54, 83)
3(91, 118)
4(138, 159)
5(191, 213)
6(246, 272)
В этот раз предсказание более точное, количество спиралей совпало в обоих случайях, координаты отличаются совсем немного, что уже успех.