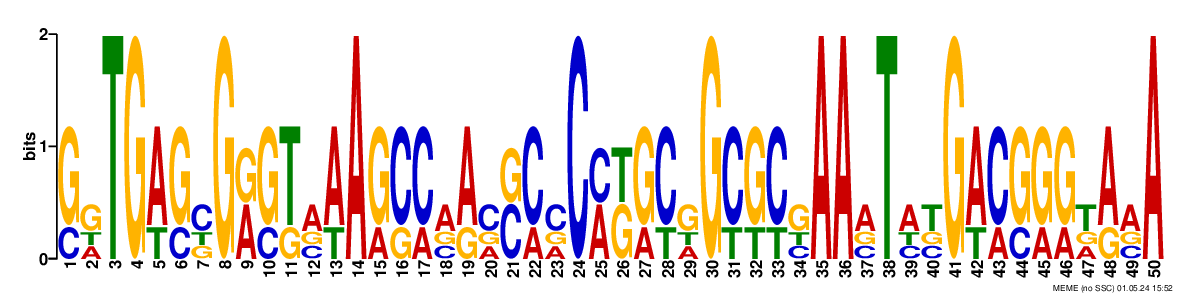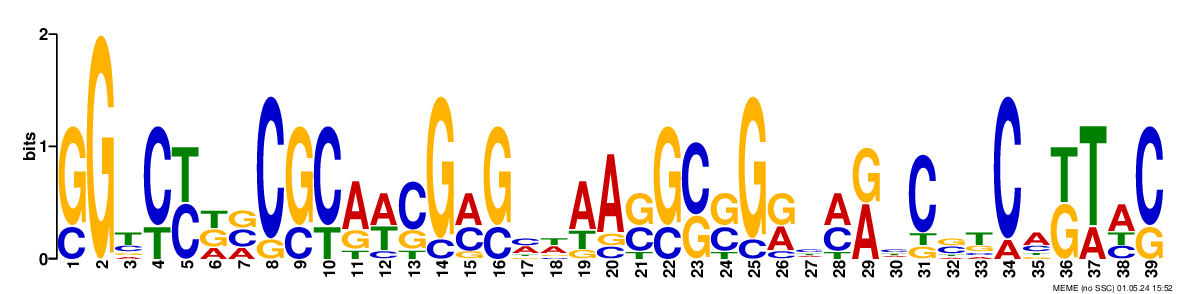Практиум 9
Поиск de novo сигналов в ДНК.
Введение.
Для выполнения данного практикума я использовала бактерию , по которой делала мини обзор на первом курсе. Из NCBI я скачала ее геном в формате fna и txt, c помощью сервиса Operon-mapper получила список оперонов. Для дальнейшей работы я использовала скрипт Георгия Муравьева, который генерирует 3 выборки: обучения, тестирования и отрицательного контроля, используя генном бактерии и список оперонов. Уточнение: данный скрипт считает промотором область в 100 нуклеотидов.
Запуск MEME.
Запуск программы MEME я осуществляла на kodomo при помощи команды:
meme housekeeping2.fasta -dna -nmotifs 3 -minw 6
Таким образом, программа выдала три лого и отчет о программе, включающий значение E-value.
Поиск сигнала с помощью FIMO.
Попробуем найти первый мотив, тк у самое маленькое E-value.
Для запуска FIMO я использовала выборки положительного (promotors2.fasta, был получен с помощью скрипта) и отрицательного (negativ2.fasta, аналогично) контроля.
fimo --norc -motif ACNCWGCCGCCGCAGGCGGTNATG -thresh 0.001 ./meme_out/meme.txt ./promotors.fasta
fimo --norc -motif ACNCWGCCGCCGCAGGCGGTNATG -thresh 0.001 ./meme_out/meme.txt ./negative.fasta
В результате я получила 2 таблицы с находками. В группе с отрицательном контролем программа нашла мотив в 91 последовательностях внутри гена, а в положительном контроле в 653 промоторах. Я считаю, это очень хороший результат, тк различия между находками почти в 7 раз.