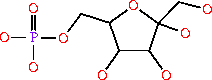
Функции, продолжение. Ферменты и метаболические пути
- Объяснение кода фермента USHA_ECOLI
Был дан белок: G6PI_ECOLI
EC-код: 5.3.1.9
Расшифровка кода:
EC 5 Isomerases (изомеразы , катализируют внутримолекулярные геометрические или структурные изменения. Проводят реакции, в ходе которых один изомер переходит в другой. )
EC 5.3 Intramolecular Oxidoreductases (внутримолекулярные оксидоредуктазы - ферменты, катализирующие лежащие в основе биологического окисления реакции, сопровождающиеся переносом электронов с восстановителя на окислитель)
EC 5.3.1 Interconverting Aldoses and Ketoses (взаимопревращения альдоз и кетоз)
EC 5.3.1.9 glucose-6-phosphate isomerase (глюкозо-6-фосфат-изомереза - катализ внутримолекулярных структурных перестроек глюкозо-6-фосфата)
Reaction: D-glucose 6-phosphate = D-fructose 6-phosphate
=
D-glucose 6-phosphate ------------------->D-fructose 6-phosphateТак же катализирует реакцию аномеризации D-глюкозо-6-фосфата.
Определение метаболических путей, в которых участвует фермент.
Имя локуса гена белка G6PI_ECOLI - b4025. В БД KEGG проведем поиск гена по названию его локуса.
Найденные метаболические пути:
eco00010 Glycolysis/Gluconeogenesis - Гликолиз / ГлюконеогенезКарта пути гликолиз/глюконеогенез
eco00030 Pentose phosphate pathway - Пентозофосфатный путь
eco00500 Starch and sucrose metabolism - Метаболизм крахмала и сахарозы
eco00520 Amino sugar and nucleotide sugar metabolism - Метаболизм аминосахаров и фосфорных эфиров нуклеозидов
eco01100 Metabolic pathways - Метаболические путиПоиск в KEGG структурных формул дезоксицитидина и тимина
Откроем страницу оглавления KEGG. Найдем ссылку на базу данных химических соединений (KEGG LIGAND). Проведем поиск по каждому из заданных веществ.
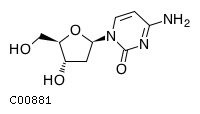
дезоксицитидин
Английское название: deoxycytidine Идентификатор соединения: C00881
Структурная формула:
тимин
Английские название: Pyruvate
Идентификатор соединения: C00178
Структурная формула: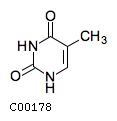
Метаболический путь от дезоксицитидина к тимину
В БД KEGG Pathway был использован инструмент "Color objects in pathways".
Выбранная цепочка ферментативных реакций:
путь: метаболизм пиримидина
цепочка: дезоксицитидин ----> тиминБыл выбран наиболее короткий путь. В данном случае возможен путь только от дезоксицитидина к тимину, но не наоборот. Красный цвет - начало пути - дезоксицитидин, желтым - промежуточные соединения, зеленый - конец пути - тимин.
Карта метаболического пути между дезоксицитидином и тимином
- Сравните метаболические пути у разных организмов
Был проведен поиск полученной цепочки ферментативных реакций пируват → глицерат и глицерат → пируват в различных организмах. Результаты представлены в виде таблицы:
Возможность цепочки: пируват → глицерат и глицерат → пируват - в разных организмах с известными полными геномами.
Организм Возможна ли цепочка реакций
(да/нет/неизвестно)Обоснование Escherichia coli K-12 MG1655 да присутствуют все ферменты, необходимые для осуществления цепочки реакций Archaeoglobus fulgidus нет неизвестен ген фермента, катализирующего 1-ю стадию (EC 2.7.1.74) Arabidopsis thaliana нет неизвестен ген фермента, катализирующего 5-ю стадию (EC 2.4.2.6) Homo sapiens да присутствуют все ферменты, необходимые для осуществления цепочки реакций
- Сравните ферменты из далеких организмов
- Был задан EC-код: 2.7.1.36
С помощью одного запроса к SRS нашли ферменты с заданным EC-кодом у человека и археи Archaeoglobus fulgidus (окончание имени "_ARCFU").
Опция использования маски (Use wildcards) была снята, чтобы находились только ферменты с кодом 2.7.1.36, а не 2.7.1.36*, так как теоретически возможны порядковые номера вида 36X, где X - цифра.
([uniprot-ECNumber:2.7.1.36*] & ([uniprot-ID:*_human*] | [uniprot-ID:*_ARCFU*]))Нашлось 2 белка, один из которых принадлежат археям, а один - человеку: KIME_ARCFU и KIME_HUMANСравнение доменной организации найденных белков
идентификатор белка доменная организация координаты доменов KIME_ARCFU GHMP_kinases_N 78 - 145
GHMP_kinases_C 182 - 274KIME_HUMAN GHMP_kinases_N 130 - 212
GHMP_kinases_C 290 - 366Поиск ортологов
KIME_ARCFU:
Name: mvk Ordered Locus Names: AF_2289
Лучший ортолог для белка архей KIME_ARCFU -rcu:RCOM_1431350 из организма Ricinus communis - Клещевина обыкновенная.
Процент совпадения: 33% Длина выравнивания: 388 аминокислотных остатка.
Лучший ортолог для человеческого белка KIME_HUMAN -tpe:Tpen_0891 из организма Thermofilum pendens.
Процент совпадения: 32,3% Длина выравнивания: 386 аминокислотных остатка.
Несмотря на дальность организмов, оба ортолога сохранили ферментативную функцию исходных белков (EC:2.7.1.36 ).
©2008-2010 Михальченко Алексей