Реконструкция и сравнение деревьев.
Расстояния между последовательностями
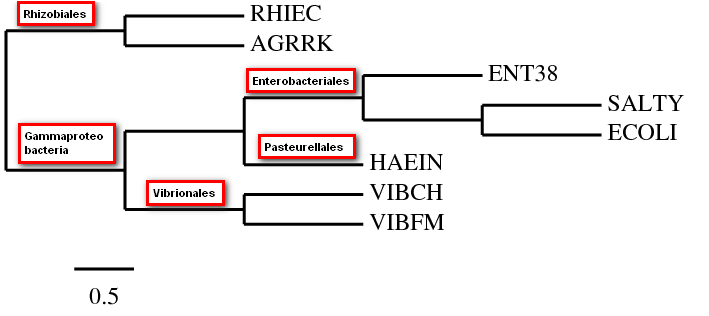
- Пользуясь таксономическим сервисом NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/, определим таксоны выбранных бактерий.
Мнемоника Таксономия RHIEC Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Rhizobium AGRRK Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Agrobacterium ENT38 Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Enterobacter SALTY Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Salmonella; Salmonella enterica; Salmonella enterica subsp. enterica ECOLI Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Escherichia HAEIN Proteobacteria; Gammaproteobacteria; Pasteurellales; Pasteurellaceae; Haemophilus VIBCH Proteobacteria; Gammaproteobacteria; Vibrionales; Vibrionaceae; Vibrio VIBFM Proteobacteria; Gammaproteobacteria; Vibrionales; Vibrionaceae; Aliivibrio
- Из списка функций белков выберите одну: по белкам соответствующего семейства вы будете реконструировать филогенетическое дерево.
Функция Мнемоника Омега-субъединица ДНК-зависимой РНК-полимеразы RPOZ Фактор элонгации трансляции Ts EFTS Рибосомный белок L2 RL2 Рибосомный белок S7 RS7 Рибосомный белок S12 RS12 Энолаза ENO Была выбрана функция "рибосомный белок L2". Из Swiss-Prot получили последовательности белков с данной функцией из отобранных бактерий (bact_l2func.fasta).
- Создайте выравнивание отобранных белков программой muscle или mafft
Выровняли полученные последовательности программой muscle (bact_l2func_aln.fasta).
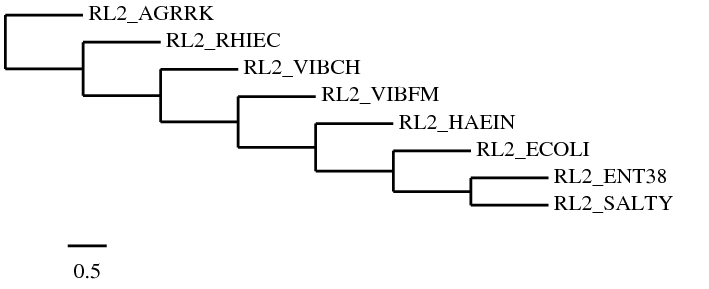
- Проведите реконструкцию дерева программой fprotpars. Сколько деревьев выдала программа? Приведите в отчёте скобочную формулу(ы) и изображение(я).
В программе fprotpars реализован метод максимальной бережливости (maximal parsimony). Программа выдаёт неукоренённое дерево без длин ветвей.
По полученному выравниванию программа построила три дерева.
(((((((RL2_SALTY,RL2_ENT38),RL2_ECOLI),RL2_HAEIN),RL2_VIBFM),RL2_VIBCH),RL2_RHIEC),RL2_AGRRK);
(((((RL2_ENT38,(RL2_SALTY,RL2_ECOLI)),RL2_HAEIN),(RL2_VIBFM,RL2_VIBCH)),RL2_RHIEC),RL2_AGRRK);
((((((RL2_SALTY,RL2_ENT38),RL2_ECOLI),RL2_HAEIN),(RL2_VIBFM,RL2_VIBCH)),RL2_RHIEC),RL2_AGRRK);
Как можно заметить, второе дерево совпадает с правильным (если учесть, что оно неукорененное).
- Оцените эволюционные расстояния между последовательностями программой fprotdist. Приведите в отчёте матрицу расстояний.
Насколько расстояния отклоняются от ультраметричности? Насколько расстояния отклоняются от аддитивности?
Матрица расстояний, построенная при помощи программы fprotdist:
AGRRK 0.000000 0.096299 0.573690 0.602874 0.648022 0.606975 0.601354 0.611322Посмотрим отклонения расстояний от ультраметричночти и аддитивности.
RHIEC 0.096299 0.000000 0.580740 0.610869 0.651251 0.601889 0.596308 0.606229
VIBCH 0.573690 0.580740 0.000000 0.100915 0.221927 0.175894 0.178876 0.175618
VIBFM 0.602874 0.610869 0.100915 0.000000 0.210635 0.207125 0.216195 0.206797
HAEIN 0.648022 0.651251 0.221927 0.210635 0.000000 0.158884 0.162592 0.161981
ECOLI 0.606975 0.601889 0.175894 0.207125 0.158884 0.000000 0.014737 0.007327
ENT38 0.601354 0.596308 0.178876 0.216195 0.162592 0.014737 0.000000 0.007323
SALTY 0.611322 0.606229 0.175618 0.206797 0.161981 0.007327 0.007323 0.000000
Для выполнения принципа ультраметричности необходимо, чтобы "из трех расстояний между тремя объектами два всегда равны
d(AGRRK, RHIEC) = 0.096299
между собой и не меньше третьего".
d(AGRRK, VIBCH) = 0.573690
d(RHIEC, VIBCH) = 0.580740
Наиболее близкие расстояния 0.573690 и 0.580740 (отклонение от равенства 0,00705) больше третьего 0.096299.
d(VIBFM, HAEIN) = 0.210635
d(VIBFM, ECOLI) = 0.207125
d(HAEIN, ECOLI) = 0.158884
Наиболее близкие расстояния 0.210635 и 0.207125 (отклонение от равенства 0,00351)) больше третьего 0.158884.
Принцип аддативности выполняется, если у четырех последовательностей: 1, 2, 3, 4, - из трех сумм d(1,2) + d(3,4); d(1,3) + d(2,4); d(1,4) + d(2,3)
две равны между собой и больше третьей.
d(AGRRK, RHIEC) + d(VIBCH, VIBFM) = 0.096299 + 0.100915 = 0.197214
d(AGRRK, VIBCH) + d(RHIEC, VIBFM) = 0.573690 + 0.610869 = 1.184559
d(AGRRK, VIBFM) + d(VIBCH, RHIEC) = 0.602874 + 0.580740 = 1.183614
Отклонение равно 0,000945.
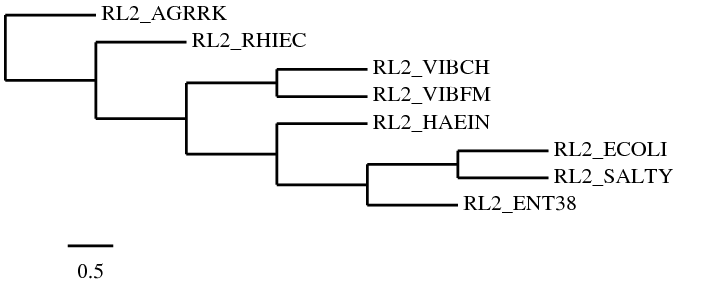
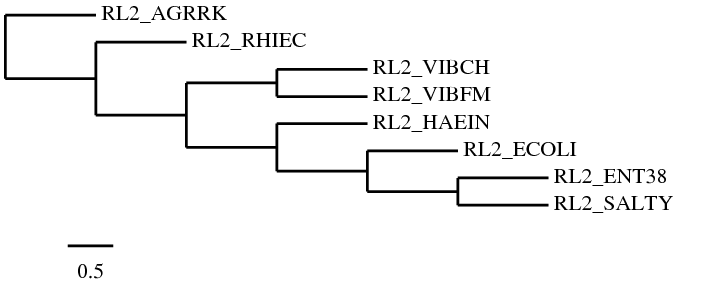
6. Получите две реконструкции дерева программой fneighbor, используя два алгоритма: UPGMA и Neighbor-Joining. Сравните результаты между собой,
с результатом fprotpars и с правильным деревом.
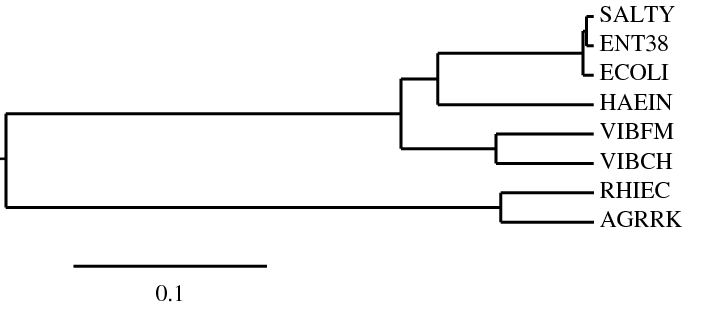
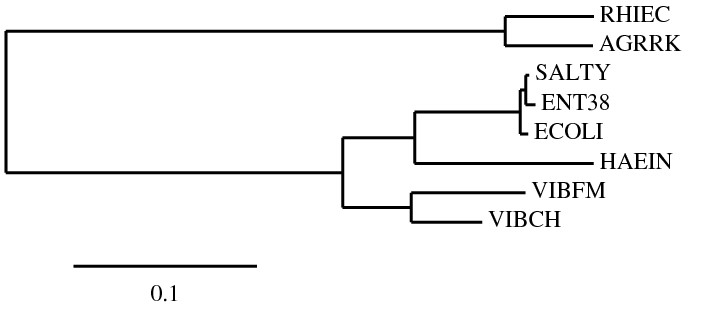
При помощи программы fneighbor были получены две реконструкции дерева, используя два алгоритма: UPGMA и Neighbor-Joining.UPGMA:
((AGRRK:0.04815,RHIEC:0.04815):0.25566,((VIBCH:0.05046,VIBFM:0.05046):0.04911,(HAEIN:0.08058,(ECOLI:0.00552,
(ENT38:0.00366,SALTY:0.00366):0.00185):0.07506):0.01899):0.20425);
Neighbor-Joining:
(RHIEC:0.04840,((VIBCH:0.03865,VIBFM:0.06227):0.03742,(HAEIN:0.09770,(ECOLI:0.00439,(ENT38:0.00536,SALTY:0.00196):0.00298)
:0.05756):0.03924):0.45601,AGRRK:0.04790);Дерево, полученное по алгоритму UPGMA обладает той же морфологией, что и полученное по алгоритму Neighbor-Joining, и совпадает с третьем деревом, построенным программой fprotpairs.
Отличия от настоящего дерева заключены в том, что ECOLI и ENT38 поменялись местами.
В настоящем дереве нет ветви {ENT38, SALTY} против {ECOLI, HAEIN, VIBFM, VIBCH, AGRRK, RHIEC}, а в полученом нет ветви {ECOLI, SALTY} против {ENT38, HAEIN, VIBFM, VIBCH, AGRRK, RHIEC}.
©2008-2010 Михальченко Алексей