Алгоритмы реконструкции деревьев
- Укоренение в среднюю точку
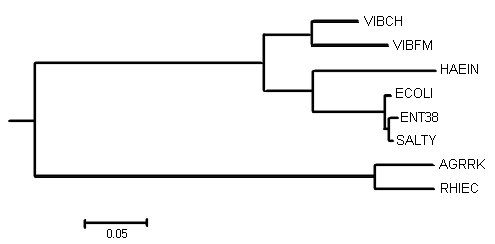
Дерево, полученное с помощью программы fneighbor, используя алгоритм Neighbor-Joining, было укоренено в среднюю точку при помощи программы retree пакета PHYLIP.
Укоренение прошло в ветвь {ECOLI, SALTY, ENT38, HAEIN, VIBFM, VIBCH} против {AGRRK, RHIEC}, и полученное дерево отличается от настоящего только отсутствием ветви {ECOLI, SALTY} против {ENT38, HAEIN, VIBFM, VIBCH, AGRRK, RHIEC}, вместо нее присутствует ветвь {ENT38, SALTY} против {ECOLI, HAEIN, VIBFM, VIBCH, AGRRK, RHIEC}.
Нельзя укоренить дерево, полученное методом максимальной бережливости, так как оно не содержит длин ветвей, и дерево, построенное по алгоритму UPGMA, потому что оно уже укоренено.
- Использование аутгруппы
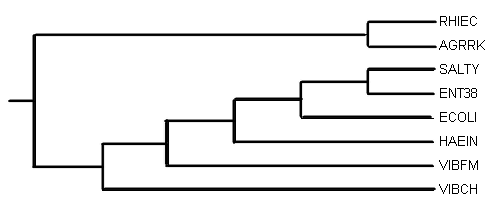
В качестве аутгруппы был выбран рибосомный белок L2 (RL2) бактерии Bacillus subtilis (сенная палочка, BACSU). Программой muscle было построено выравнивание последовательностей белков. Дерево было построено программой fprotpars и обработано программой retree. Из полученного дерева аутгруппа была удалена.
Полученное дерево отличается от настоящего еще одной заменой ветвей по сравнению с результатом укоренения в среднюю точку: в полученном дереве присутствует ветвь {ECOLI, SALTY, ENT38, HAEIN, VIBFM} против {VIBCH, AGRRK, RHIEC}, и отсутствует {VIBFM, VIBCH} против {ECOLI, HAEIN, ENT38, SALTY, AGRRK, RHIEC}.
- Бутстрэп
Для проведения бутстрэп анализа было получено 100 бутстрэп-реплик выравнивания белков протеобактерий программой fseqboot. Потом для каждой реплики было построено дерево программой fprotpars. Из полученных деревьев было создано единое дерево по принципу "расширенного большинства" (extended majority rule tree). Для этого файл с деревьями, выданный программой fprotpars, был подан на вход программе fconsense.
+------AGRRK
+-100.0-| | +------RHIEC
+-92.3-|
| | +------VIBFM
+-99.5-| +-48.8--|
| | +------VIBCH
+-57.8-| | | | +---------------------HAEIN
+------| |
| | +----------------------------ECOLI
| |
| +-----------------------------------SALTY
| +------------------------------------------ENT381. ENT38 2. SALTY 3. ECOLI 4. HAEIN 5. VIBFM 6. VIBCH 7. RHIEC 8. AGRRKЭто дерево совпадает по морфологии с одним из деревьев, полученных с помощью fporpars по множественному выравниванию, но отличается от настоящего, хотя среди полученных с помощью fprotpars было совпадающее по морфологии с настоящим. Таким образом бутстреп анализ не улучшил реконструкцию филогении по сравнению с результатом fprotpars.
Среди ветвей, не получивших поддержки анализа, есть ветвь .**..... с поддержкой 33.80, хотя эта ветвь есть в филогенетическом дереве бактерий ({ECOLI, SALTY} против {ENT38, HAEIN, VIBFM, VIBCH, AGRRK, RHIEC}).
©2008-2010 Михальченко Алексей