Анализ деревьев, содержащих паралоги.
Особенности работы с нуклеотидными последовательностями.
- Построение дерева по нуклеотидным последовательностям
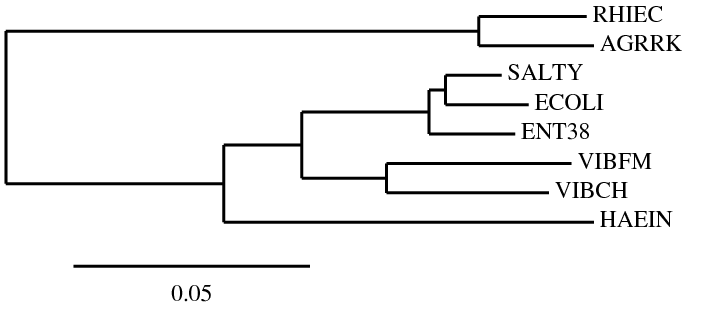
Филогенетическое дерево тех же бактерий было построено, используя последовательности РНК малой субъединицы рибосомы (16S rRNA).
Последовательности 16S рибосомальной РНК каждой из бактерий были получены при помощи сервиса Silva. Эти последовательности были выровнены при помощи muscle (16sRNA_aln.fasta). Матрица расстояний была получена при помощи программы fdnadist (16sRNA_matrix.fdnadist). Программа fneighbor использовала эту матрицу для построения дерева алгоритмом neighbor-joining (16sRNA_tree.fneighbor).
(RHIEC:0.02281,(HAEIN:0.07826,((VIBCH:0.03431,VIBFM:0.03910):0.01795,(ENT38:0.01818,(ECOLI:0.01757,SALTY:0.01188):0.00347):0.02694):0.01649):0.14607,AGRRK:0.02434);Полученное дерево отличается от настоящего одной ветвью: присутствует ветвь {ECOLI, SALTY, ENT38, VIBFM, VIBCH} против {HAEIN, AGRRK, RHIEC}, вместо истиной ветви {ENT38, SALTY, ECOLI, HAEIN} против {VIBFM, VIBCH, AGRRK, RHIEC}. Топология данного дерева не совпадает ни с одной таковой у деревьев, построенных по последовательностям рибосомных белков L2 бактерий.
- Построение и анализ дерева, содержащего паралоги
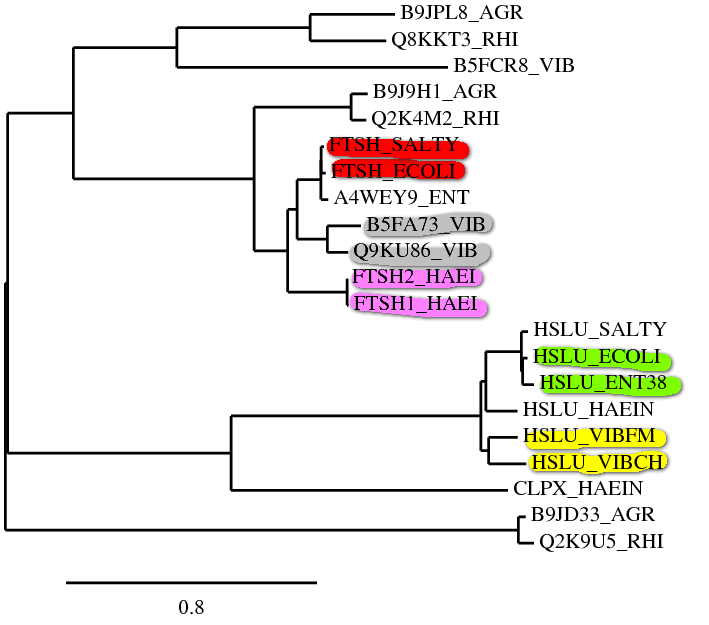
В выбранных бактериях были найдены достоверные гомологи белка FTSH_ECOLI при помощи программы BLASTP (порогом на E-value 0,0001) (FTSH_gomologs.fasta).Последовательности этих гомологов выровняли при помощи muscle (FTSH_gomologs_aln.fasta). Матрица расстояний была получена при помощи программы fdnadist (16sRNA_matrix.fdnadist). Программа fneighbor использовала эту матрицу для построения дерева алгоритмом neighbor-joining (16sRNA_tree.fneighbor).
(B9J9H1_AGR:0.05240,(((B5FCR8_VIB:0.86379,(Q8KKT3_RHI:0.24292,B9JPL8_AGR:0.27075):0.42413):0.33036,((Q2K9U5_RHI:0.04909,B9JD33_AGR:0.02276):1.64364,(CLPX_HAEIN:0.88284,
((HSLU_VIBCH:0.11991,HSLU_VIBFM:0.09321):0.02457,(HSLU_HAEIN:0.10127,((HSLU_ENT38:0.03635,HSLU_ECOLI:0.01530):0.00400,HSLU_SALTY:0.02090):0.11347):0.01581):0.79638):0.71238)
:0.20981):0.57603,((FTSH1_HAEI:0.00478,FTSH2_HAEI:-0.00225):0.18916,((Q9KU86_VIB:0.06662,B5FA73_VIB:0.10720):0.09642,(A4WEY9_ENT:0.02311,(FTSH_ECOLI:0.01448,FTSH_SALTY:0.00894)
:0.00031):0.07592):0.03012):0.10743):0.30979,Q2K4M2_RHI:0.04801);Желтым, зеленым и красным помечены ортологи, розовым и серым - паралоги.
Два гомологичных белка будем называть ортологами, если они а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. Два гомологичных белка из одного организма будем называть паралогами.
©2008-2010 Михальченко Алексей