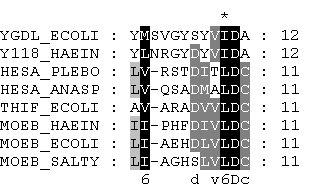Паттерны и профили
1. Создание паттернов аминокислотных последовательностей.
Импортирую в GeneDoc выравнивание белка MOEB_ECOLI и его гомологов, полученное с помощь программы muscle (myproteins_aligned.fasta):
Для исследования я выбрал следующий фрагмент данного выравнивания (.msf, .gif):
Создал три паттерна: первый паттерн в точности является фрагментом последовательности белка MOEB_ECOLI; второй ("сильный") паттерн распознает все белки выравнивания, и только их; третий ("слабый") - на основе второго с более мягкими требованиями к последовательности.
Таблица результатов поиска по паттернам в базе данных SwissProt:
Характеристика паттерна Паттерн В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? Все ли последовательности из Вашего выравнивания найдены? Фрагмент последовательности LIAEHDLVLDC 1 Нет. Найдена только последовательность белка MOEB_ECOLI. Сильный [LYAI]-[VIML]-X(0,1)-[ARPQV]-[GSRHE]-[YAHTF]-[DS]-[LVMIY]-[VAT]-[LI]-D-[CA] 16 Все Слабый [LYAI]-[VIML]-X(3,4)-[DS]-[LVMIY]-[VAT]-[LI]-D-[CA] 149 Все Первый паттерн.
Первый паттерн в точности является фрагментом последовательности белка MOEB_ECOLI, состоит из 11 а.о. В PROSITE при поиске в Swiss-Prot была найдена только последовательность исследуемого белка MOEB_ECOLI.
Второй паттерн.
"Сильный" паттерн, распознающий все белки выборки (и только их). Основная идея при составлении такого паттерна заключается в том, чтобы включить все позиции фрагмента выравнивания, а в каждой позиции разрешить все буквы, встретившиеся в какой-либо из последовательностей.
Как и предполагалось, все восемь белков выборки он распознал, но он также распознал другие восемь белков, не вошедших в выборку:HESA1_ANAVT, HESA2_ANAVT - эти два белка являются гомологами MOEB_ECOLI (E-value 1e-28 и 2e-29 соответственно при поиске, проведенном на прошлом занятии) и даже являются белками бактерий (одно из условий при составлении выборки). Просто они не были включены в выборку, хотя вполне могли бы в нее войти.
LAC1_MELAO - не является белком бактерий (Melanocarpus albomyces - гриб), поэтому не мог быть включен в выборку. О гомологии с MOEB_ECOLI узнать не удалось, по крайней мере при поиске в BLAST среди гомологов его не было.
LAC2_PODAN - не является белком бактерий (Podospora anserina - гриб), поэтому не мог быть включен в выборку. О гомологии с MOEB_ECOLI узнать не удалось, по крайней мере при поиске в BLAST среди гомологов его не было.MOCS3_DROME, MOCS3_DROSE, MOCS3_DROWI, MOCS3_DROYA - эти белки тоже являются гомологами MOEB_ECOLI (E-value 2e-54, 7e-56, 5e-56, 1e-55 соответственно при поиске, не ограничиваясь таксоном Bacteria), но они не являются белками бактерий (род Drosophila - насекомые), что было условием при составлении выборки.
Третий паттерн.
"Мягкий" паттерн, конечно же, распознал больше всего белков (149 фрагментов последовательностей). Многие найденные белки не принадлежат семейству MOEB исследуемого белка MOEB_ECOLI. Есть существенная вероятность того, что будет найдено много "лишних" белков. По такому "слабому" паттерну нельзя проводить поиск мотива в возможных гомологах исслудуемого белка.
2. Все описанные в PROSITE мотивы в заданном белке MOEB_ECOLI.
Идентификатор документа PROSITE (AC) Название мотива Краткое описание мотива Тип подписи (паттерн, профиль) Паттерн (регулярное выражение) Специфична ли подпись? Сколько мотивов нашлось в белке? PS00006 CK2_PHOSPHO_SITE Cайт фосфорилирования казеинкиназой II паттерн [ST]-x(2)-[DE] неспецифична 5 PS00008 MYRISTYL Сайт N-миристоилирования паттерн G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} неспецифична 7 PS00001 ASN_GLYCOSYLATION Cайт N-гликозилирования паттерн N-{P}-[ST]-{P} неспецифична 1 PS00005 PKC_PHOSPHO_SITE Сайт фосфорилирования протеинкиназой C паттерн [ST]-x-[RK] неспецифична 2
©2008 Михальченко Алексей