Функции, продолжение. Мембранные белки, транспортные белки
Построить выравнивание заданного белка и белка-прототипа с разметкой трансмембранных сегментов.
Заданный белок: MDR2_CRIGR. Белок-прототип: MDR3_MOUSE.
- Сравнить нумерацию остатков белка-прототипа в UniProt и PDB
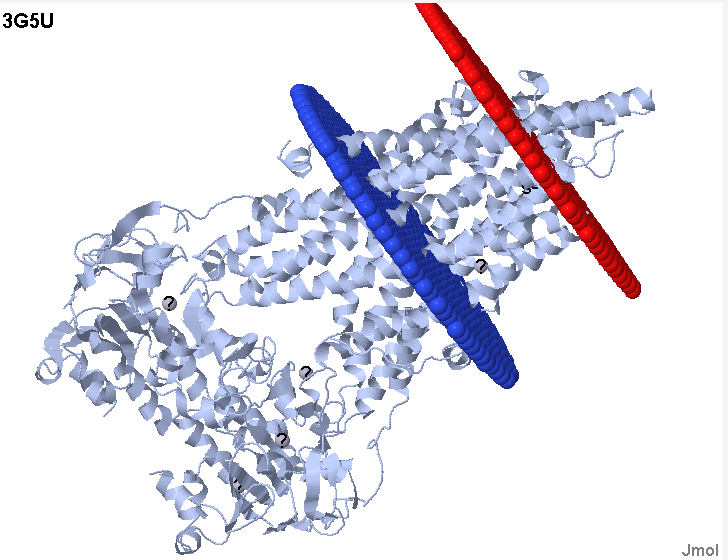
На странице БД PDBsum, предоставляющей краткое схематическое отображение информации о структуре, найдем документ с заданным идентификатором PDB белка прототипа - 3G5U. Подведя курсор к выравниванию последовательности в структуре и щелкнув по картинке, получим окно с выравниванием последовательности из UniProt и последовательности из PDB.
Импортируем выравнивание в Genedoc и сохраните как файл algn1.msf.Нумерация в обеих базах данных совпадает.
- Построение полного глобального выравнивания.
В БД UniProt были получены последовательности заданного белка MDR2_CRIGR и белка-прототипа MDR3_MOUSE.Глобальное выравнивание было построено при помощи программы needle:
needle -asequence P21449.fasta -bsequence P21447.fasta -outfile P21449-P21447.needle
Gap_penalty: 10.0
Extend_penalty: 0.5Создать по данным БД ОРМ разметку трансмембранных сегментов в белке-прототипе
На домашней странице БД ОРМ, по PDB ID 3G5U найдем описание ТМ-сегментов в белке-прототипе.
P-glycoprotein:
Type: 1. Transmembrane
Class: 1.1. Alpha-helical transmembrane
Superfamily: 1.1.09. ABC transporters
Family: 1.1.09.06. Multidrug resistance exporter (MDR)
Species: Mus musculus
Localization: Eukaryotic plasma membrane
A - Tilt: 3° - Segments: 1( 48- 72), 2( 111- 131), 3( 185- 207), 4( 210- 233), 5( 290- 314), 6( 327- 348), 7( 708- 729), 8( 750- 773), 9( 827- 847), 10( 853- 872), 11( 931- 953), 12( 971- 988)
Исходя из этих координат были найдены трансмембранные сегменты.
На основании описания добавим в созданное в предыдущем пункте выравнивание строчку с разметкой трансмембранных сегментов.
Внешняя часть мембраны изображена красным, внутренняя - синим.Полученное выравнивание (mark.msf).
4. Предсказание топологии заданного белка с помощью программы TMHMM
Предскажем топологию заданного белка MDR2_CRIGR с помощью сервера TMHMM.
На вход подадим ему файл MDR2_CRIGR.
Выдача программы:
# sp_P21449_MDR2_CRIGR Length: 1276
# sp_P21449_MDR2_CRIGR Number of predicted TMHs: 11
# sp_P21449_MDR2_CRIGR Exp number of AAs in TMHs: 250.10729
# sp_P21449_MDR2_CRIGR Exp number, first 60 AAs: 11.68149
# sp_P21449_MDR2_CRIGR Total prob of N-in: 0.99408
# sp_P21449_MDR2_CRIGR POSSIBLE N-term signal sequence
sp_P21449_MDR2_CRIGR TMHMM2.0 inside 1 48
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 49 71
sp_P21449_MDR2_CRIGR TMHMM2.0 outside 72 114
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 115 137
sp_P21449_MDR2_CRIGR TMHMM2.0 inside 138 188
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 189 211
sp_P21449_MDR2_CRIGR TMHMM2.0 outside 212 215
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 216 238
sp_P21449_MDR2_CRIGR TMHMM2.0 inside 239 293
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 294 316
sp_P21449_MDR2_CRIGR TMHMM2.0 outside 317 325
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 326 348
sp_P21449_MDR2_CRIGR TMHMM2.0 inside 349 715
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 716 738
sp_P21449_MDR2_CRIGR TMHMM2.0 outside 739 752
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 753 775
sp_P21449_MDR2_CRIGR TMHMM2.0 inside 776 844
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 845 867
sp_P21449_MDR2_CRIGR TMHMM2.0 outside 868 935
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 936 958
sp_P21449_MDR2_CRIGR TMHMM2.0 inside 959 970
sp_P21449_MDR2_CRIGR TMHMM2.0 TMhelix 971 993
sp_P21449_MDR2_CRIGR TMHMM2.0 outside 994 1276
График, отображающий предположение о топологии:
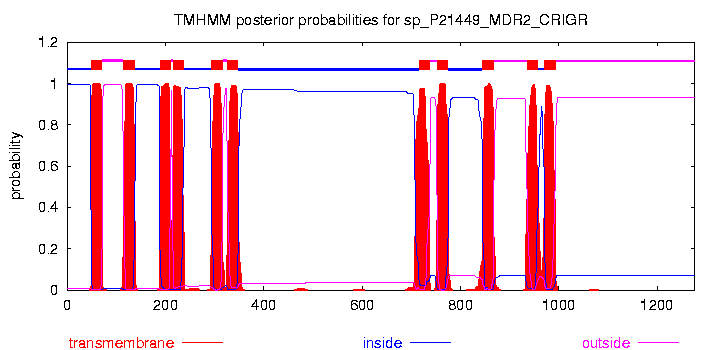
Полученное предсказание добавим к выравниванию файла mark.msf в виде искусственной последовательности с разметкой трансмембранных спиралей под названием "TMHMM".
Полученное выравнивание сохранено в формате msf (mark2.msf).
- Сравните полученное предсказание с данными ОРМ.
Рассмотрим полученное выравнивание. По имеющимся уже данным OPM и TMHMM построим таблицу:
Результаты предсказания топологии мембранного белка MDR2_CRIGR
Число а.к. остатков Всего а.к. остатков 1276 Остатки, предсказанные как локализованные в мембране (всего) 253 Правильно предсказали (true positives, TP) 211 Предсказали не то, что нужно (а.о. предсказаны как мембранные, а по данным ОРМ таковыми не являются, false positives, FP) 42 Правильно не предсказали ( не предсказаны, и по данным ОРМ не находятся в мембране, true negatives, TN) 965 Не предсказали то, что нужно (остатки по данным ОРМ находятся в мембране, false negatives, FN) 57 Чувствительность (sensivity) = TP / (TP+FN) 0.7873 Специфичность (specificity) = TN / (TN+FP) 0.9583 Точность(precision) = TP /(TP+FP) 0.8340 Сверхпредсказание = FP/ (FP+TP) 0.1660 Недопредсказание = FN / (TN+FN) 0.0558
Необходимо отметить, что одна трансмембранная спираль не была предсказана TMHMM. На месте 9 и 10 трансмембранной спирали в OPM в TMHMM предсказана только одна трансмембранная спираль, в результате чего далее неверно определена ориентация.
Сверхпредсказание преобладает над недопредсказанием почти в 3 раза..
©2008-2010 Михальченко Алексей