Практикум №15
Сборка de novo
1) Подготовка чтений программой trimmomatic
Код доступа проекта по секвенированию бактерии Buchnera aphidicola - SRR4240359.
С сайта ENA был скачан соответствующий файл в формате fastq с помощью команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/009/SRR4240359/SRR4240359.fastq.gz
Далее был создан файл в формате fasta со всеми возможными адаптерами, которые нужно вырезать из прочтений, с помощью команды:
cat /mnt/scratch/NGS/adapters/* > adapters.fasta
Также были удалены адаптеры (в adapters.fasta) из чтений в файле SRR4240359.fastq.gz с помощью программы Trimmomatic с использованием команды:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240359.fastq.gz SRR4240359_trimmed.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7
- Изначально: 13557938 чтений
- Осталось: 13502066 (99.59%) чтений
- Удалилось: 55872 (0.41%) чтений
С правого конца (3') чтений были удалены нуклеотиды, не прошедшие порог качества (меньше 20). Оставлены чтения, длина которых не меньше 32 нуклеотидов. Команда:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240359_trimmed.fastq.gz SRR4240359_final.fastq.gz TRAILING:20 MINLEN:32
- Изначально: 13502066 чтений
- Осталось: 12184080 (90.24%) чтений
- Удалилось: 1317986 (9.76%) чтений
2) Подготовка k-меров с помощью программы velveth
Была сформирована директория с k-мерами (длиной 31) из обработанных чтений с помощью команды:
velveth SRR4240359 31 -short -fastq.gz SRR4240359_final.fastq.gz
3) Сборка на основе k-меров с помощью velvetg
Была создана сборка с помощью команды:
velvetg SRR4240359
Длина N50: 70607
Самыми длинными контигами оказались: NODE_1 (длина - 108447, покрытие - 43,4), NODE_11 (длина - 125674, покрытие - 44,55), NODE_14 (длина - 71403, покрытие - 39,4).
Были найдены контиги с аномальными покрытиями: один из них имеет длину 47 и покрытие 139,5 (аномально большое), другой - длину 31, покрытие 2,03 (аномальное малое).
4) Анализ с помощью megablast
Далее с помощью программы megablast было проведено сравнение каждого из трёх самых длинных контигов с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253).
Ниже представлены карты локального сходства. Каждые контиги картировались по 15, 25 и 14 участкам соответственно. Между ними участки, не имеющие гомологов в геноме. Их характеристики представлены ниже в соответствующих таблицах для каждого контига.
Рис.1 Картирование контига NODE_1 на геном.
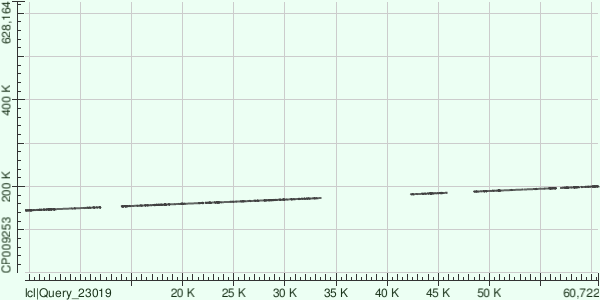

Рис. 2 Картирование контига NODE_11 на геном.
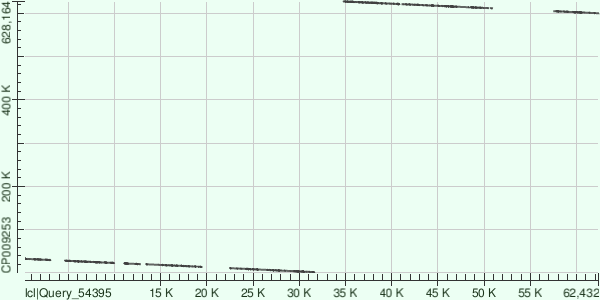

Рис. 3 Картирование контига NODE_14 на геном.
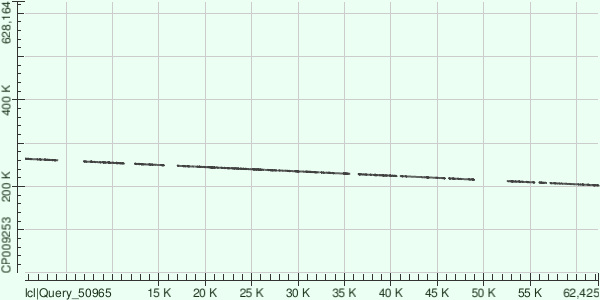

Картирование контигов произошло почти друг за другом и с большим количеством разрывов. Они покрыли почти половину генома бактерии. Контиги NODE_11 и NODE_14 оказались инвертированными по отношению к геному бактерии. Контиг NODE_11 наложился на точку начала последовательности генома бактерии.