Практикум №4
Результаты этого практикума сравнивались с эталонным деревом, полученным в первом практикуме.
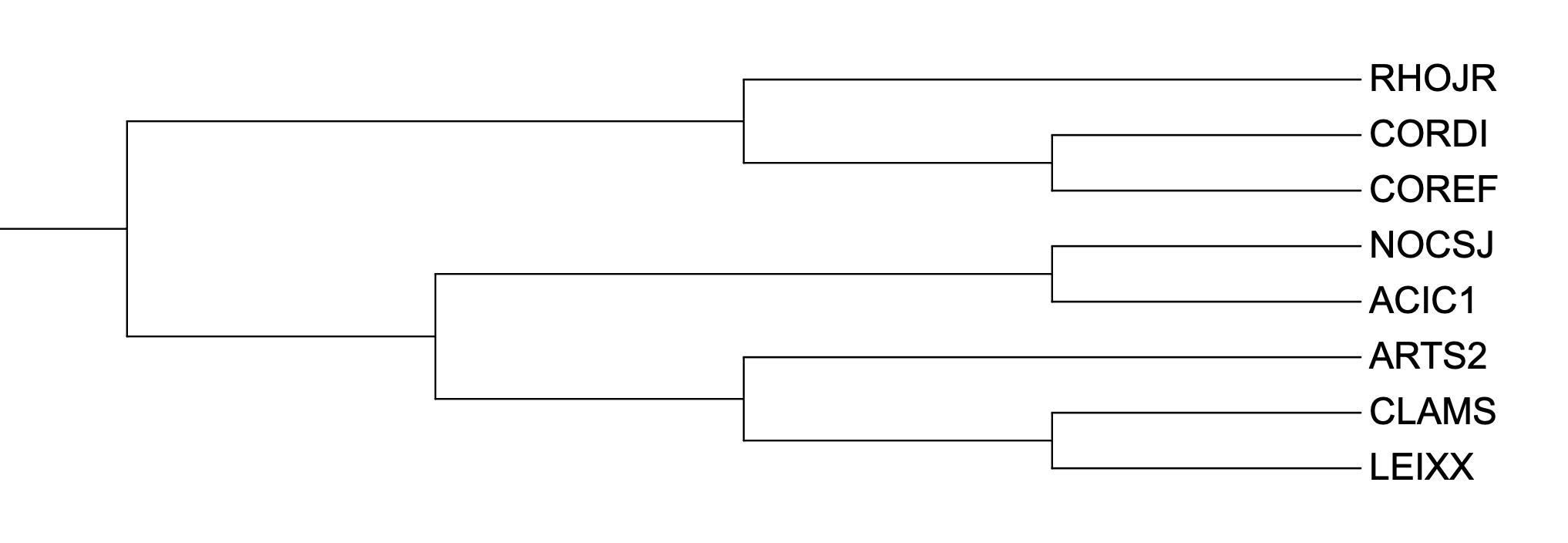
Рис. 1 Эталонное дерево
Составление списка гомологичных белков, включающих паралоги
Из директории /P/y21/term4/Proteomes были скачаны протеомы отобранных бактерий. Затем они были объеденены в единый файл proteomes.fasta с помощью команды:
cat *.fasta > proteomes.fasta
Полученный файл был проиндексирован комнадой:
makeblastdb -in proteomes.fasta -dbtype prot -parse_seqids
Далее локальным запуском blastp были найдены гомологи белка CLPX_ECOLI из полученных выше данных (порог для e-value = 0,001):
blastp -query P0A6H1.fasta -db proteomes.fasta -out final.tab -evalue 0.001 -outfmt 7
Реконструкция и визуализация
Все находки были объединены в единый файл.
Далее было реконструировано дерево найденных гомологов программой FastME c параметрами: 'Gamma distributed rates across sites' — No, 'Starting tree' — BIONJ, 'No refinement', 100 бутстреп реплик.
Дерево в формате Newick
Считая дерево реконструированным верно, можно привести примеры ортологов и паралогов:
Ортологи: CLPX_COREF и CLPX_CORDI, CLPX_LEIXX и CLPX_CLAMS, 00S8C7_RHOJR и 06NFB1_CORDI
Паралоги: CLPX_LEIXX и Q6ACQ0_LEIXX, 06NFB1_CORDI и CLPX_CORDI, AOLRB8_ACIC1 и FTSH_ACIC1
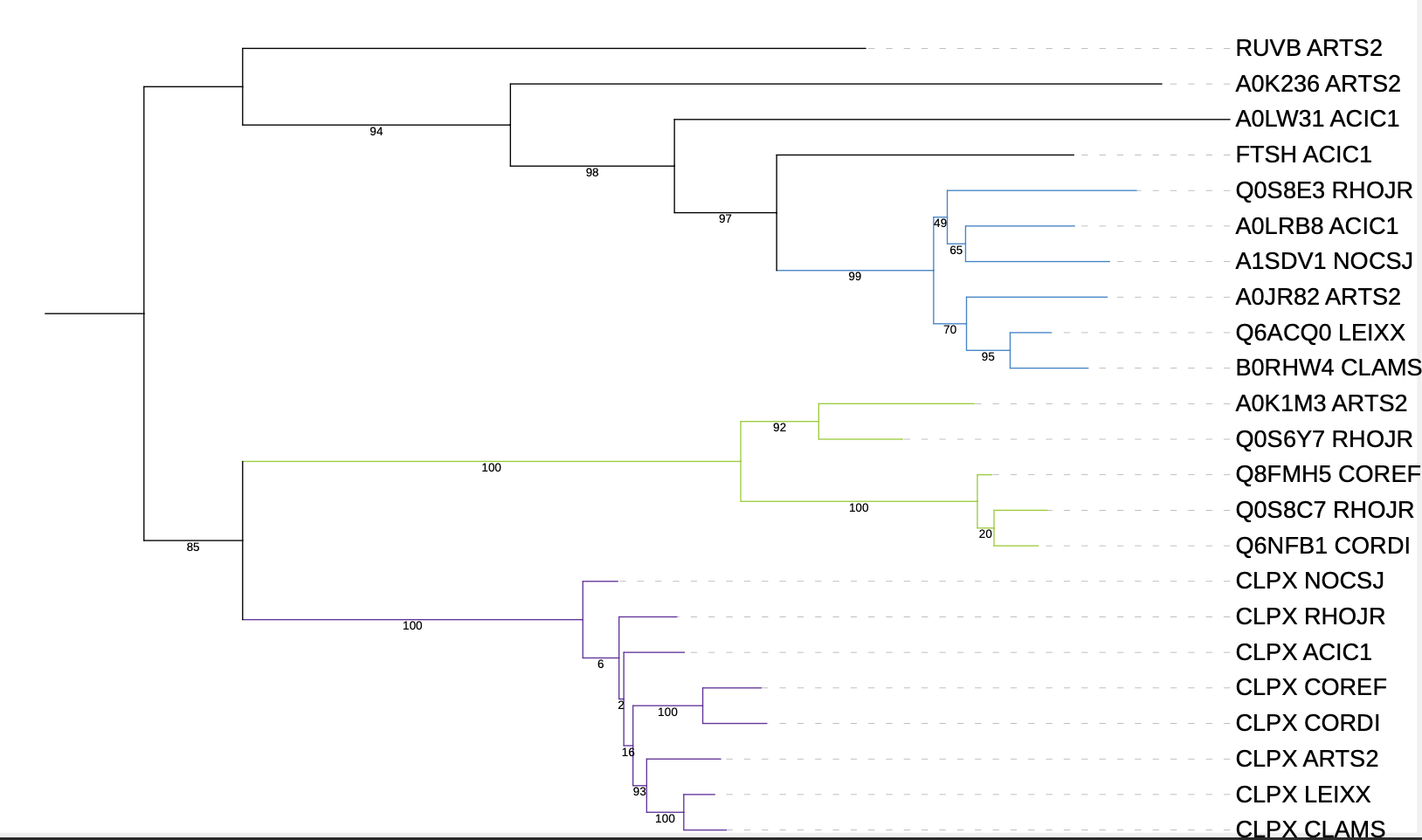
Рис. 2 Дерево гомологов CLPX_ECOLI. Ортологичные группы выделены цветами, группы с малым количеством ортологов (меньшим или равным трём) выделены чёрным.
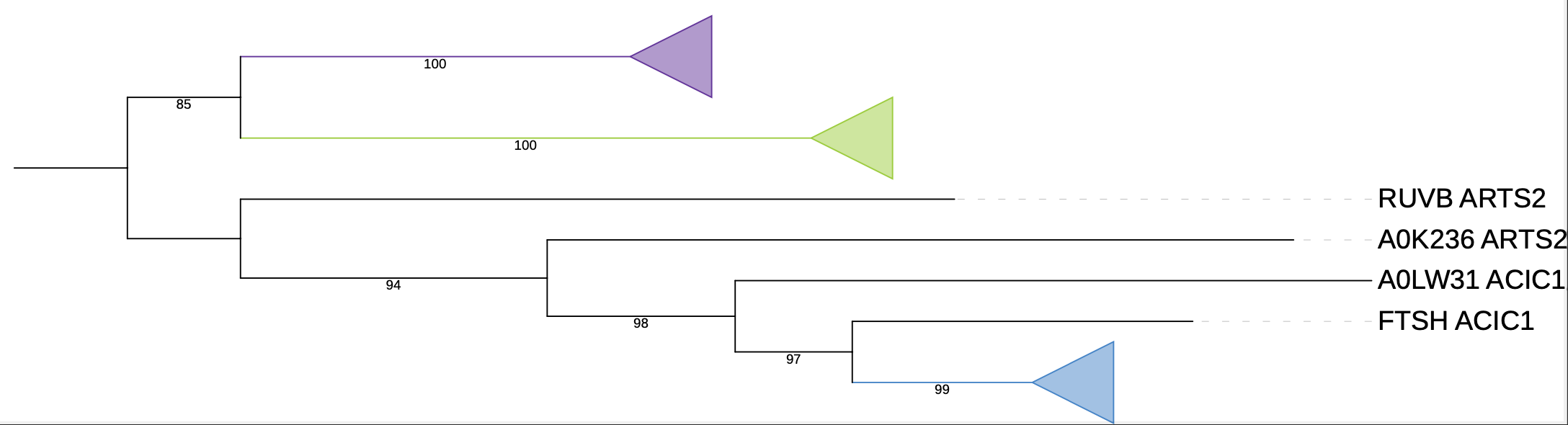
Рис. 3 Дерево из Рис. 2, на котором ортологичные группы (с сохранением соответствующих цветов) 'схлопнуты'. В фиолетовую группу вошли все из семи отобранных видов бактерий, ветви {CLAMS, LEIXX, ARTS2}, {COREF, CORDI} соответсвуют аналогичным ветвям эталонного дерева. Все гены из данной группы являются АТФ-зависимыми протеазами Clp. В зеленую группу вошли бактерии ARTS2, RHOJR, COREF, CORDI - она филогенетически не очень хорошо соответствует референсному дереву. В голубую группу вошли RHOJR, ACIC1, NOCSJ, ARTS2, LEIXX, CLAMS - ветви {CLAMS, LEIXX, ARTS2}, {NOCSJ, ACIC1} соответствуют аналогичным ветвям эталонного дерева.