В данном практикуме работали со штаммом Tuc7 бактерии Buchnera aphidicola (Acyrthosiphon pisum) (AC проекта по секвенированию: SRR4240356). Секвенировали по технологии Illumina.
- Далее скачали архив с чтениями на kodomo при помощи команды wget:
- С помощью программы FASTQC визуализировали качество прочтений (ссылка):
- Из директории /mnt/scratch/NGS/adapters были скопированы адаптеры для одиночных чтений в один файл adapters-SE.fasta:
- Фильтруем чтения при помощи программы trimmomatic (удаляем адаптеры, нуклеотиды с правого конца с качеством ниже 20, оставляем чтения длиной не меньше 32):
- С помощью программы FASTQC визуализировали качество прочтений (после удаления адаптеров, после второй фильтрации):
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/006/SRR4240356/SRR4240356.fastq.gz
fastqc SRR4240356.fastq.gz
cat TruSeq2-SE.fa > adapters-SE.fasta cat TruSeq3-SE.fa >> adapters-SE.fasta
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240356.fastq.gz SRR4240356_noadapters.fq.gz ILLUMINACLIP:adapters-SE.fasta:2:7:7 java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240356_noadapters.fastq.gz SRR4240356_out.fq.gz TRAILING:20 MINLEN:32
fastqc SRR4240356_out.fq.gz
Сравним, что изменилось после тримирования. В результате фильтрации чтений оказалось, что 152380 последовательностей (2.03%) оказались остатками адаптеров.
| Параметры | До тримирования | После удаления адаптеров | После второго тримирования |
|---|---|---|---|
| Размер файла (AC.fastq.gz) (в байтах) | 174262033 байт (174,26 Mb) | 171398665 байт (171,4 Mb) | 162162102 байт (162,22 Mb) |
| Количество чтений | 7511529 | 7359149 | 7059570 |
| Длина чтений | 36 | 1-36 | 32-36 |
- Запускаем программу velveth со следующими параметрами для коротких одноконцевых чтений (подготовка k-меров длины k=31 (параметр "hash_length")):
- Запускаем программу velvetg со следующими параметрами для сборки de novo на основе k-меров:
velveth velveth 31 -short -fastq SRR4240356_out.fq.gz
velvetg velveth
Итогом программы является информация о N50 (65554), а также файлы contigs.fa (контиги) и stats.txt (характеристики контигов).
- С помощью команды sort получили информацию о самых больших контигах (табл.2):
- С помощью команды sort получили информацию о контигах с аномально большим покрытием (табл.3):
- С помощью команды sort получили информацию о контигах с аномально малым покрытием (табл.4):
sort -n -r -k 2 stats.txt | head
sort -n -k 6 -r stats.txt | head -n 3
sort -n -k 6 stats.txt | head -n 3
| ID | Длина | Покрытие | Файл с последовательностью |
|---|---|---|---|
| 8 | 111962 | 38.668870 | contig8 |
| 6 | 107488 | 34.195585 | contig6 |
| 10 | 80939 | 37.546325 | contig10 |
| ID | Длина | Покрытие | Файл с последовательностью |
|---|---|---|---|
| 64 | 1 | 266957.0 | - |
| 129 | 1 | 1134.0 | - |
| 28 | 282 | 458.432624 | contig28 |
Как видно из этой таблицы, встречаются контиги с длиной, равной один (т.е. у них всего 31 нуклеотид), которые имеют большое покрытие. Возможно, это шум.
Последовательности контигов 64, 129, а также тех, которые слабо покрывают геном (см. табл.4), не встречаются в файле contigs.fa.
| ID | Длина | Покрытие | Файл с последовательностью |
|---|---|---|---|
| 251 | 3 | 1.0 | - |
| 253 | 2 | 1.0 | - |
| 274 | 1 | 1.0 | - |
Что касается контигов с малым покрытием, встречается один с покрытием, равным один, и длиной, равной единице. Также контиги с длинами, равными, соответственно, 2 и 3, слабо покрывают геном (покрытие равно единице).
Megablast
Запускаем на сайте NCBI megablast, введя две последовательности: контиг и геном штамма BAg бактерии Buchnera aphidicola (Aphis glycines) (AC: CP009253). Проанализируем каждое выравнивание. В табл. 5 представлены ссылки на отчёты работы megablast, а в 7-8 - характеристики выравниваний для каждого контига: число однонуклеотидных замен, количество гэпов, участок генома Buchnera aphidicola и участок конкретного контига, которые вошли в выравнивание.
| ID контига | E-value | Per.Identity | Отчёт |
|---|---|---|---|
| 8 | 0.0 | 81.46% | contig8 |
| 6 | 0.0 | 78.76% | contig6 |
| 10 | 0.0 | 74.88% | contig10 |
Contig8
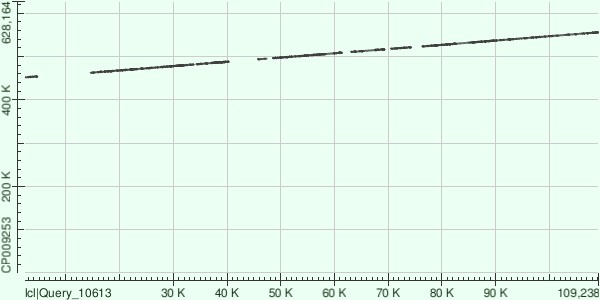
| Координаты участка генома | Координаты участка контига | Число гэпов | Число однонуклеотидных различий |
|---|---|---|---|
| 451729-454069 | 2390-4733 | 55 | 488 |
| 462496-467421 | 14624-19565 | 162 | 992 |
| 467412-474667 | 19595-26906 | 208 | 1489 |
| 474844-480660 | 27009-32884 | 255 | 1288 |
| 480874-481545 | 33090-33769 | 20 | 102 |
| 481997-488106 | 34243-40300 | 308 | 1309 |
| 493487-494864 | 45773-47149 | 13 | 262 |
| 495033-495148 | 47283-47401 | 5 | 7 |
| 496111-500325 | 48567-52845 | 154 | 914 |
| 500370-508806 | 52961-61406 | 351 | 1750 |
| 510438-516539 | 63097-69275 | 187 | 1150 |
| 517766-521500 | 70536-74265 | 99 | 763 |
| 523105-528679 | 76268-81855 | 207 | 1109 |
| 528794-550219 | 81925-103395 | 545 | 3211 |
| 550361-555905 | 103601-109238 | 133 | 950 |
По DotPlot`у видно, что между участками контига, выравнивавшимися на банковский геном, происходило несколько делеций.
Contig6
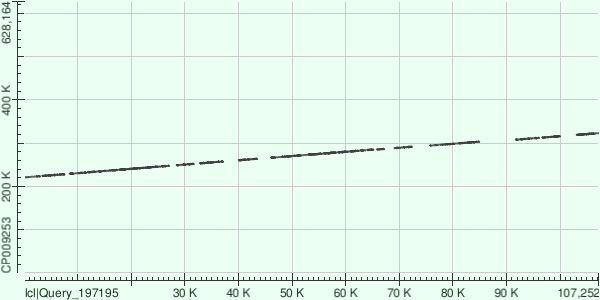
| Координаты участка генома | Координаты участка контига | Число гэпов | Число однонуклеотидных различий |
|---|---|---|---|
| 220869-223720 | 146-2996 | 19 | 483 |
| 224057-228137 | 3385-7496 | 163 | 799 |
| 228944-232057 | 8396-11516 | 97 | 573 |
| 232358-236859 | 11665-16194 | 130 | 985 |
| 236918-247596 | 16292-26990 | 390 | 2272 |
| 248967-252161 | 28467-31669 | 94 | 625 |
| 253244-257546 | 32780-37082 | 192 | 978 |
| 260224-263784 | 39869-43440 | 111 | 717 |
| 266073-275551 | 45989-55468 | 363 | 1689 |
| 275566-283706 | 55527-63756 | 421 | 1579 |
| 283963-285070 | 64004-65113 | 46 | 422 |
| 285200-286535 | 65810-67144 | 27 | 295 |
| 288181-291560 | 68934-72299 | 98 | 671 |
| 294227-295755 | 75721-77247 | 14 | 279 |
| 295935-303252 | 77556-84909 | 186 | 1547 |
| 307878-312179 | 91741-96052 | 120 | 889 |
| 312679-315982 | 96698-100006 | 89 | 681 |
| 318826-323043 | 103039-107252 | 174 | 950 |
По DotPlot`у видно, что между участками контига, выравнивавшимися на банковский геном, происходило несколько делеций, аналогично с контигом 8.
Contig10
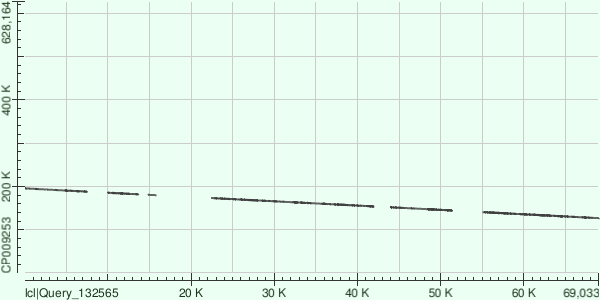
| Координаты участка генома | Координаты участка контига | Число гэпов | Число однонуклеотидных различий |
|---|---|---|---|
| 126623-127815 | 67840-69033 | 11 | 184 |
| 127825-140555 | 55035-67775 | 544 | 2723 |
| 144368-151796 | 43997-51396 | 243 | 1430 |
| 153752-161738 | 33933-42017 | 266 | 1557 |
| 161898-166752 | 28867-33727 | 108 | 894 |
| 166750-173180 | 22393-28836 | 159 | 1393 |
| 179654-180620 | 14869-15834 | 1 | 144 |
| 181712-185328 | 10021-13675 | 112 | 774 |
| 187938-192665 | 2708-7482 | 99 | 859 |
| 192777-193984 | 1427-2632 | 4 | 222 |
| 194042-195400 | 37-1400 | 13 | 1121 |
По DotPlot`у видно, что между участками контига, выравнивавшимися на банковский геном, происходило несколько делеций. Кроме того, прямая проходит в обратном направлении, что свидетельствует о том, что контиг записан в обратном порядке. Это также можно проследить в таблице 6.