Задание 1.
Исходные ab1-файлы: прямая, обратная последовательности. В основном, работа велась в бесплатной программе Unipro UGENE и Chromas.
Хроматограмма, в целом, очень хорошая, проблемных зон и шумов очень мало, однако в начале и в конце есть нечитаемые участки (табл.1). Было проведено сравнение предсказания таких участков в UGENE (для проведения своей оценки) и в Chromas (автоматически выделяет проблемные концы). В целом, я согласна со второй программой, и моя оценка концов совпала за исключением конца обратной последовательности: программа не нашла никаких проблем. Уровень шума в forward-файле средний, но он выше, чем в reverse-файле, и достигает почти что середины пиков.
| Последовательность\Участки | Начало | Конец |
|---|---|---|
| Прямая | 1-27 | 375-381 |
| Обратная | 1-26 | 375-379 |
Сначала экспортировала исходные файлы в fasta-формат, причём обратную последовательность преобразовала в комплиментарную и перевернула. Затем в Jalview выровняла прямые последовательности, получила автоматически консенсус (без редактирования), загрузила выравнивание в UGENE. С помощью Chromas визуализировала хроматограмму последовательности, комплиментарную обратной, для удобства поиска проблемных участков при анализе выравнивания (потому что UGENE не захотел работать вообще, а через другие программы мало, что выходило; пришлось самостоятельно вбивать изменения). Далее редактировала сам консенсус.
Рассмотрим проблемные участки:
1. Пики гуанина на 134 и 135 нуклеотидах, которые находятся примерно на середине пиков цитозина, можно посчитать шумом. Последовательность отредактирована в соответствии с обратной последовательностью.
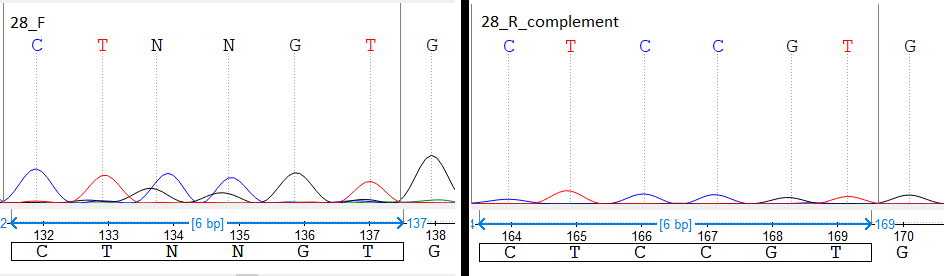
2. Пик гуанина (226 нуклеотид) перекрывает пик аденина, что говорит о возможности возникновении на этом месте полиморфизма. Отредактирован в соответствии с обратной последовательностью.
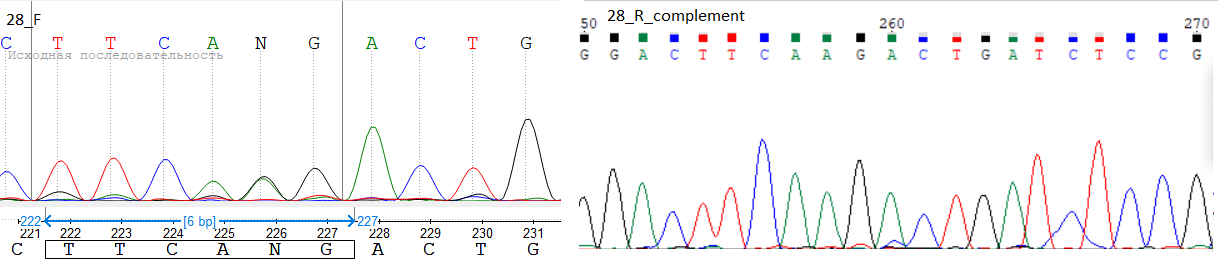
3. На картинке представлена вставка ещё одного цитозина, а затем перекрывание пиков тимина и гуанина. Возникает сигнал аденина, несколько слабее (но секвенатор распознал сигнал; причём расстояние между пиками этого аденина и следующего гуанина на прямой последовательности меньше, чем на обратной). На обратной последовательности подобного не наблюдается. Консенсус отредактирован таким образом: неизвестные нуклеотиды (N) были заменены на цитозин, вставился ещё один цитозин и неизвестный нуклеотид.
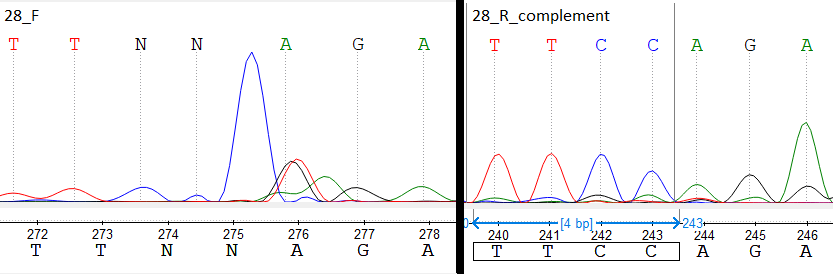
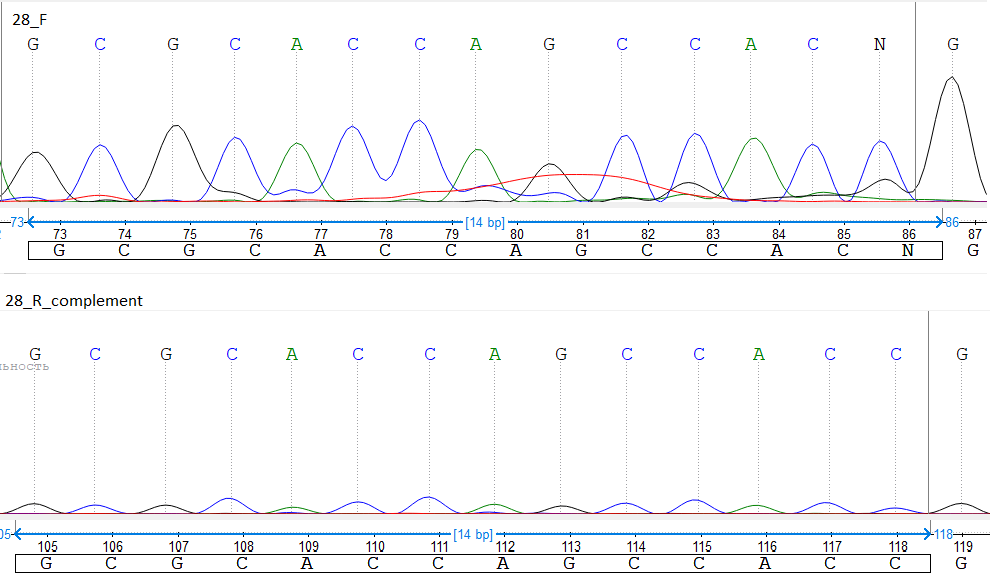
4. Замечены вторичные пики цитозина. Странная ситуация на 80-83 позициях, где наблюдается увеличение сигнала аденина (возможный шум), который выше среднего уровня шума.
Итоговые файлы (с исправлениями): итоговое выравнивание с отредактированным консенсусом, консенсусная последовательность после изменений.
Задание 2.
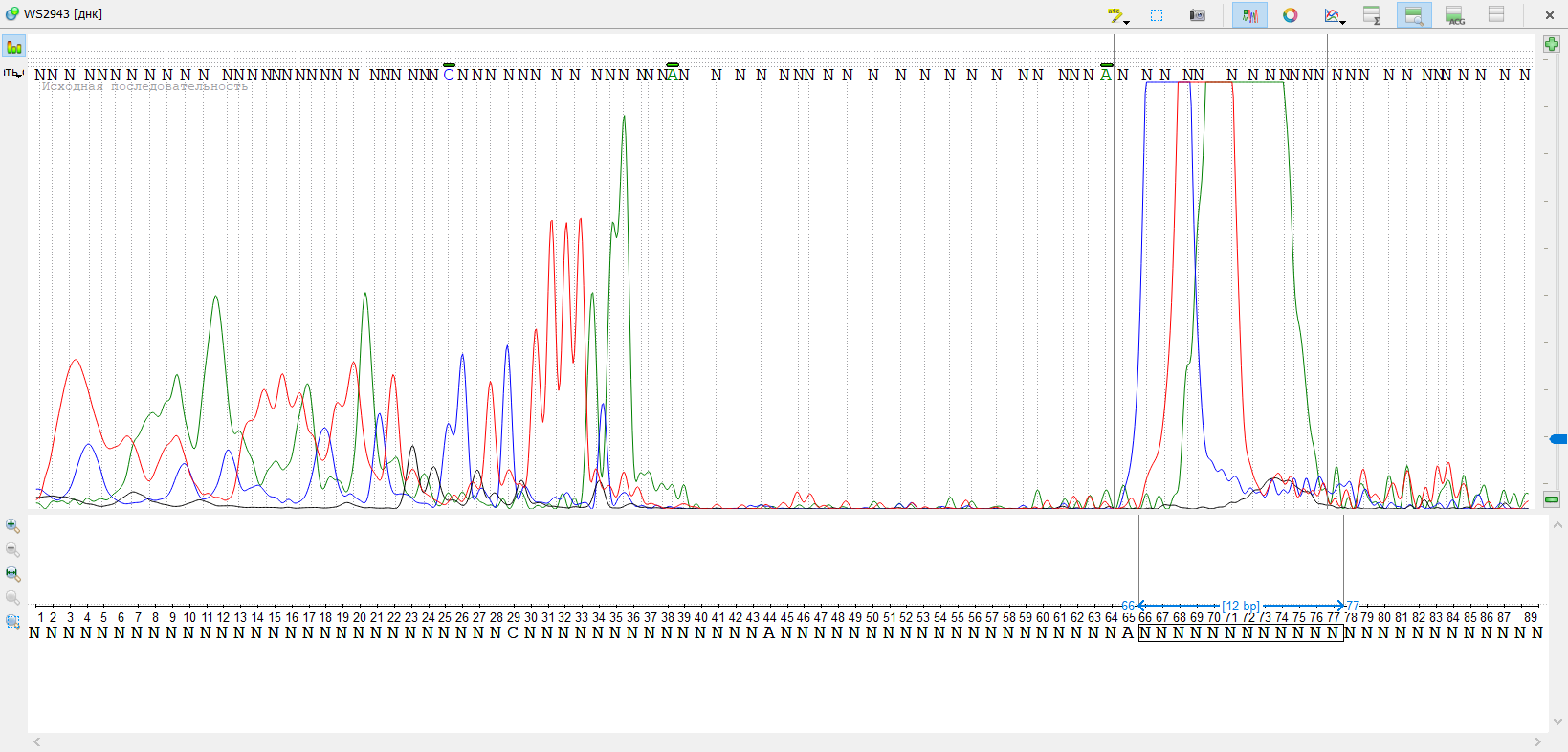
Из папки bad диска Р был взят файл WS2943_SP6R.ab1.
Рассмотрим участок 66-77 п.н. Можно сразу же заметить пятна краски по трём нуклеотидам, что, возможно, связано с ошибками во время электрофореза. Начиная с 73 позиции, наблюдаем появление вторых пиков. Можно предположить, что попала на хроматограмму вторая ДНК.