В данном практикуме работали с выданным α-спиральным белком (см. ниже) и β-листовым белком (см. ниже). Последовательность и трёхмерная структура первого были скачаны.
Знакомство с базой данных OPM
В базе данных OPM, разделе "Classes" был выбран трансмембранный белок, содержащий β-бочонок (Beta-barrel transmembrane). Этот белок TolC - белок внешней мембраны Escherichia coli, участвует в экспорте гемолизина, антибиотиков, энтеробактина из клетки, играет роль в выработке устойчивости к антибиотикам. Информация о нём представлена в табл.1.
| Тип | Трансмембранный |
|---|---|
| Класс | Трансмембранный β-бочонок |
| Суперсемейство | Белок эффлюкс-системы наружней мембраны |
| Локализация | Наружняя мембрана грам-отрицательной бактерии Escherichia coli |
| Толщина гидрофобной части белка в мембране* | 24.6 Å |
| Координаты трансмембранных участков | 1(40-50),2(65-77),3(246-256),4(282-290) (в трёх субъединицах А, В, С) |
| Количество β-тяжей белка | 12 |
| Среднее количество остатков в одном β-тяже белка | 11 |
| PDB ID | 1ek9 |
| Uniprot ID | TOLC_ECOLI |
*Толщину гидрофобной части белка в мембране можно также измерить в JMol (после скачивания pdb-файла из ОРМ) как расстояние между двумя слоями липидов мембраны (указана на рис.1). Она совпадает с данными из БД (24.6 Å).

DeepTMHMM
Запустим сервис DeepTMHMM для наших α-спирального и β-листового белков, чтобы предсказать их трансмембранные элементы. Результаты представлены в текстовом и графическом видах (рис.2, рис.3).
Пояснения к графической выдаче: цветами показано расположение участков в мембране: красный - в мембране (для второго белка - бета-слой (мембранный)), розовый - внутри клетки, голубой - вне клетки, оранжевый - сигнальный пептид для локализации в периплазме, зелёный - в периплазме (между плазматической и наружней мембранами) (на первом графике - подписи на вертикальной оси), - по горизонтальной оси - координаты аминокислотных остатков; на втором графике по вертикали - вероятность того, что какой-то остаток принадлежит к определённой топологии.
- α-спиральный белок: YWIC_BACSU
Текстовая выдача: файл.
Графическая выдача: рис.2.
Итог: предсказано девять трансмембранных участков; N-конец находится внутри клетки, а С-конец - вне её.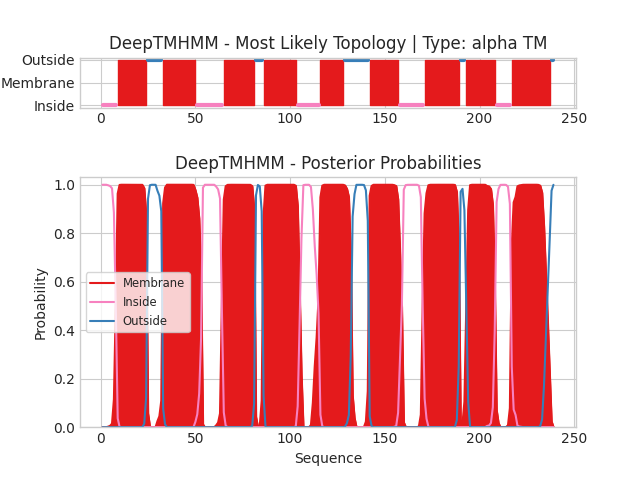
Рис.2. Предсказание для YWIC_BACSU. - β-листовой белок: TOLC_ECOLI
Текстовая выдача: файл.
Графическая выдача: рис.3.
Итог: предсказано четыре трансмембранных участка; N-конец - сигнальный пептид, а С-конец погружён в периплазматическое пространство.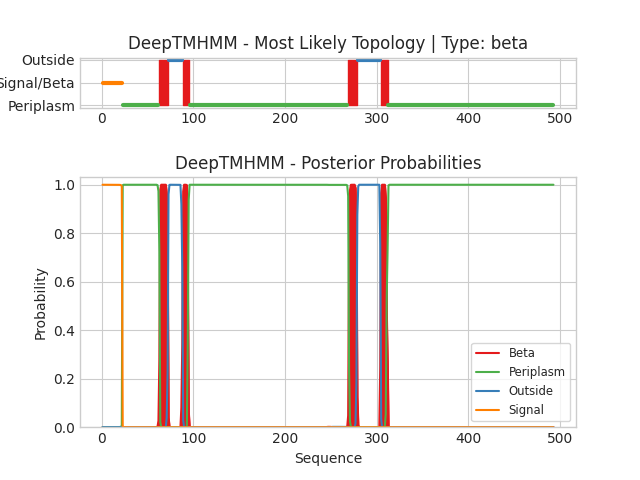
Рис.3. Предсказание для TOLC_ECOLI.
PPM
Используем сервер PPM версии 3.0, чтобы предсказать положение выданного α-спирального белка в мембране (табл.2).
- Параметры:
- Число мембран - 1.
- Тип мембраны - наружняя мембрана грам-отрицательной бактерии (по данным ОРМ).
- Allow curvature - нет (чтобы не усложнять себе
жизньанализ выдачи). - Топология (N-конец) - внутри (по данным DeepTMHMM).
- Файл с координатами - файл с предсказанной структурой из AlphaFold.
| Название | Неохарактеризованный белок YwiC |
|---|---|
| Uniprot Swissprot ID | YWIC_BACSU |
| Предсказанная структура РРМ | pdb-файл |
| Локализация | В мембране грамположительной бактерии Bacillus subtilis |
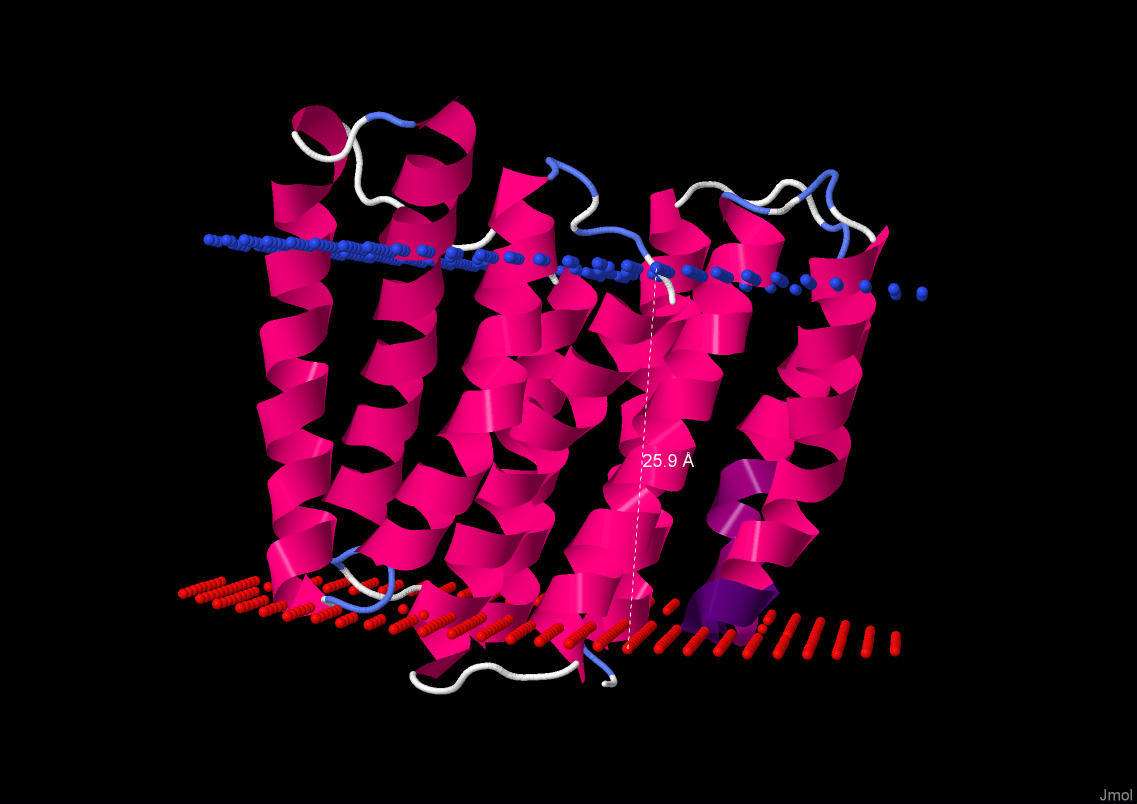
| Толщина гидрофобной части белка в мембране* | 25.9 ± 1.2 Å |
| Координаты трансмембранных участков | 1(9-24), 2(30-46), 3(65-85), 4(86-103), 5(111-132), 6(141-160), 7(171-189), 8(194-208), 9(219-237) |
| Среднее количество остатков в одном β-тяже белка | 16 |
*Толщину гидрофобной части белка в мембране можно также измерить в JMol (после скачивания pdb-файла из ОРМ) как расстояние между двумя слоями липидов мембраны (указана на рис.4). Она равна 25.9 Å.
Сравнение алгоритмов предсказания трансмембранных спиралей
Сравним результаты предсказания трансмембранных участков DeepTMHMM с РРМ для выданного белка:
| DeepTMHMM | РРМ |
|---|---|
|
|
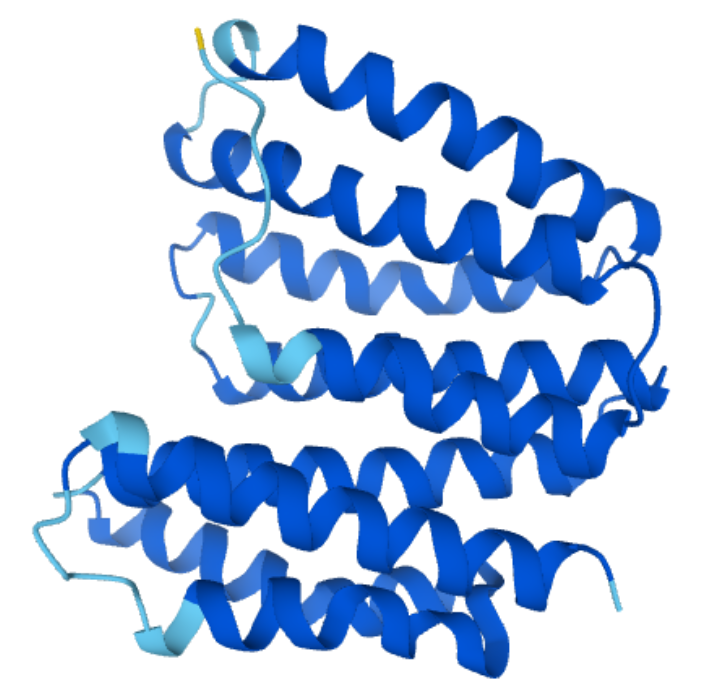
Видим, что предсказания обеих программ мало друг от друга отличаются (разница в 1-9 аминокислотных остатка, С-концы примерно одинаковые), но спирали найдены примерно в одинаковых участках белка. На сайте AlphaFold представлено предсказание участков с оценкой качества предсказания в разных участках полученной структуры (рис.5). Синим цветом (большинство аминокислотных остатков) предсталены участки с весом больше 90 (pLDDT > 90), голубым (32 аминокислотных остатка) - 90 > pLDDT > 70, жёлтым - 70 > pLDDT > 50 (здесь отмечен один участок - первая аминокислота метионин с весом 61.46), красным - ниже 50 (таких нет).
Если взглянуть на информацию, представленную на сайте AlphaFold, то можно заметить, что и там несколько различаются координаты α-спиралей, но положение их примерно одинаковое, поэтому можно сказать, что предсказание, полученное предыдущими программами, хорошее, но погрешности присутствуют. Достоверность модели мало повлияла на результаты предсказания РРМ, поскольку значения незначительно, но отличаются.
База данных TCDB
Используем БД TCDB для поиска наших двух белков. В итоге, белок YWIC_BACSU не был найден (искали по АС Uniprot (P46909), который вбивали в поле для поиска), а TOLC_ECOLI - да (ссылка).
Код TC TOLC_ECOLI: 1.B.17.1.1. Обозначения: 1 - каналы/поры, В - β-бочонок (порин), 17 - семейство факторов внешней мембраны, 1.1 - код данного белка.
На страничке представлена информация о белке TOLC_ECOLI: последовательность, название белка (здесь их несколько: MTCB, MUKA, REFI, B3035), локализация (наружняя мембрана клетки), топология (белок, который несколько раз пронизывает мембрану), длина (493), молекулярная масса (53741), организм, идентификаторы в других БД, Gene Onthology, литературные источники, PDB-идентификаторы, предсказание количества трансмембранных участков (поле "Analyze"), поиск с помощью Blast и другим БД, а также информация о функции в бактериях, строении белка со ссылками на источники.