Описание одного сигнала, закодированного в геноме
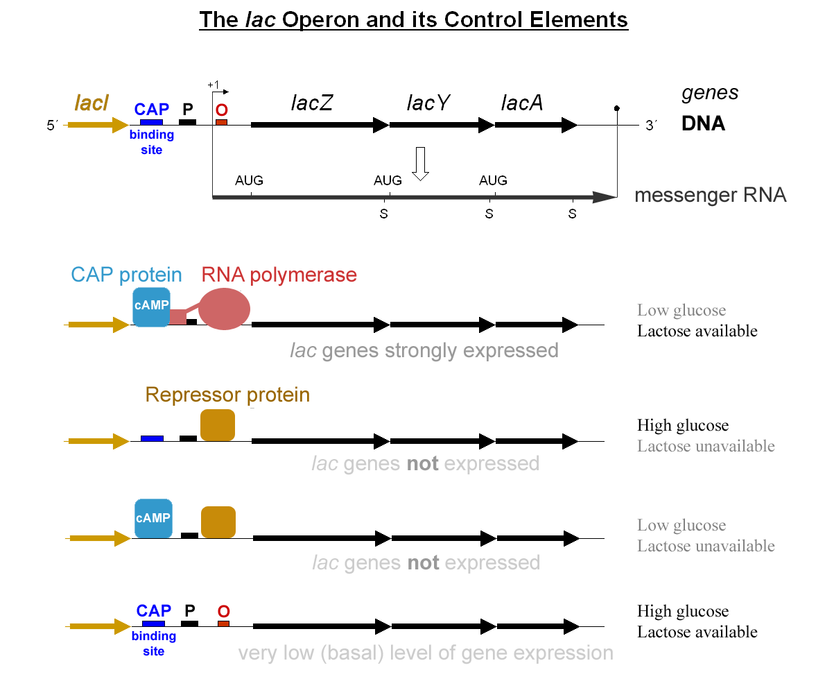
В данном задании был выбран сигнал, находящийся в лактозном опероне бактерий (рис.1). Белок CAP, или CRP (белок-активатор катаболизма сахаров), узнаёт этот сигнал и, связваясь с cAMP (цАМФ), активирует катаболизм лактозы, если отсутствует глюкоза в клетках, необходимая для нормального существования организмов, или её очень мало.
- В случае, когда есть глюкоза в клетках (и может присутствовать лактоза), то лактозный оперон не транскрибируется, т.к. репрессор lacI садится на промотор и не даёт РНК-полимеразе прочитать последовательность, т.к. лактоза не нужна, когда есть глюкоза.
- Если глюкозы становится слишком мало или её нет вовсе, то фермент аденилатциклаза осуществляет превращение из АТФ в цАМФ, который служит сигналом "голода" из-за недостатка сахара. цАМФ связывается с CAP-белком, который затем узнаёт сайт в геноме, перед промотором. Данный комплекс имеет высокое сродство с РНК-полимеразой, и поэтому с его помощью активируется транскрипция лактозного оперона.
Мотив сигнала[1]: 5`-TAATGTGAGTTAGCTCACTCAT-3`
Эту последовательность узнаёт CAP-белок, связанный с цАМФ, который сигнализирует о голоде и что необходимо расщепить лактозу для получения глюкозы, чтобы восстановить нормальное функционирование клеток (для этого необходимо протранскрибировать гены лактозного оперона). Сигнал необходим для активации транскрипции лактозного оперона. Таким образом, адресатом является сам белок, который реагирует именно в случае низкого содержания или полного отсутствия глюкозы и связывается с сайтом. По сравнению со слабым сигналом промотора оперона, этот сигнал эффективнее.
Интересно заметить, что паттерн в CBS (CAP-binding site) лактозного оперона отличается на семь букв от других сайтов, с которым связывается белок: 5′-AATGTGATCTAGATCACATTT-3′.
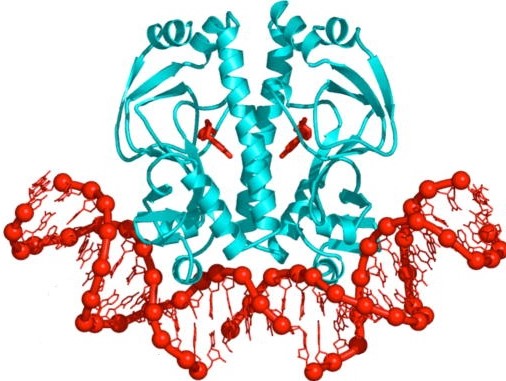
Структура CAP-белка показана на рис.2[2].
Построение позиционной матрицы (PWM) для последовательности Козак человека
Из отфильтрованной таблицы с генами человека в практикуме 6 были выбраны 32 гена, из которых вырезали 13 нуклеотидов: ATG + 3 нуклеотида до него + 7 нуклеотидов после. Затем разделили на две части: в первой 13 генов (40%), а во второй - 19 (60%). Составили выравнивание без гэпов.
PMW-матрица: ссылка (листы "human_genes", "human_PMW_40%", "human_PMW_60%"). Светло-голубые столбцы - ATG-кодон.
Далее проделываем то же самое, но для коронавируса SARS-CoV-2. Из gb-файла вытаскиваем координаты полипротеина (266-21555) и поздних генов (см.таблицу, лист "virus_PMV_positive", "virus_PMV_negative"; с учётом того, что необходимы фрагменты по 13 нуклеотидов, пересчитывали начало и конец отдельно). С помощью пакета EntrezDirect скачиваем последовательности с координататми:
efetch -db "nuccore" -id "NC_045512.2" -format "fasta" -seq_start {start} -seq_stop {stop} >> virus.fasta
efetch -db "nuccore" -id "NC_045512.2" -format "fasta" -seq_start {start} -seq_stop {stop} >> virus_atg.fasta
Видим из матрицы, что распределения весов для положительного и отрицательного контроля отличаются: вес у отрицательного контроля ниже, чем у положительного (как и ожидалось, т.к. в отрицательном контроле присутствуют ATG-кодоны, которые находятся не перед стартом транскрипции, а просто разбросаны по геному).
Исправления
Выражаю большую благодарность Екатерине Кузнеченковой за написанный ею понятный скрипт для вычисления PWM для тестовой выборки (19 генов человека), взятой в качестве положительного контроля, и отрицательного контроля (сайты ATG, не являющиеся началом транскрипции в геноме SARS-CoV-2). Результаты представлены в той же google-таблице (лист "PMW_test_correct"). Видно, что веса у положительного контроля больше, чем у отрицательного контроля. Следовательно, последовательность, находящаяся до ATG-кодона, довольна специфична.
Информационное содержание сигнала старта трансляции - последовательности Козак и построение Logo
Для вычисления информационного содержания необходимо:
- Посчитать, как и при вычислении PMW, частоту встречаемости каждого нуклеотида во фрагменте (в штуках и в пересчёте на количество фрагментов), но без расчёта псевдокаунтов.
- Посчитать по следующей формуле:
IC(b,j) = f(b,j)*log2[f(b,j)/p(b)] = f(b,j)*w(b,j), где:
f(b,j)=N(b,j)/N - частота встречаемости нуклеотида b на позиции j; p(b) — базовая частота основания.
Если f(b,j) = 0, то IC(b,j) = 0.
Расчёты также представлены в таблице (лист "human_IC").
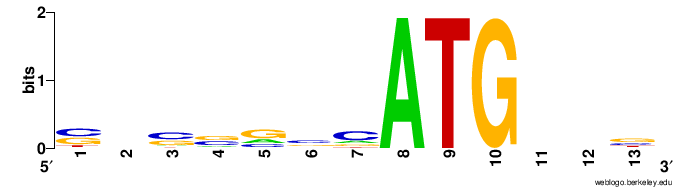
С помощью сервиса WebLOGO3 был получен LOGO для последовательности Козак человека (рис.3).
Список литературы
- Ebright, R. H., Ebright Y. W., Gunasekera, A., Consensus DNA site for the Escherichia coli catabolite gene activator protein (CAP): CAP exhibits a 450-fold higher affinity for the consensus DNA site than for the E.coli lac DNA site , Nucleic Acids Research, Volume 17, Issue 24, 25 December 1989, Pages 10295–10305
- Lawson, C. L., Swigon, D., Murakami, K. S., Darst, S. A., Berman, H. M., & Ebright, R. H. (2004). Catabolite activator protein: DNA binding and transcription activation. Current opinion in structural biology, 14(1), 10–20.