Мотив сайта разрывной транскрипции sgRNA в геноме одного из коронавирусов (Coronaviridae)
В данном задании был выбран Night heron coronavirus HKU19. Далее скачали его геном и составили координаты полипротеина и каждого позднего гена. С помощью python вырезали из генома интересующие нас upstream-области перед каждым геном белка и записали последовательности в файл.
Дальше находим мотив сайта CS с помощью MEME, параметры выставлены так, как написано в самом практикуме.
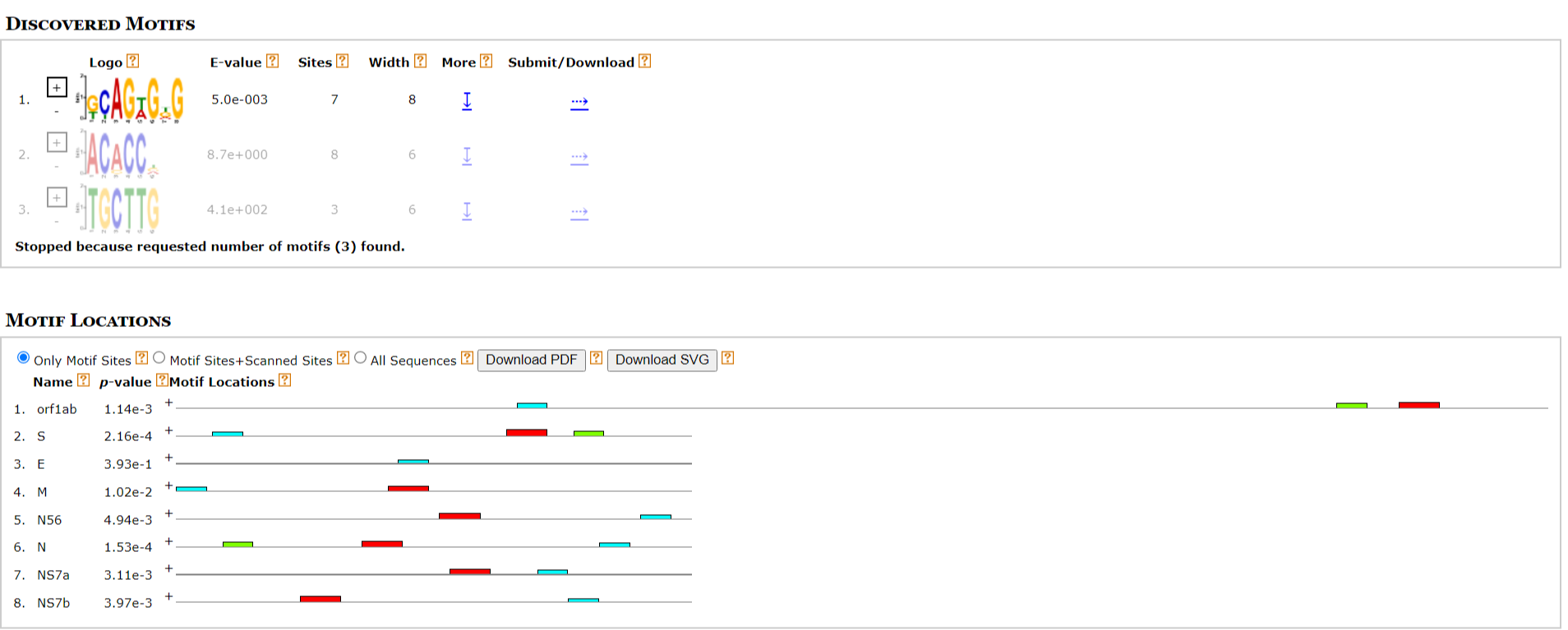
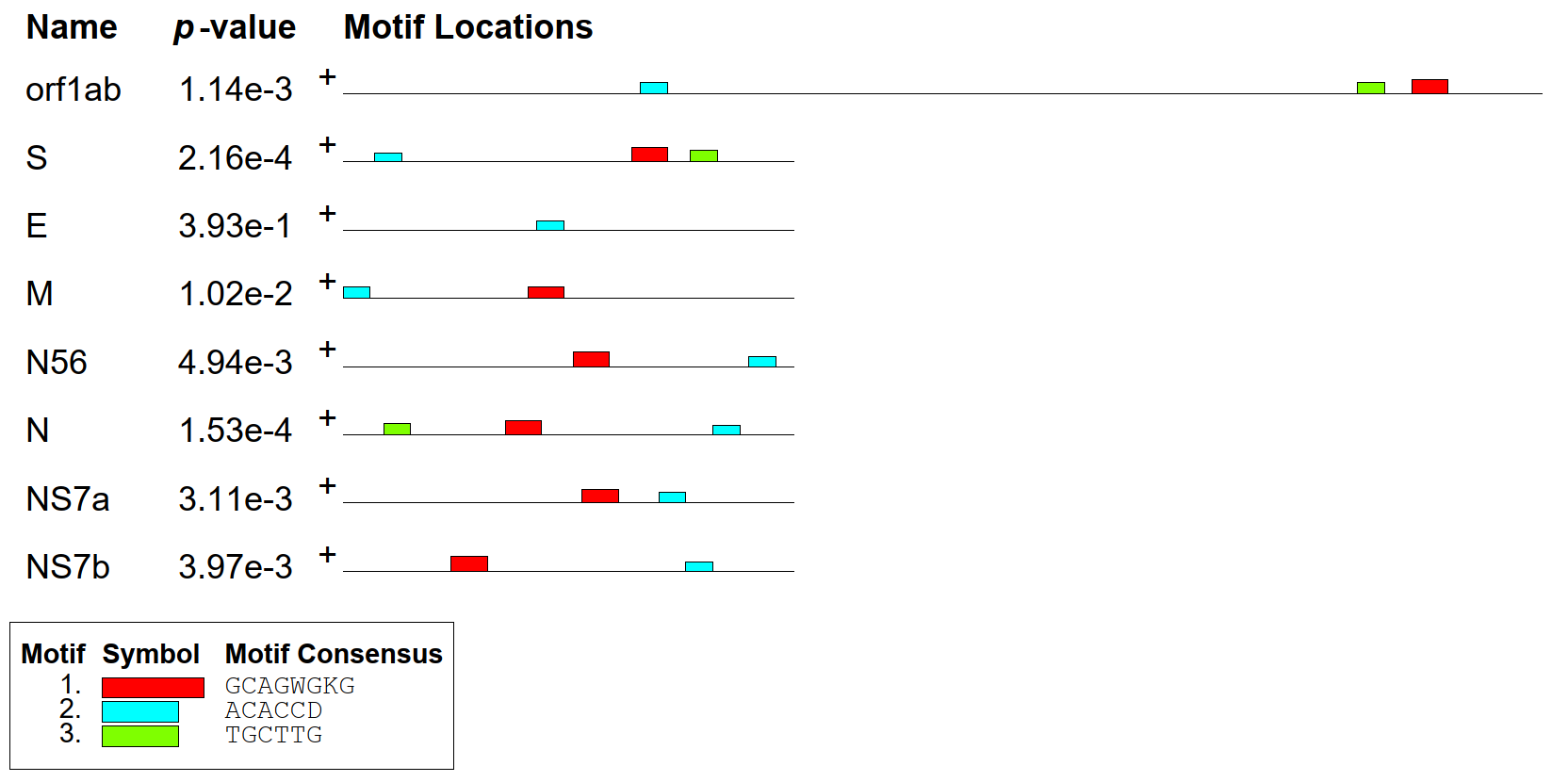
В итоге, нашлось три мотива (выдача, см. рис.1). Также видим из рис.3, что первый мотив нашёлся во всех последовательностях, за исклюением третьей; второй - во всех, а третий - в первой, второй и шестой.
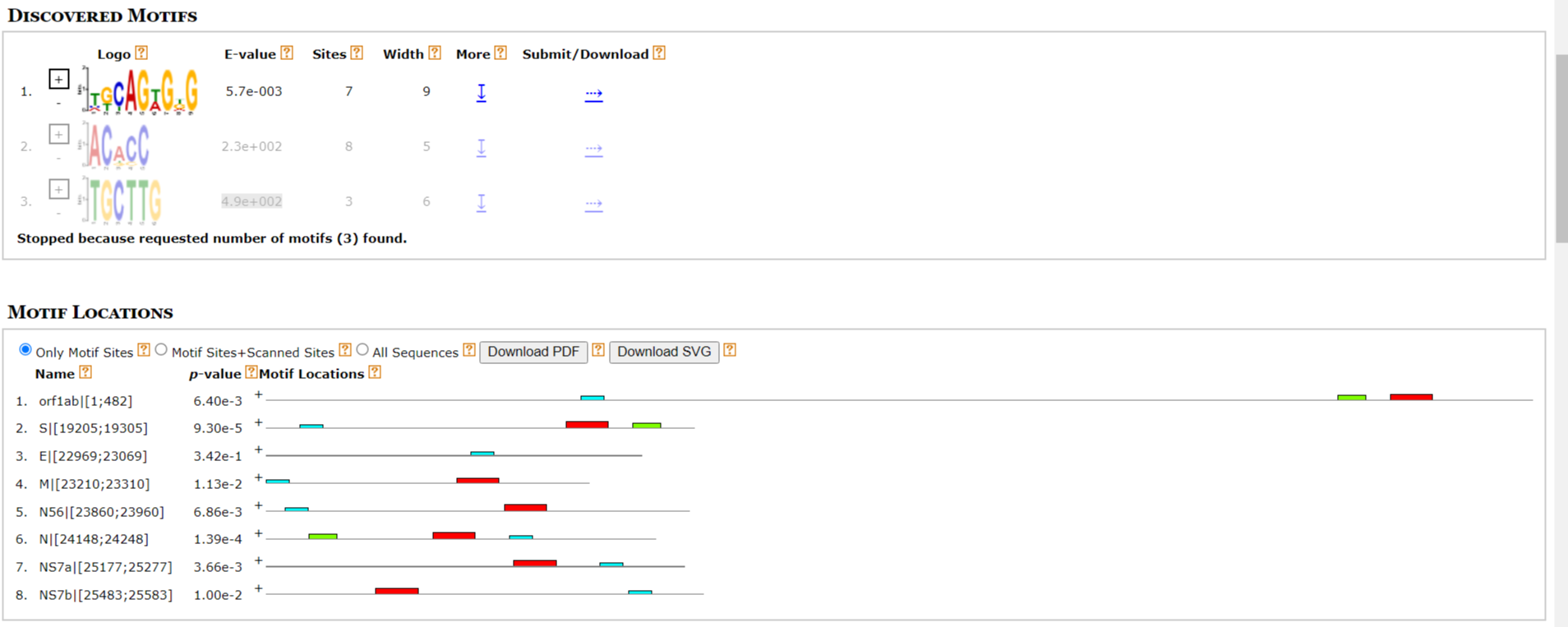
E-value: у первого мотива - 5.0e-003, у второго - 8.7e+000, у третьего - 4.1e+002. У второго и третьего мотива довольно большие E-value, поэтому попробовали изменить длину последовательностей (выдача); E-value снизился у первых двух мотивов, а у третьего даже увеличился. Дальнейшие попытки изменения входных параметров (в конце концов, минимум длины поставили на 3, максимум - на 10) и изменения длины последовательностей (fasta-файл с редактурой) привели к увеличению E-value у третьего мотива (4.9e+002), но у второго хотя бы до 2.3e+002 снизился (рис.2)... Локализация мотивов у N56 после редактуры поменялись местами, особо значительных перемен в локализации более не произошло.
Мотивы представлены на рис.4-6. MEME также выдаёт в качестве выходного файла PWM-матрицу каждого мотива (ссылка).
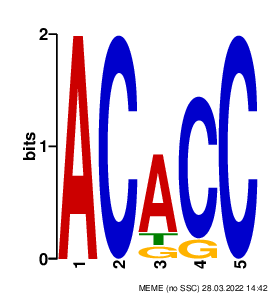

Затем воспользуемся программой FIMO для поиска сигналов в последовательности по PWM этого сигнала прямо на сайте, где работали с MEME. Задаём в качестве базы геном выбранного коронавируса (AC: NC_016994.1), а в качестве мотива - самый первый мотив (т.к. часто встречается и имеет низкое E-value). Указали в параметрах, что ищем только в одной цепи. Результат работы - tsv-таблица, которая представлена и в таблице 1 (первые четыре колонки из изначальной выдачи удалены, чтобы не перегружать эту табличку количеством не совсем нужной информации и не дублировать одно и то же по 8 раз). Совпадения нашлись на прямой цепи.
Нужный нам мотив нашёлся на 8 сайтах, что совпадает с результатом выдачи MEME, p-value у находок низкий, следовательно, сигнал достоверный.
| Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|
| 19269 | 19277 | 9.05e-06 | 0.0815 | TGCAGAGGG |
| 23321 | 23329 | 1.25e-05 | 0.0815 | TGCAGAGTG |
| 23911 | 23919 | 1.25e-05 | 0.0815 | TGCAGAGTG |
| 24184 | 24192 | 1.25e-05 | 0.0815 | TGCAGAGTG |
| 25230 | 25238 | 1.58e-05 | 0.0823 | TTCAGTGGG |
| 25507 | 25515 | 4.81e-05 | 0.209 | AGCAGTGTG |
| 237 | 245 | 7.96e-05 | 0.27 | CTCAGTGGG |
| 5023 | 5031 | 8.28e-05 | 0.27 | CTCAGTGTG |