Практикум 4. Валидация
Задание 1
В этом и следующих заданиях работали со структурой с PDB-ID 5C8T, представляющей собой комплекс неструктурных белков nsp14-nsp10 коронавируса SARS-CoV, вызывающий острый респираторный синдром, с лигандом S-аденозилметионин (SAM). Неструктурные белки (nsp1-nsp16) образуются в результате саморасщепления полипротеина репликазы[link]. На сайте Uniprot упомянуты следующие функции данных белков, входящих в состав полипротеина репликазы 1ab. Неструктурный белок 14 (nsp14) нужен во время процесса репликации РНК. У него две активности: гуанин-N7-метилтрансферазная активность способствует формрованию кэп-структуры (m7G) на 5`-конце молекулы через взаимодействие с S-аденозилметионином, как корректирующая экзонкулеазная активность уменьшает чувствительность коронавируса к мутагенам. Неструктурный белок 10 (nsp10) стимилирует гуанин-N7-метилтрансферазную, а неструктурный белок 16 (nsp16) - 2'-O-метилтрансферазную активность, важен для метилирования кэп-структуры РНК.
Из отчёта по валидации wwPDB можно узнать информацию о метриках качества:
- Rfree = 0.256
- Clashscore = 31
- Ramachandran outliers = 0.2%
- Sidechain outliers = 17.8%
- RSRZ outliers = 3.4%
- Rvalue = 0.243
- Разрешение = 3.20 Å (удовлетворительное)
Разница между Rfree и Rvalue по модулю составляет 0.013 (1,3%) (модель не переоптимизирована), однако сами эти значения по отдельности меньше 40% и больше 20%. Это показывает не самое лучшее, средненькое качество модели. У модели не самые лучшие значения второго и четвёртого показателя из вышеприведённого списка.
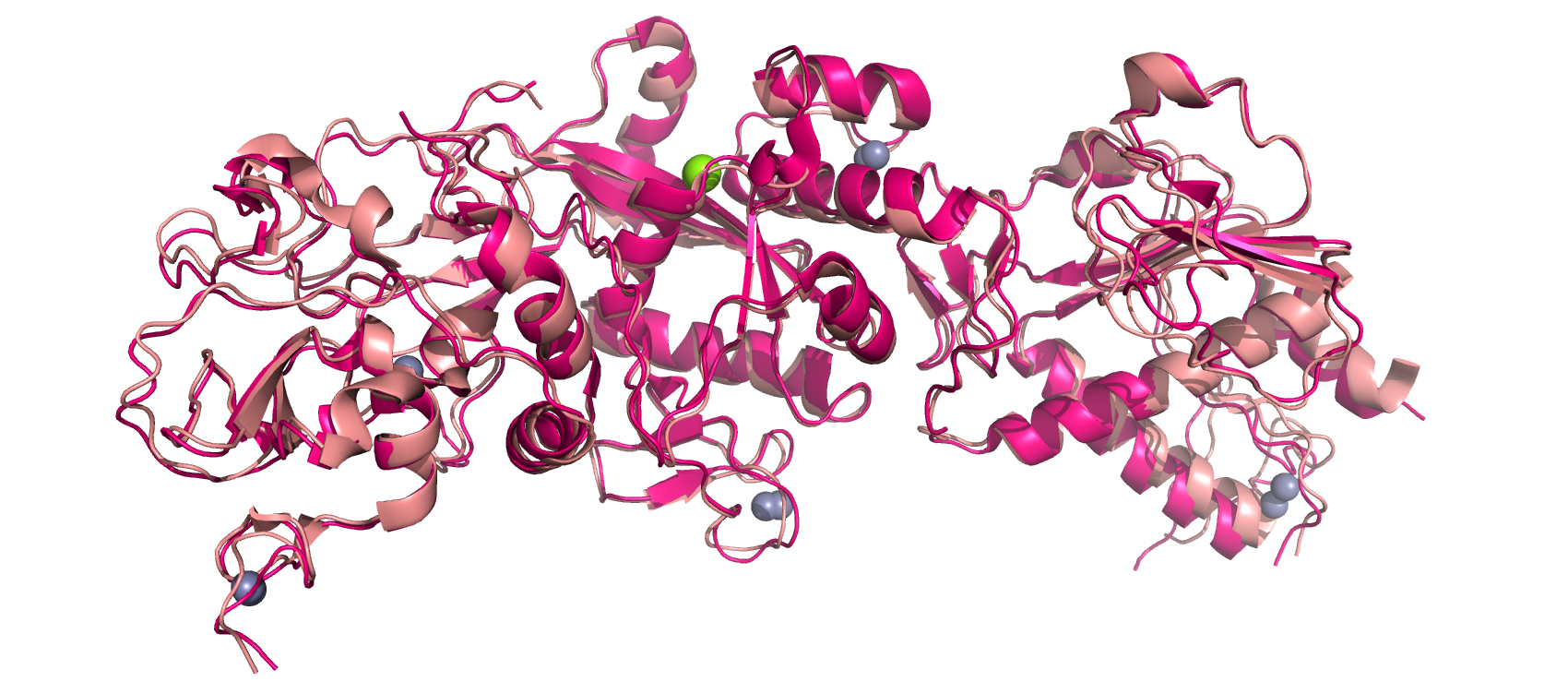
Посмотрев на электронную плотность структуры, можно заметить, что есть малочисленные участки (в основном, это боковые радикалы аминокислот), не вписавшиеся в неё, на поверхности белка. Эти участки, судя по данным из Uniprot, не имеют никакой функциональной значимости, поэтому не берём их в расчёт. На рис.1 представлены ЭП двух биологических сборок: A - первая сборка (цепи А и В), B - вторая сборка (цепи C и D). Цепи А и С - неструктурный белок 10 коронавируса, цепи B и D - гуанин-N7-метилтрансфераза коронавируса, которые записаны под одним AC Uniprot (P0C6X7), поскольку находятся в составе одного полипротеина. Выровнив эти две сборки друг на друга, оказалось, что они выровнились с небольшим сдвигом относительно друг друга (рис.2), а также есть различия в конформации выпетливаний, однако аминокислоты там одни и те же, поэтому не столь важно. Альтернативных структур нет, судя по данным из БД PDB. Однако нельзя точно говорить о том, что данная модель пригодна для анализа структуры белка, если принимать в расчёт её не очень хорошие показатели качества в целом.
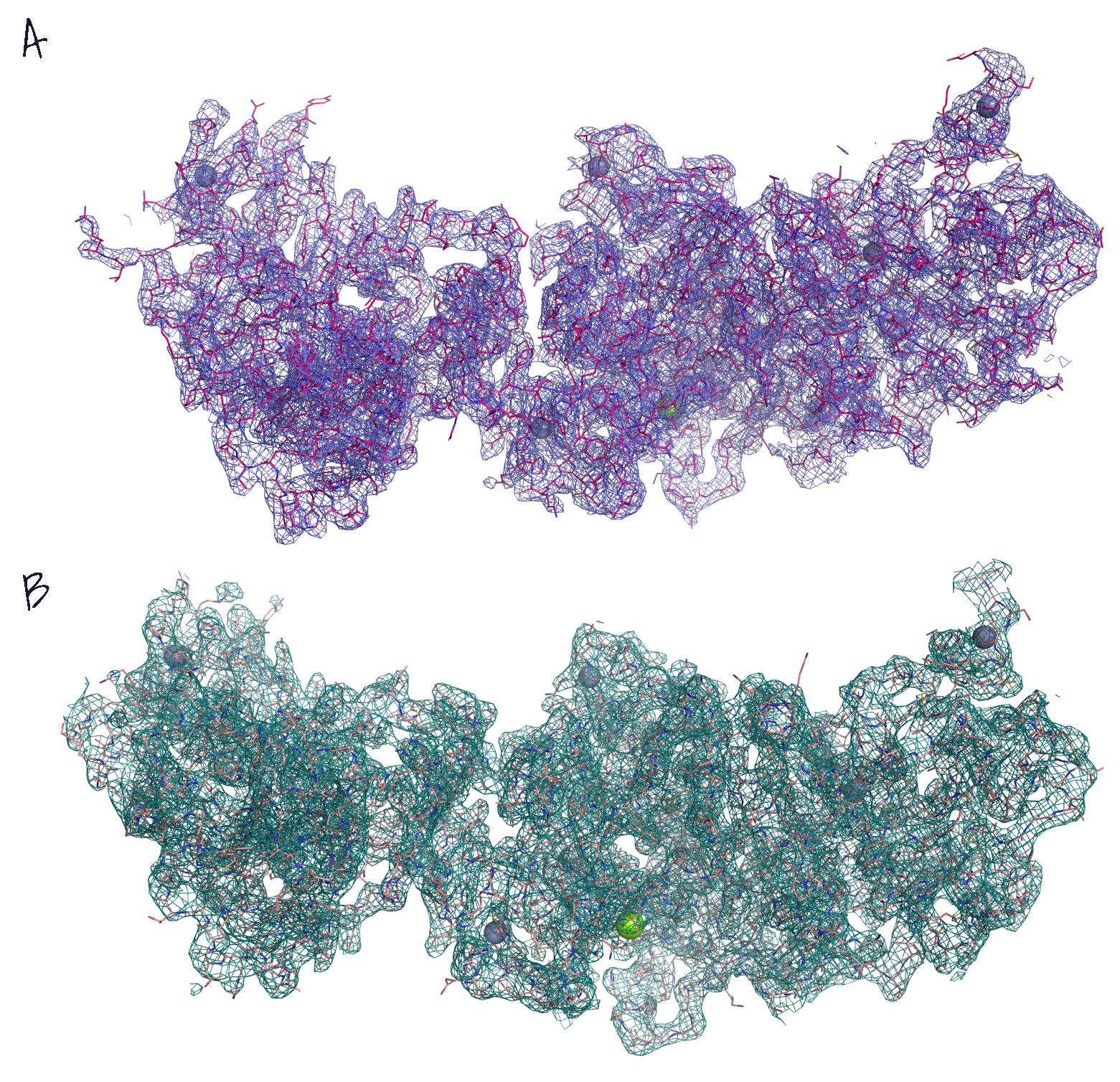
Уровень подрезки (σ) = 1, carve = 2
Задание 2
Далее отбирали пять аминокислотных остатков, ионов или молекул воды, маргинальных по различным показателям. Для этого просмотрели отчёт по валидации: таких маргиналов оказалось довольно много. Доли аминокислотных остатков, имеющие отклонения по нескольким параметрам, для nsp10 (первой сборки) следующие: цепь А: ~42% по одному параметру, ~8% - по двум, ~8% - остатки, отсутствующие в модели, но не в образце (на графике в отчёте приведены округлённые величины); цепь С: 41% - по одному параметру, 7% - по двум, 8% - остатки, отсутствующие в модели, но не в образце. Для гуанин-N7-метилтрансферазы (второй сборки): цепь В: 41% - по одному параметру, 10% - по двум; цепь D: 40% - по одному параметру, 11% - по двум.
Информация по критериям в разделе "Качество модели":
- В цепи В маргинал по длине связи - Cys-207;
- В цепи В маргиналы по углам: Cys-207, Ala-85, Val-29;
- Маргиналов по хиральным центрам нет, но есть по планарности (в скобках - названия цепей): Lys-87 (A), Gly-88 (A), Asp-375 (B), Cys-473 (B), Thr-5 (B), Arg-84 (B);
- Маргиналов по перекрыванию Ван-дер-Ваальсовых радиусов в одной ассимметрической ячейке всего 627, между ячейками всего 2;
- Маргиналов по торсионным углам всего 3 (цепь В);
- 199 маргиналов-аминокислот с неротамерными боковыми цепями; из них 22 можно было бы повернуть для того, чтобы обеспечить стабильную водородную связь и убрать перекрывания. (+ ион + инверсия)
Воспользуемся сервисом MolProbity для анализа геометрии (без водородов и с водородами и заменой инверсий Gln/Asn/His (рис.3)) и посмотрим на сводную таблицу по остаткам. Сервис предсказал куда больше маргиналов по ротамерам (214) и чуть больше по длинам связей (отличие с wwPDB составляет одну аминокислоту) и углам (на четыре аминокислоты различается). После замены инверсий и добавления водородов численные показатели структуры в сводной таблице не улучшились.
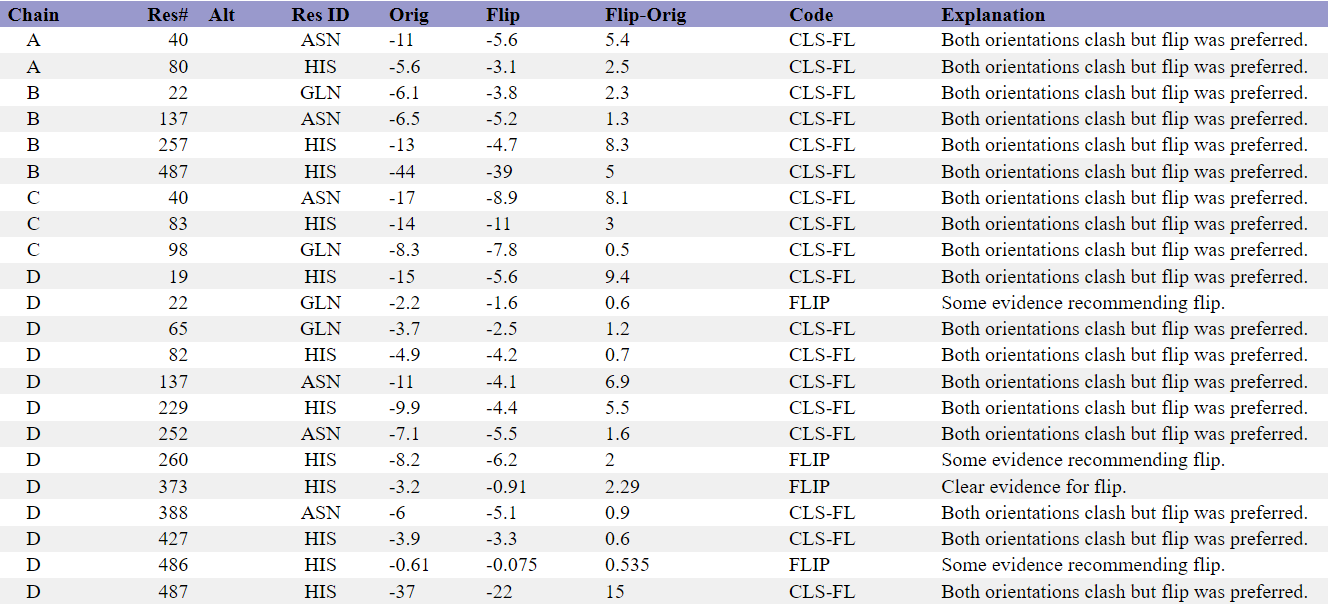
Рассмотрим выбранные маргиналы (ссылка на сессию):
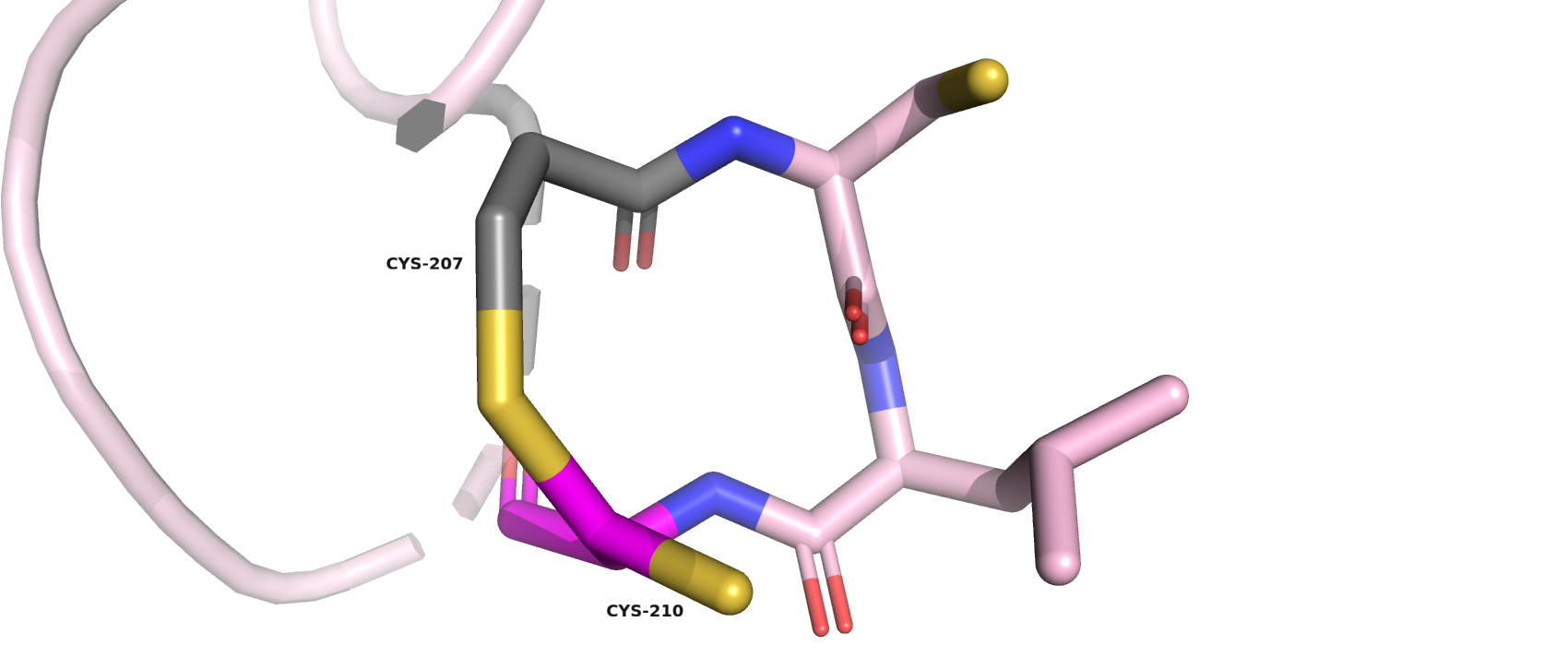
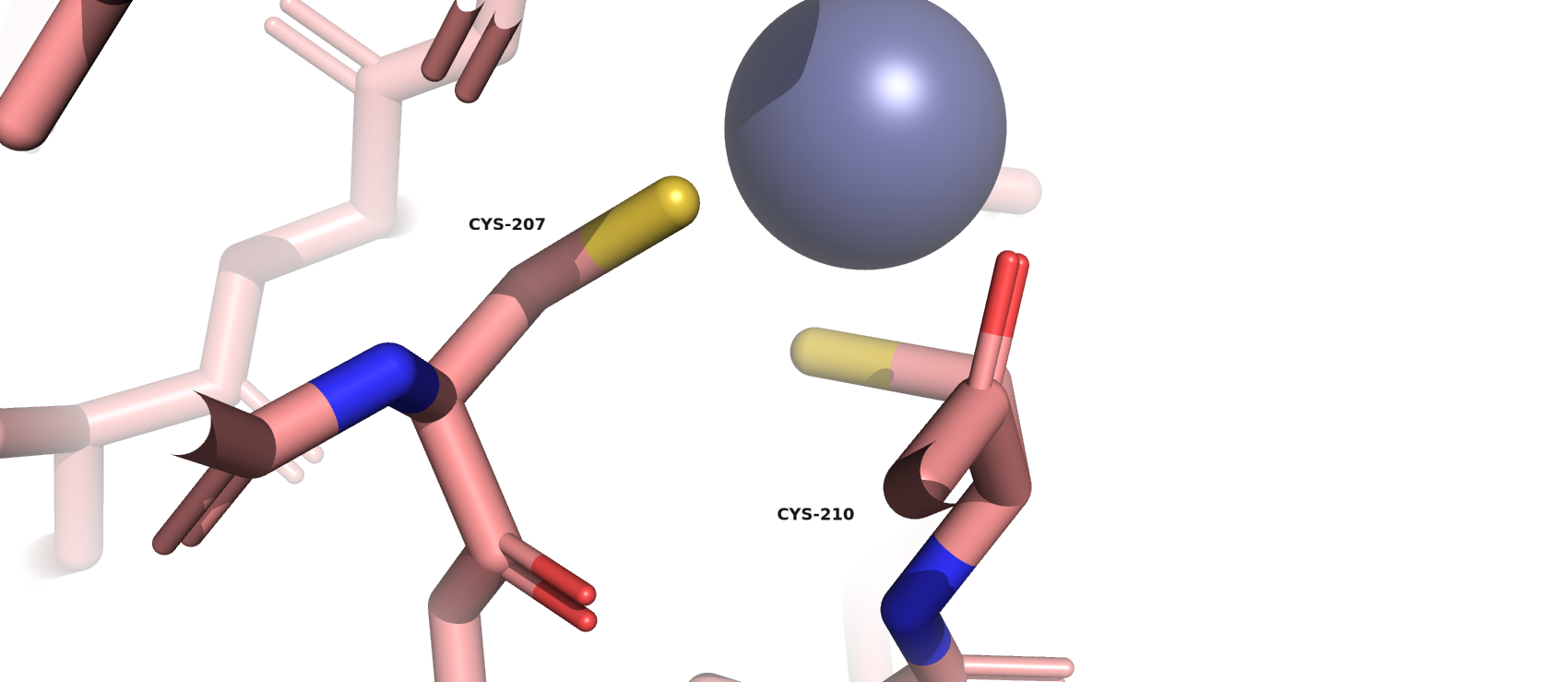
- Cys-207 (цепь В) является маргиналом по длине связи между атомами Cβ-Sγ (набл. = 1.93 Å, идеал. = 1.82 Å) и по углам (в градусах) (набл. = 128.40, идеал. = 114.00), однако в выдаче MolProbity он значится маргиналом только по значению угла между атомами Cα-Cβ-Sγ. В изначальной структуре он соединён связью с Cys-210 через серу (рис.4), что само по себе очень странно... Получается, что эти два остатка настолько близко расположены, что программа рисует между ними связь. Тут уже скорее ошибка авторов, которые эту структуру и загружали.
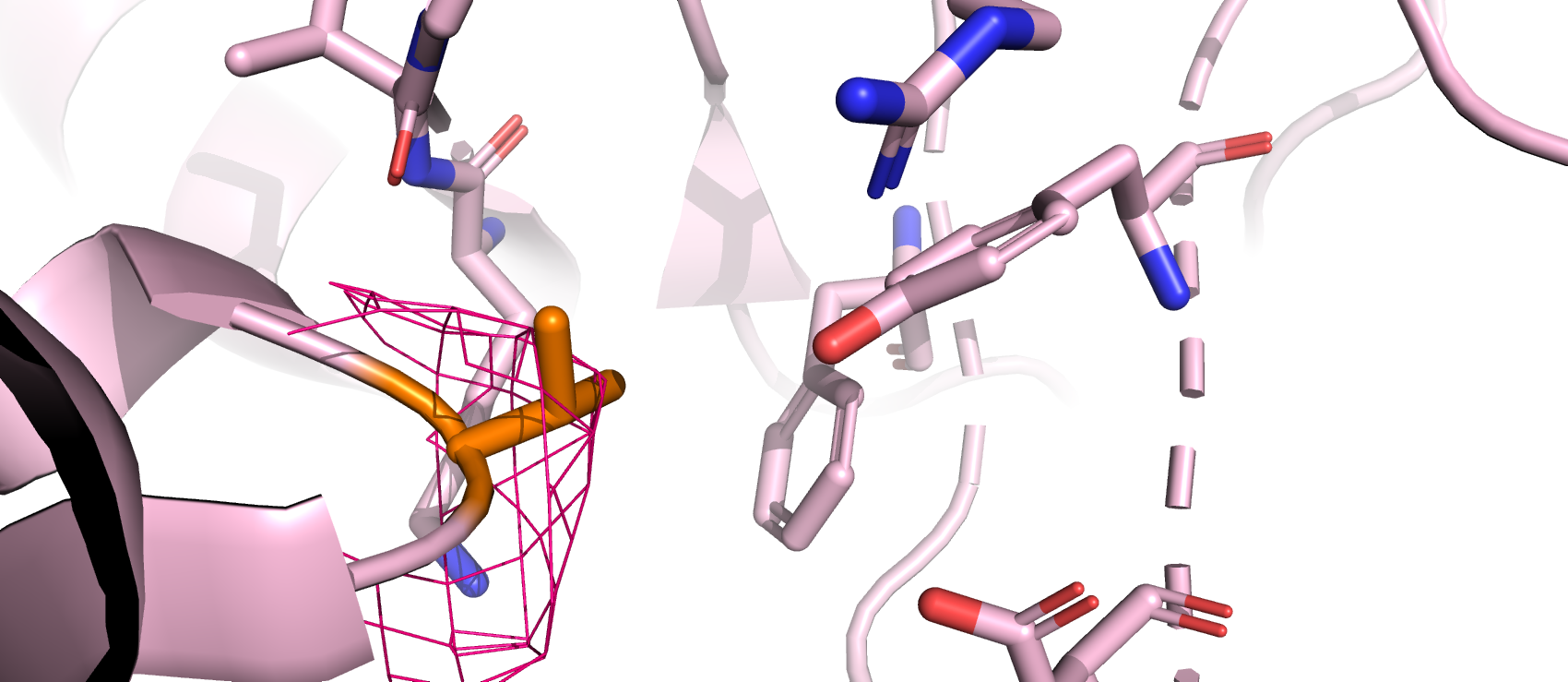
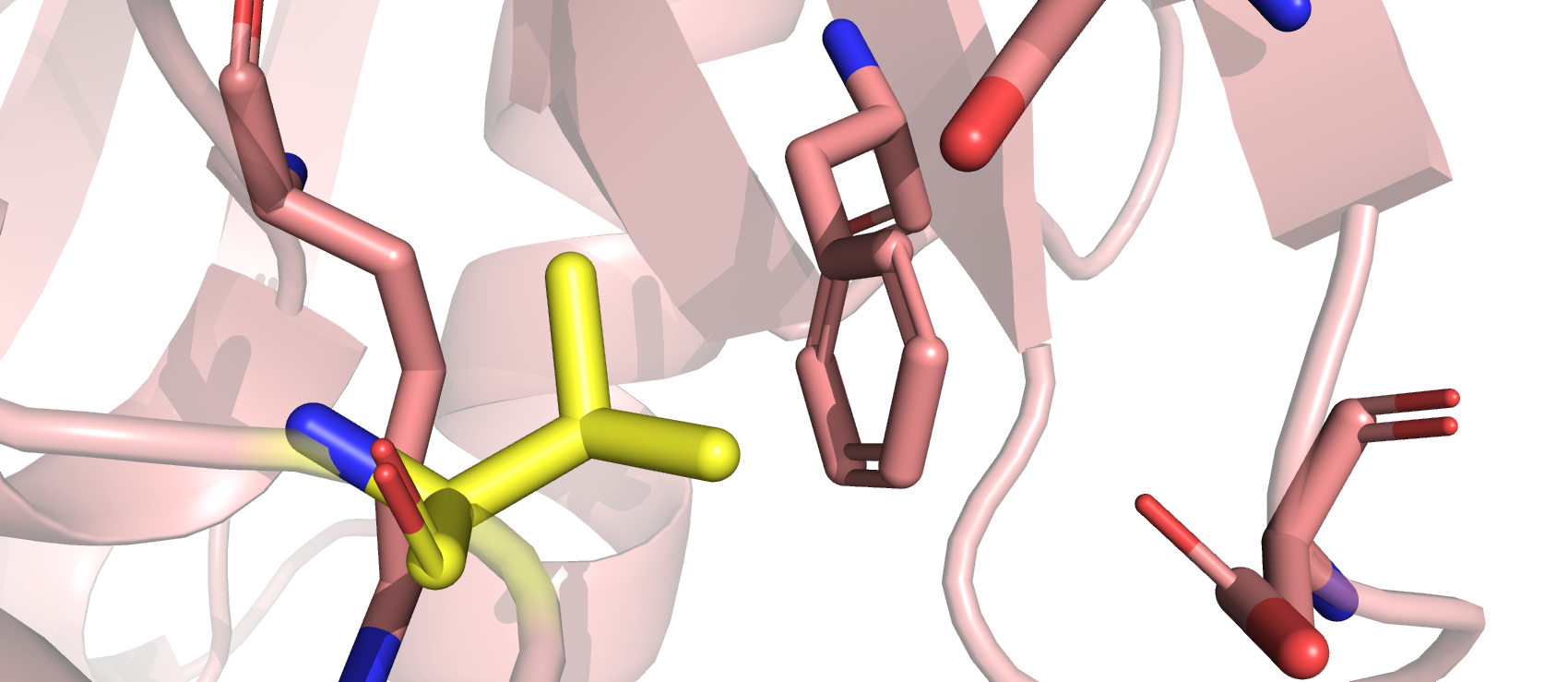
- В цепи А Val-21, судя по данным wwPDB и MolProbity, - маргинал с нарушением ротамерной структуры боковой цепи. Рассмотрев окружение валина и его ЭП (рис.5), можно сказать, что ЭП не полностью покрывает его, а вокруг него находится гидрофобный фенилаланин (у изолейцина боковой радикал смотрит в другую сторону) и глицин, тирозин, аргинин, аспарагин (два последних расположены дальше тирозина). Исходя из всего этого вряд ли можно предположить какие-то взаимодействия (кроме двух (с натяжкой) гидрофобных), которые стабилизируют данный маргинал; возможно, что имеет место быть ошибка расшифровки РСА.
- В цепи B Arg-135 - маргинал с нарушением ротамерной структуры боковой цепи. В его окружении два гистидина, с одним из которых возможны водородные связи (чтобы не было перекрываний), и валин. ЭП хорошо легла на аминокислоту. Окружение данной аминокислоты и она сама находятся на периферии белка, и они не играют важной функциональной роли.
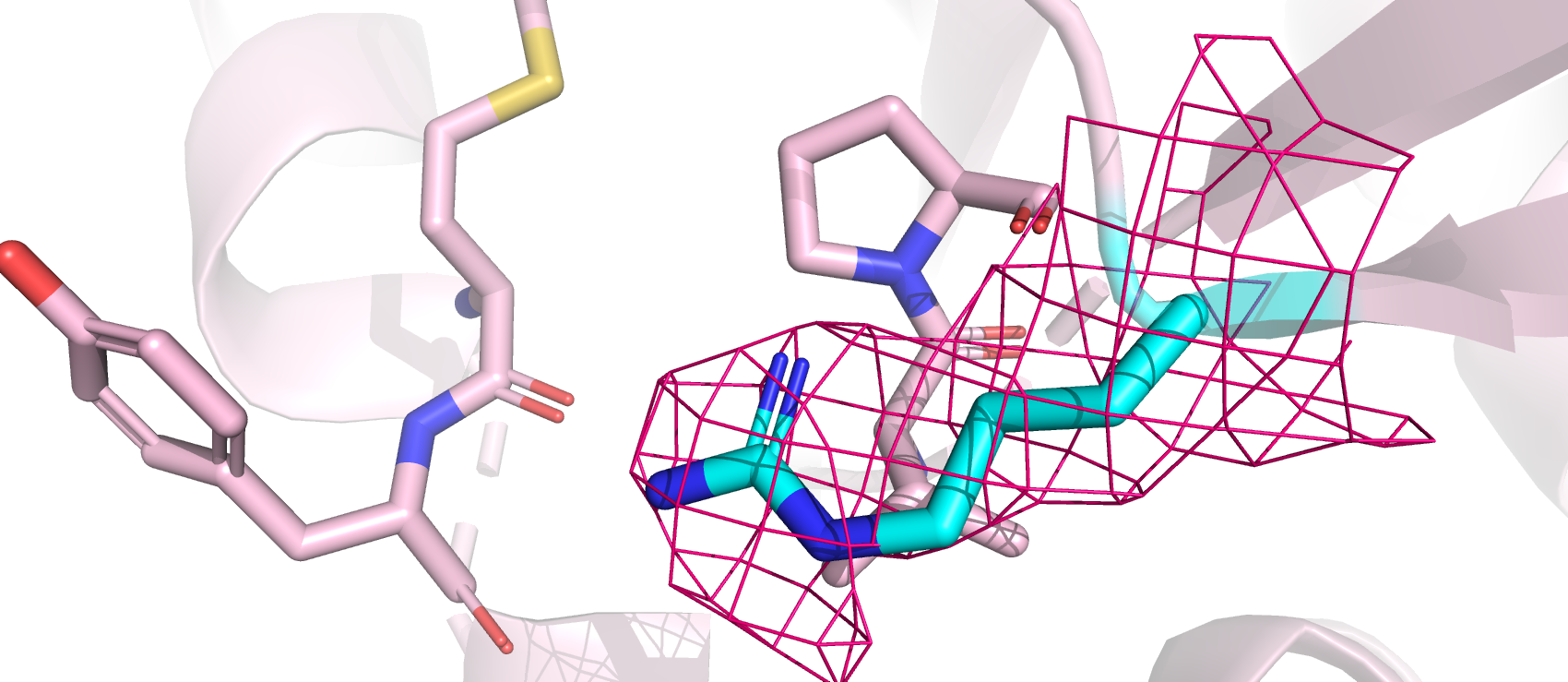
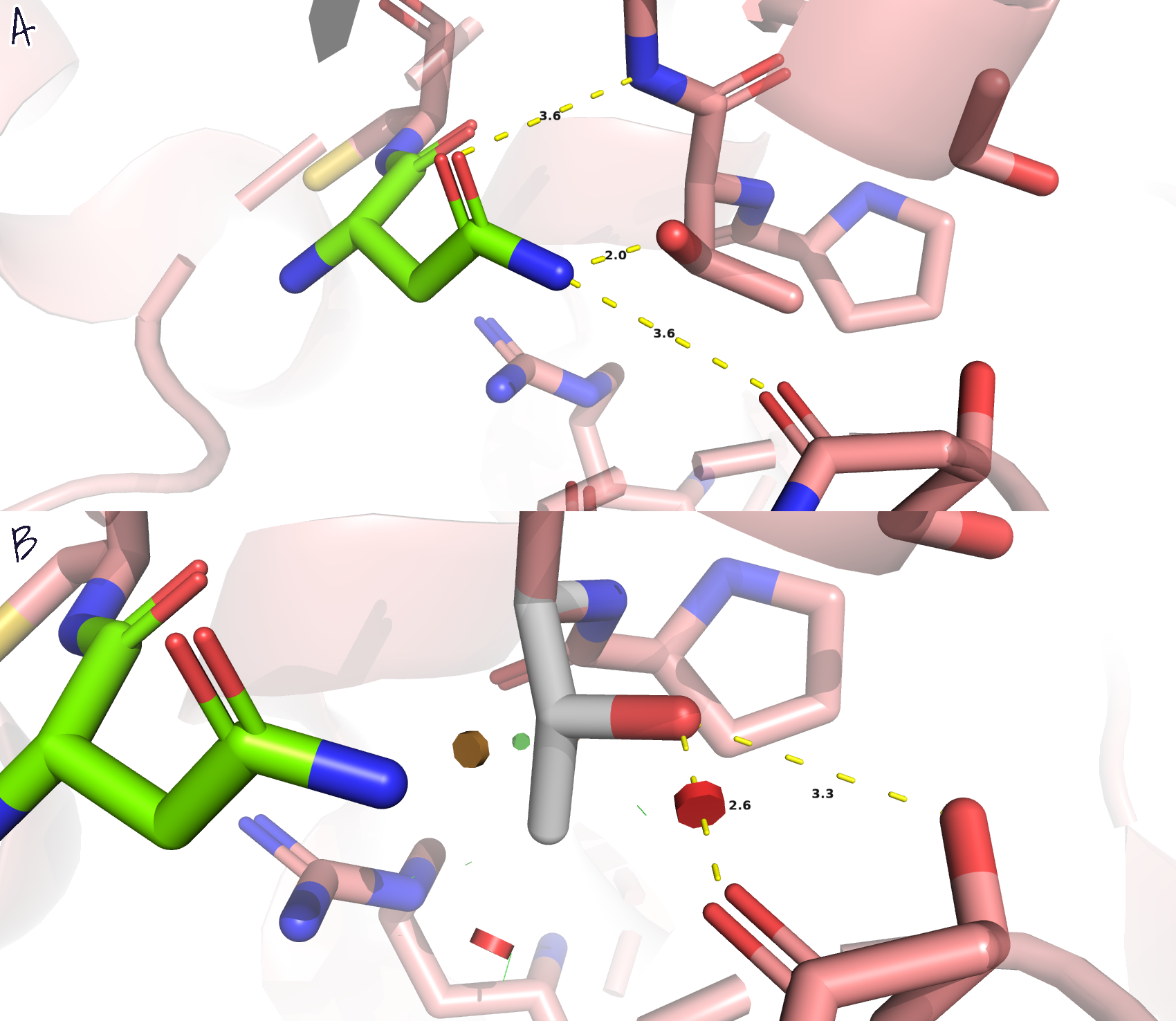
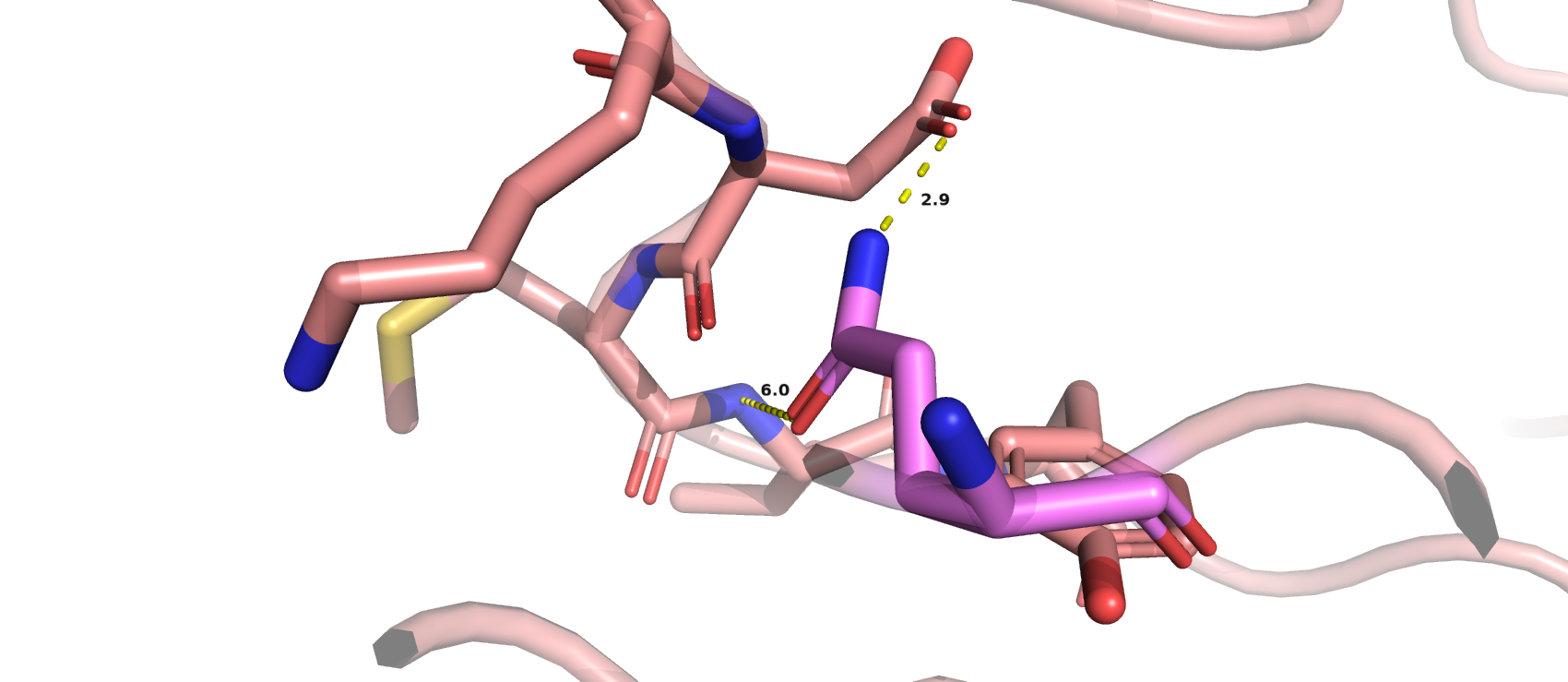
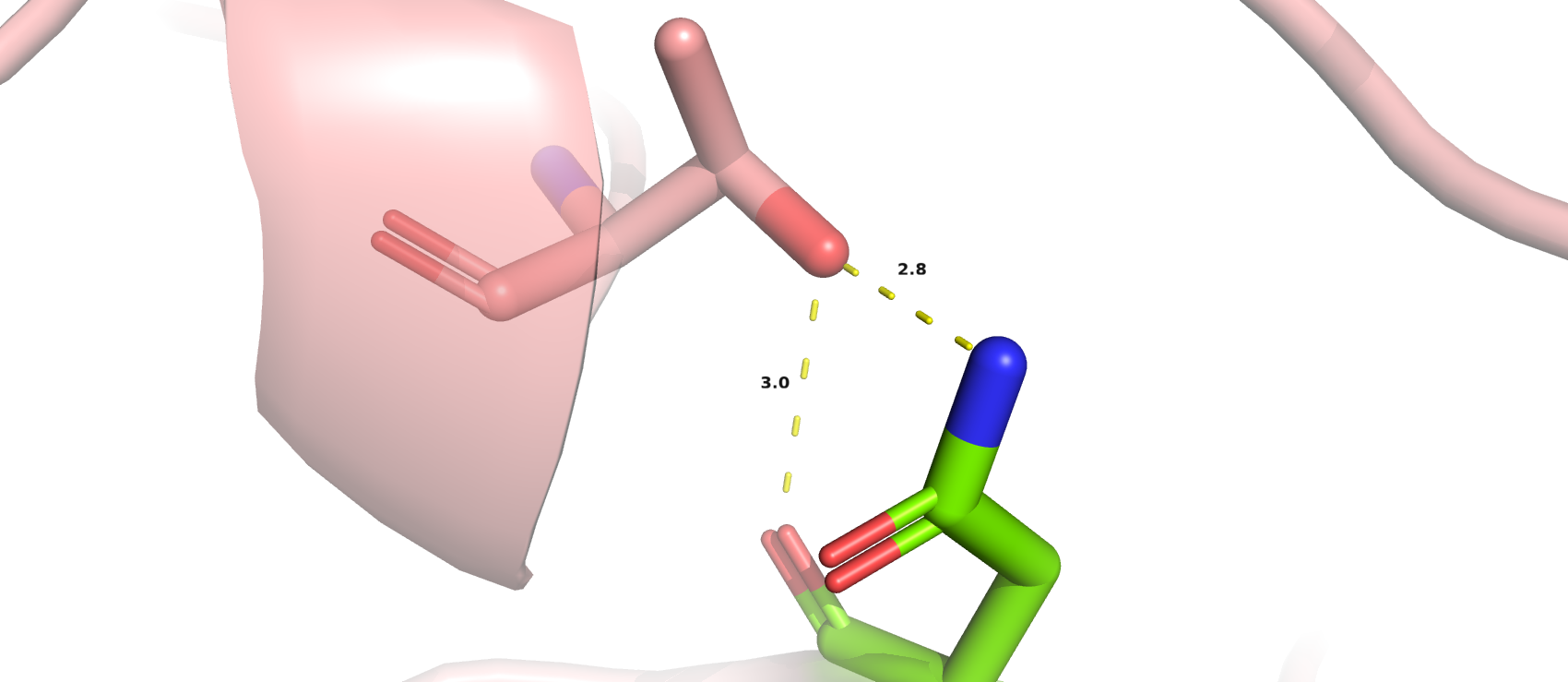
- Рассмотрим Gln-22, для которого MolProbity и wwPDB предсказали инверсию. Электронная плотность полностью покрывает остаток. Рассмотрев окружение аминокислоты (рис.7А), можно заметить, что она будет связана водородными связями с тирозином и атомами остова. Однако было показано, что водородная связь между глутамином и лизином не может быть, поскольку лизин находится на расстоянии больше 3-3.5 Å, как и одной водородной связи между глутамином и остовом аспартата (3.7 Å). Попробовали повернуть глутамин и посмотреть, какие взаимодействия могут появиться для стабилизации данного остатка. Так появилась водородная связь между ним и остовом аспарагиновой кислоты (рис.7В) в пределах вышеуказанных значений, но ни один ротамер глутамина не может образовать водородную связь с лизином (из-за далёкого расстояния и возможного перекрывания с другими остатками). Так что, возможно, есть ошибка в расшифровке РСА данных.
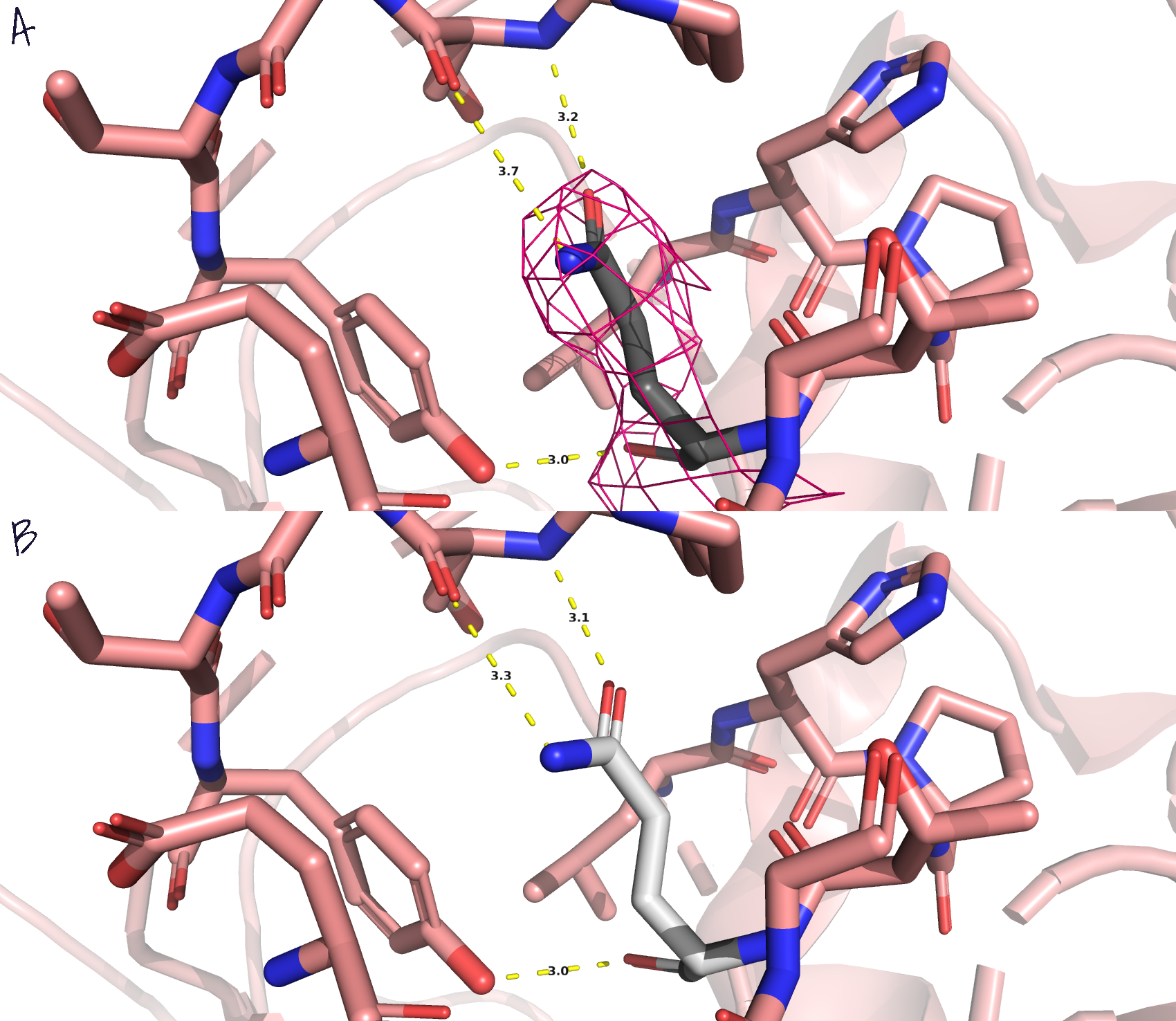
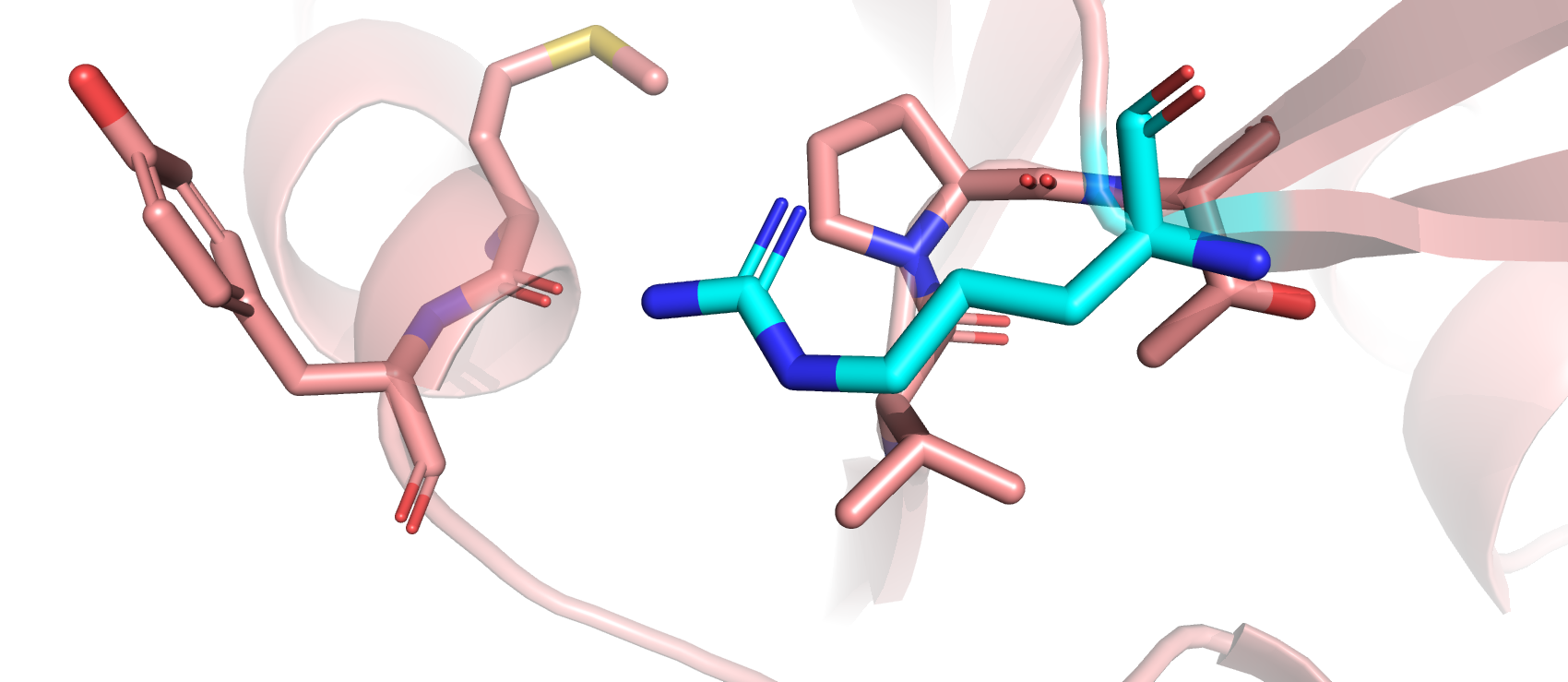
- Наконец, рассмотрим взаимодействия между Asn-40 и Thr-25 (цепь С и D, соответственно), для которых программы предсказали перекрывания Ван-дер-Ваальсовых радиусов атомов азота и кислорода, соответственно (рис.8). А также в отчёте по валидации Thr-25 (цепь D) является маргиналом с нарушением ротамерной структуры. Попробовав повернуть треонин, ситуация с перекрыванием стала несколько лучше, однако появилось перекрывание с остовом (красный диск между остатком и остовом на рис.8).
Задание 3
Дальше посмотрели, есть ли обновлённая структура в PDBRedo. Обновлённый показатель Rfree равен 0.2273, что уже лучше по сравнению с оригинальной версией. Обновлённая структура намного лучше с плане качества и количества/наличия маргиналов, перечисленных выше, чем старая структура.
- Атом серы Cys207 больше не связан с углеродом Cys210.
- Изменён ротамер Val-21.
- Arg-135 остался без изменений.
- У Gln-22 изменился ротамер, но был выбран не самый лучший с точки зрения образования водородных связей: одна связь невозможна, так как её длина превышает вышеупомянутых значений. По этим же причинам связи между азотом глутамина и остовом быть не может. Поэтому предложенный вариант (рис.7В) куда лучше смотрится.
- Ситуация с перекрыванием между Asn-40 (цепь С) и Thr-25 (цепь D) исправилась следующим образом: был изменён ротамер треонина так, что перекрывания ван-дер-ваальсовых радиусов исчезли, а также произошла инверсия остова. Данный ротамер треонина поддерживается водородными связями.
Таким образом, подтверждается гипотеза о том, что ошибки, в основном, кроются в неточной расшифровке результатов РСА, однако есть совсем плохие случаи, когда сами авторы не перепроверили структуру на наличие странных связей (Cys-207 и Cys-210). В случае с Arg-135 в новой и старой структуре он остался без изменений, значит, ошибка была не в расшифровке РСА. В общем отчёте по валидации обновлённой структуры было прописано количество исправленных маргиналов: 18 аминокислот с нарушением ротамеров, 52 инверсии полипептидного остова, у 482 остатков изменена ЭП в лучшую сторону, но у 5 в худшую. Разрешение чуть-чуть улучшилось (3.18 Å), но не сильно (прежнее значение составляло 3.20 Å).