Практикум 5. Разметка взаимодействий
Задание 1
В этом задании работали со структурой с PDB-ID 5FBD, представляющую собой комплекс нуклеазы S1 с фосфатом и 2'-дезоксицитидином. Данный комплекс был выделен из Aspergillus oryzae. Структура была получена при pH = 3.8 и температуре 291°K. Разрешение составляет 1.75 Å. Нуклеаза S1 является ферментом из класса гидролаз (Zn2+-зависимый фермент, оптимум работы при рН = 4.0-4.3). Фермент гидролизует связь между атомами P–O3’ в оцДНК и РНК с образованием 5'-фосфорилированных моно- и олигонуклеотидов[1], что позволило генным инженерам использовать его в своей работе с нуклеиновыми кислотами (например, для обработки липких концов, чтобы их "затупить"), а также биохимики с его помощью определяют вторичную структуру нуклеиновых кислот.
Целью данного задания является предсказать эффекты, которые произойдут со структурой белка, если увеличить рН. Для протонирования остатков воспользуемся сервисом PDB2PQR. Его встроенный алгоритм PROPKA рассчитывает возможность влияния электростатики от полярных участков молекулы белка на протонирование титруемых химических групп. Запустив алгоритм с нашим белком и параметрами по умолчанию, получим log- и pqr-файл.
В строчке "Summary of this prediction" log-файла представлен вывод программы по предсказанию протонирования. Нам необходимо найти остатки Asp, Glu, His с самыми высокими рКа, которые также выше рНкристаллизации = 3.8.
- Список остатков с экстремальными значениями:
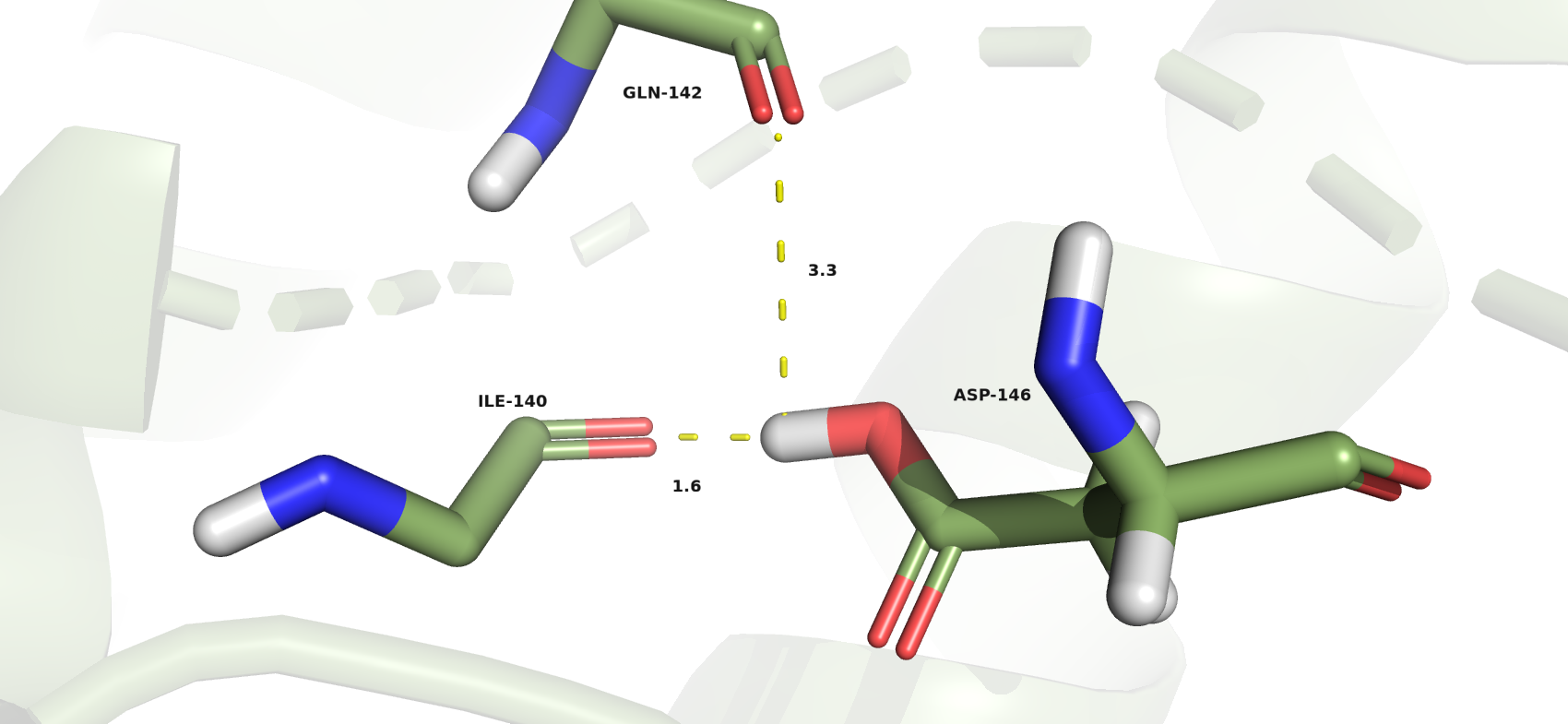
- Asp-146 с предсказанной рКа=10.71. Это выше, чем рНкристаллизации, поэтому при кислых рН он должен быть протонирован, что мы и видим в визуализаторе PyMol. Факт нахождения протона подтверждается наличием водородной связи (рис.1), однако только одна из них возможна ввиду оптимальной геометрии. Протон находится на одной линии с атомом кислорода остова изолейцина 140, а потому именно с ним строится водородная связь, а с глутамином 142 - нет (однако при изменении положения протона связь может появиться, но первая пропадёт). При повышении рН протон исчезнет, а соответственно и водородная связь. Судя по данным из Uniprot, Asp-146 не играет функциональной роли в белке, поэтому, возможно, его функциональность не пострадает.
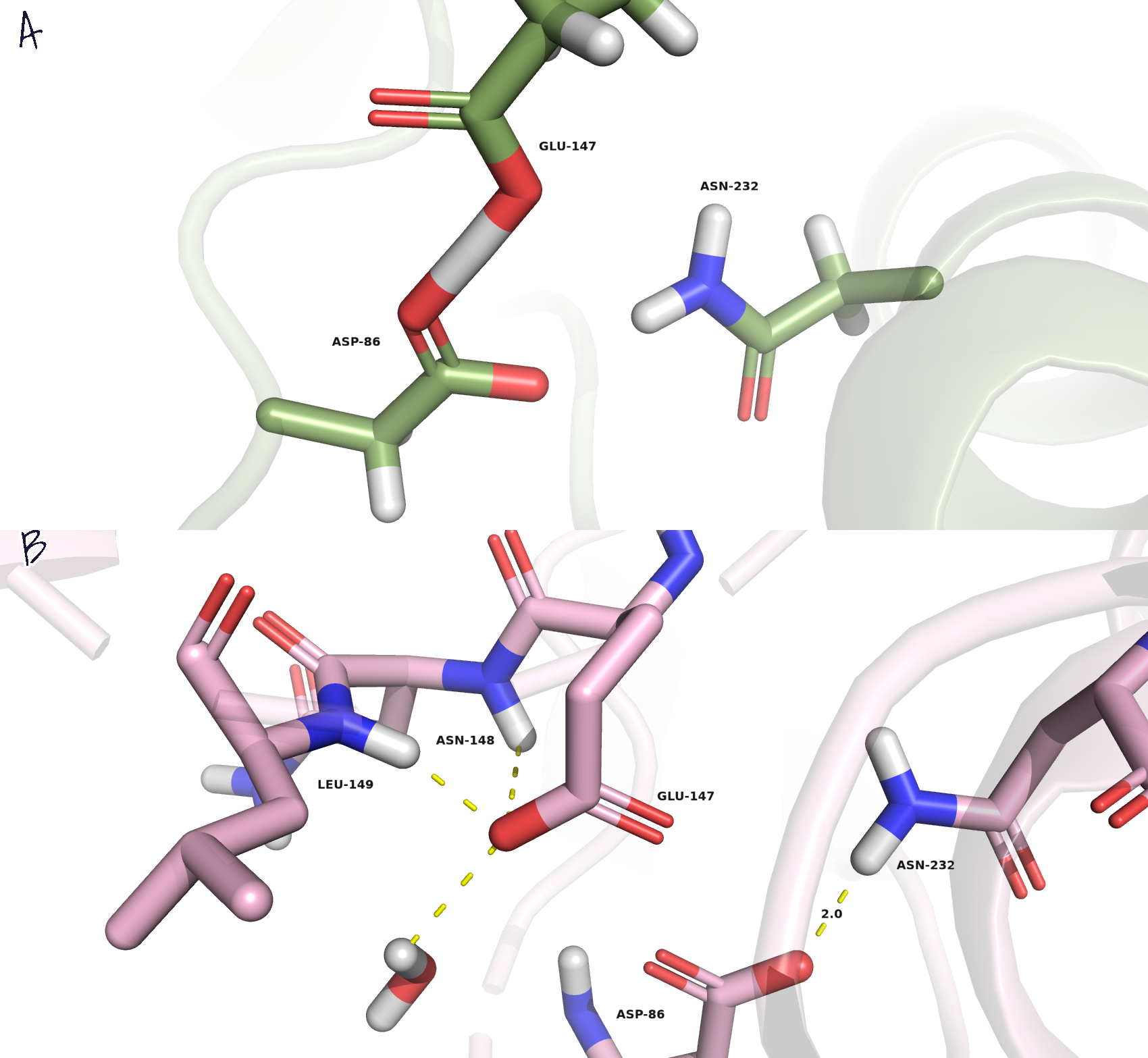
- Glu-147 с предсказанной рКа=8.87. При кислых рН он должен быть протонирован, что и видно в структуре, однако при добавлении водорода возникает возникновение странной (не водородной) связи между боковыми группами Glu-147 и Asp-86, поскольку при предсказании протонирования групп программой PROPKA могут сместиться положения аминокислот, что и случилось в данном случае (рис.2А): у глутаминовой кислоты сменился ротамер при протонировании, из-за чего возникла данная ситуация. В изначальной структуре глутамат смещён относительно аспартата, что показано на рис.2В. Протон на рис.2А, если не принимать во внимание возникновение подобной связи, может быть в структуре, и он поддерживается водородной связью с аспартатом. В изначальной структуре непротонированный глутамат поддерживается куда большим числом водородных связей, поэтому, скорее всего, в нативной структуре более предпочтительна такая форма остатка, а не протонированная. По этой же причине депротонирование при высоких рН приведёт к возникновению связей с другими остатками (например, с указанными на рис.2В, а также с Asn-232).
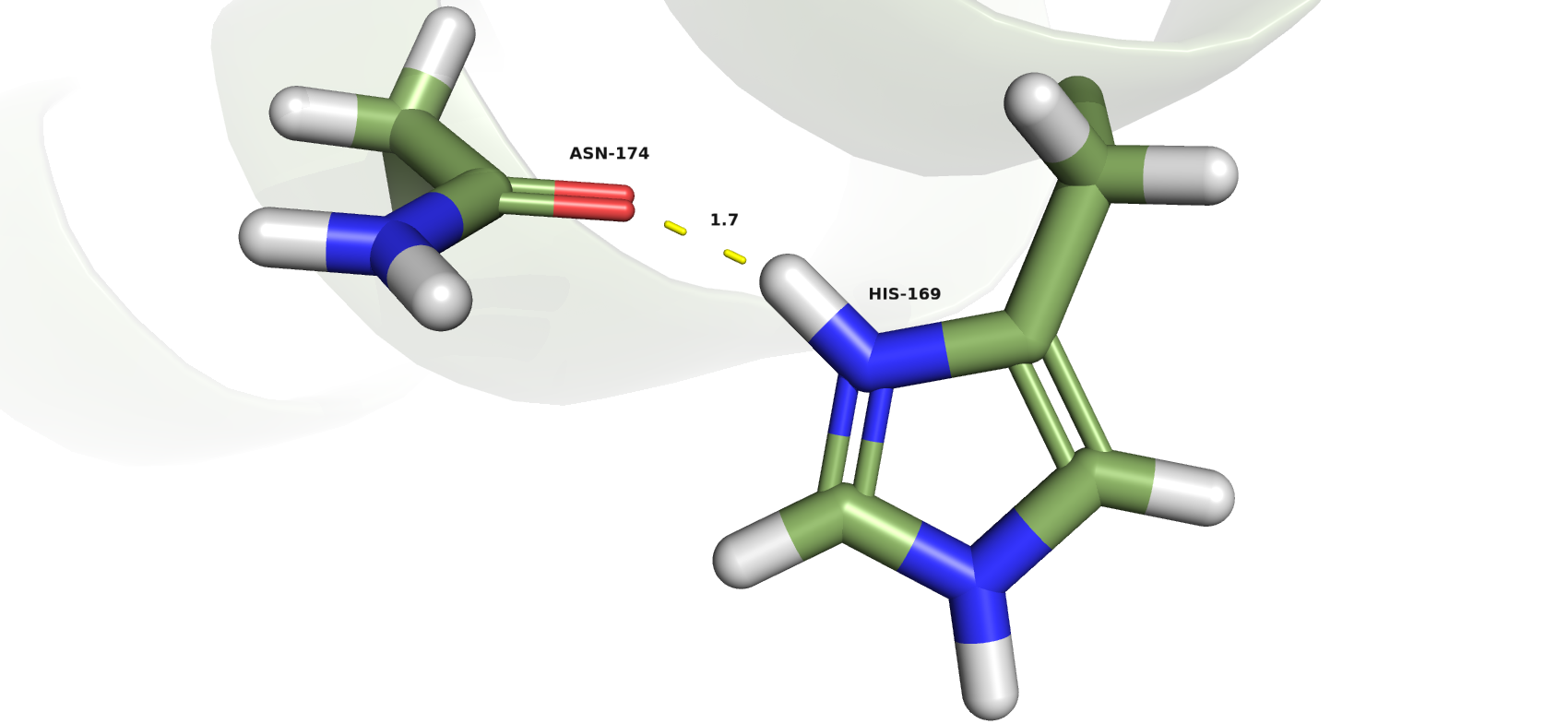
- His-169 с предсказанной рКа=6.52. При кислых рН он должен быть протонирован, что и видно в структуре. В протонированной форме связывается с остовом Asn-174 водородной связью. Боковой радикал гистидина торчит не внутрь белка, а наружу, в сторону раствора, поэтому не участвует во внутрибелковых взаимодействиях и не нарушает структуру. Депротонирование данного аминокислотного остатка особо картины не изменит. Скорее всего, в нативной структуре протона не наблюдается.
Задание 2
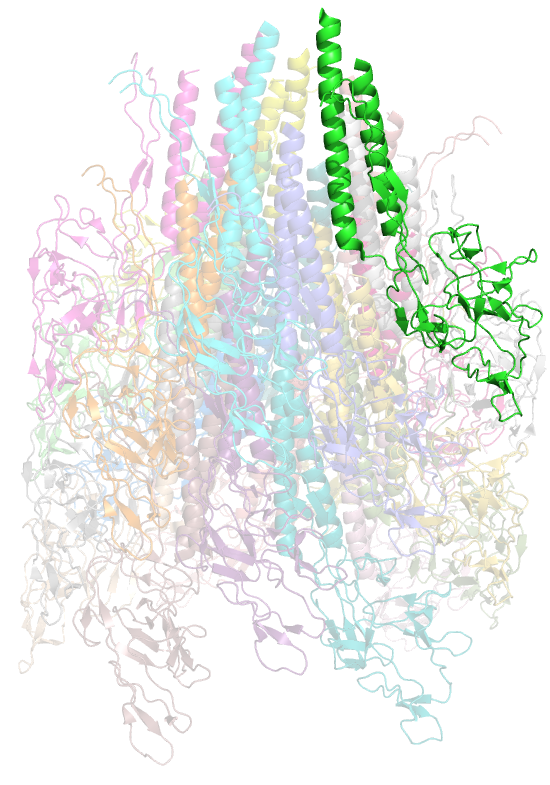
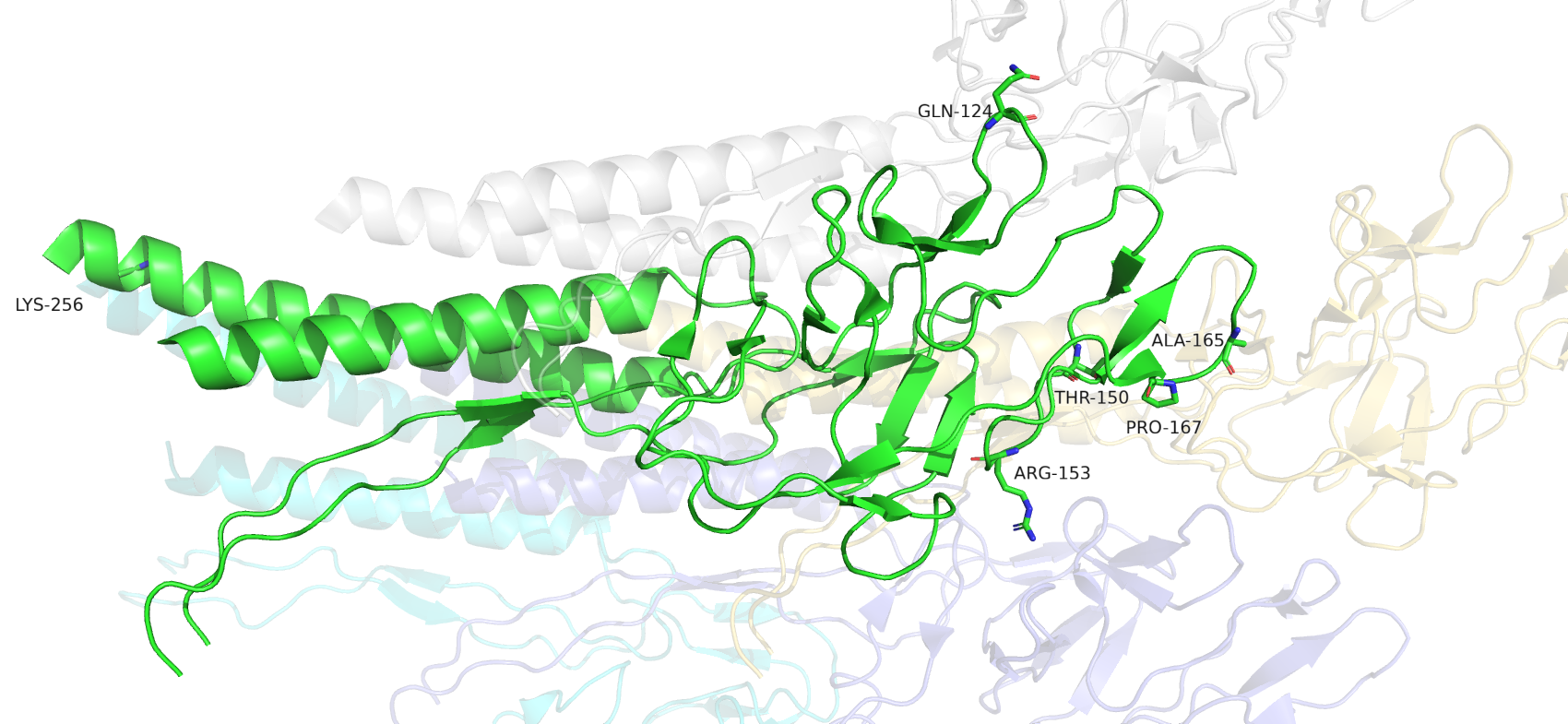
В этом задании работали со структурой с PDB ID 6JZR, представляющую собой стержень жгутика (флагеллы), пронизывающий мембрану бактерии Salmonella enterica subsp. enterica serovar Typhimurium. Данный белок, как указано в описании PDB-файла, является моторным и участвует в движении жгутика бактерии и, соответственно, в её движении в жидкой среде. Структура была получена посредством метода электронной микроскопии (в частности, методом КриоЭМ) и РСА (указано в статье[2]) при разрешении 7.40 Å. Один мономер (цепи А-V) представляет собой белок FlgG базального тела стержня в основании жгутика. Структура всего белка изображена на рис.4. В данном задании мы будем работать с цепью А. Она контактирует с тремя белковыми цепями: F, G, L (auth a - обозначение из cif-файла).
Нам необходимо найти контакты с этими тремя белковыми цепями, определить остатки в составе цепи А, отвечающие за них, и представленность эти контактов. Для этого воспользуемся colab-блокнотом (комментарии сохранены оригинальные, из практикума). В результате, мы получили девять контактов, восемь из которых являются проксимальными (выдача в качестве таблицы представлена в той же colab-сессии) и один полярный совместно с перекрыванием Ван-дер-Ваальсовых радиусов (между рабочей цепью А и цепями F, G, Q (auth f), B, L (auth a)). Больше всего контактов (по два) образуют следующие остатки в цепи А: Arg-153 и Gln-124, меньше всего (по одному): Lys-256, Pro-167, Ala-165, Thr-150.
Список литературы
- Kovaľ T, Østergaard LH, Lehmbeck J, Nørgaard A, Lipovová P, et al. (2016) Structural and Catalytic Properties of S1 Nuclease from Aspergillus oryzae Responsible for Substrate Recognition, Cleavage, Non–Specificity, and Inhibition. PLOS ONE 11(12): e0168832.
- Saijo-Hamano Y, Matsunami H, Namba K, Imada K. Architecture of the Bacterial Flagellar Distal Rod and Hook of Salmonella. Biomolecules. 2019; 9(7):260.