Практикум 8. Структурные домены. Вторичная структура
Задание 1
В этом задании работали со структурой с PDB-ID 7DGD, которая была получена методом электронной микроскопии. Она представляет собой метаботропный глутаматный рецептор 1 (mGluR)[1], сопряжённый с G-белком (GCPR, класс С), человека. При связывании с лигандом (глутаматом) происходит конормационная перестройка, вследствие чего запускается сигнальный каскад через G-белки, который активирует кальций-фосфатидилинозитольную систему вторичных посредников[2]. Это играет большую роль в центральной нервной системе.
Для данной структуры реализизуем алгоритм DOMAK для разбиения структуры на домены (работаем с цепью А), а контакты посчитаем с помощью алгоритма arpeggio (ссылка на страничку с кодом, результатом и картинкой).
Задание 2
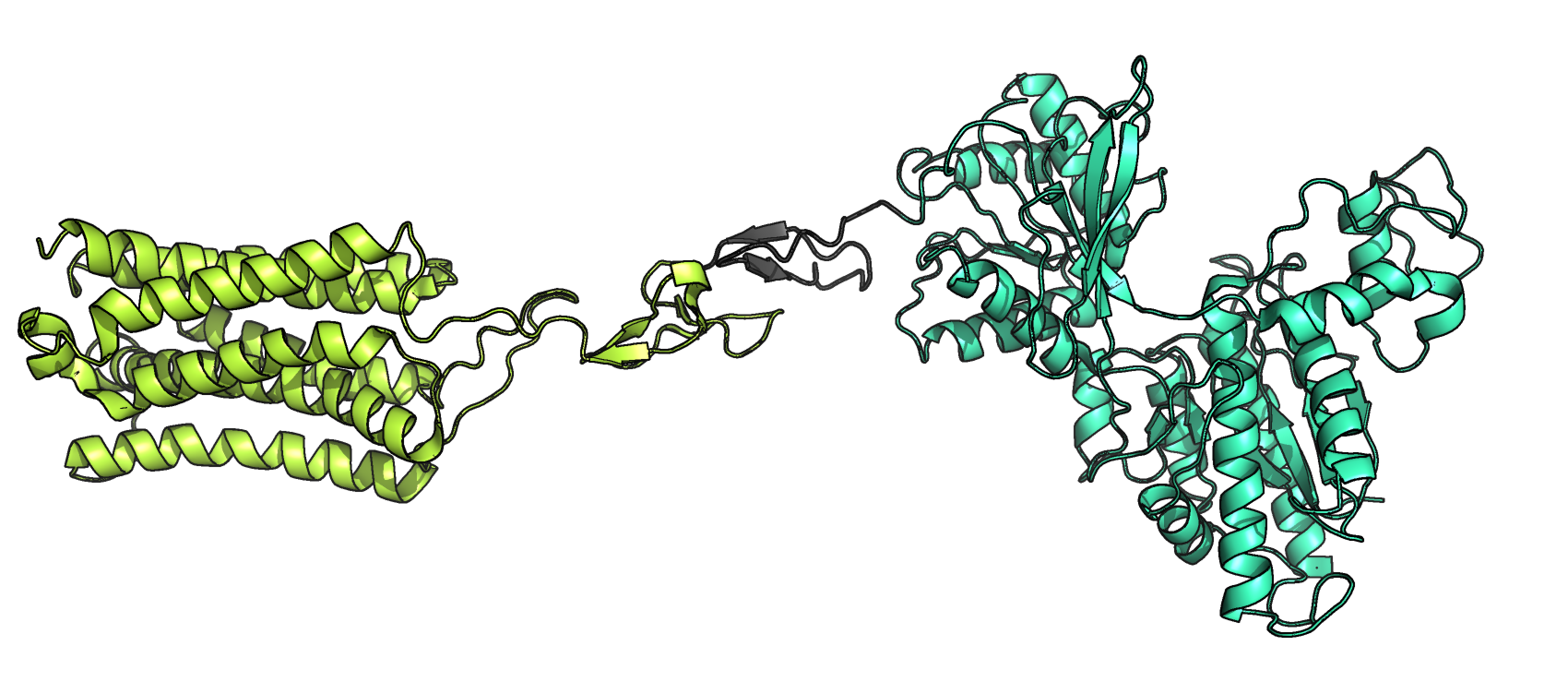
Далее на сайте PDB нашли информацию о структурных доменах из базы данных SCOP (рис.2):
- Type 1 solute binding protein-like (белок, связывающий растворитель, типа 1): 35-512.
- G protein-coupled receptor-like (рецептор, распознающий G белок): 581-839.
Видно, что позиции и количество доменов несколько различаются (31-522 - первый домен, 522-580 - второй, 580-863 - третий по предсказанию DOMAK). По данным из БД Pfam количество доменов равно трём, однако функции, по сравнению с данными из SCOP, и разметка (в отличие от DOMAK) сильно различаются (рис.3):
- Лиганд-связывающий домен: 77-487.
- Домен с девятью цистеинами: 519-571.
- Трансмембранный домен: 586-834.
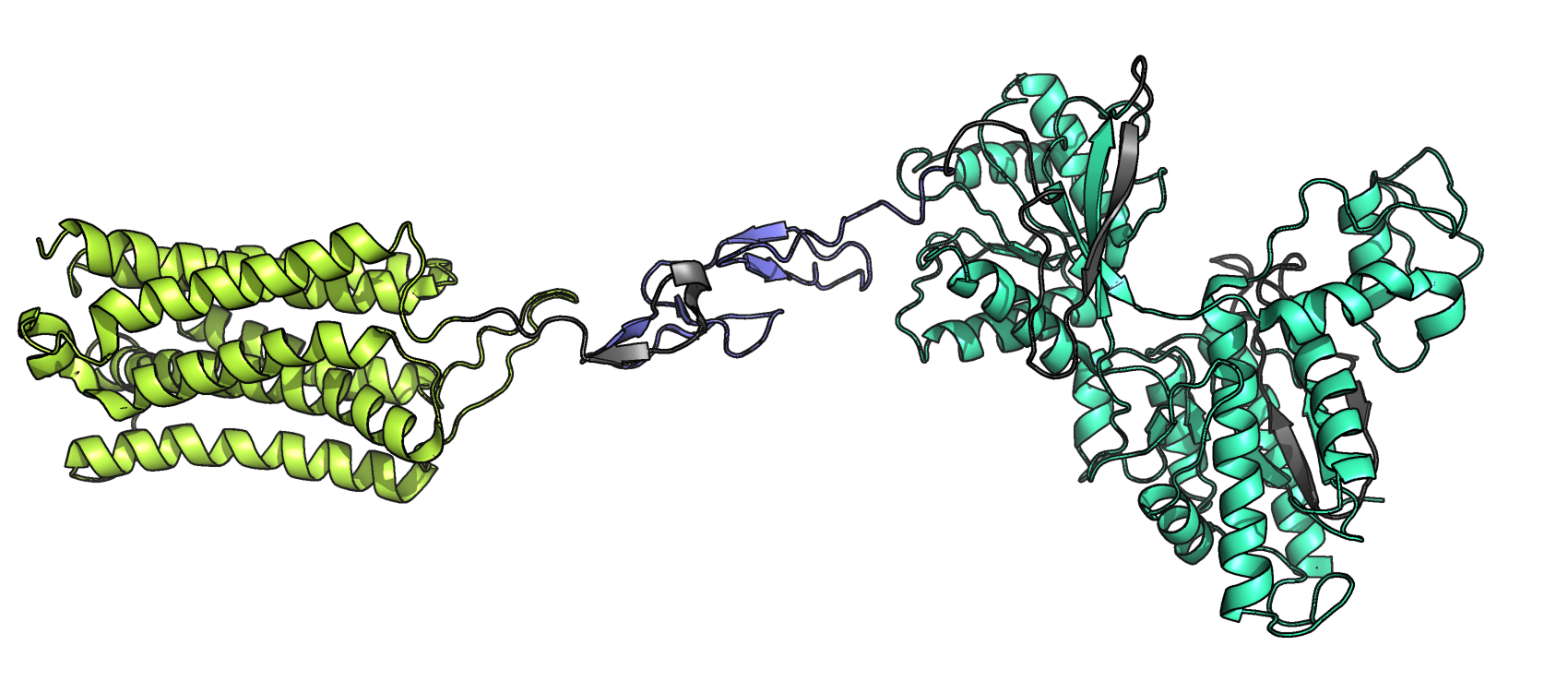
Итого, по разметке доменов SCOP чуть ближе к предсказанию по SplitValue, за исключением того, что один домен отсутствует по данным из SCOP. Однако предсказание по количеству доменов совпадает с данными Pfam. Если сравнить разметку с Pfam, то разметка второго домена близка к результатам SplitValue, первого - существенно отличается, третьего - отличие в С-конце домена, N-конец не на много отличается.
Задание 3
Далее найдём информацию об эволюционных доменах по базе InterPro (рис.4). По количеству доменов результаты совпадают и с Pfam, и с DOMAK, однако по разметке, по крайней мере, первых двух доменов совпадает с информацией из Pfam (координаты приведены выше), а не с выдачей SplitValue. Разметка третьего домена от InterPro (С-концевой домен - 586-854) отличается от Pfam и от БД PROSITE profiles (592-854), но более ближе к выдаче SplitValue, второго - отличие в C-конце, первого - существенные различия в обоих концах.
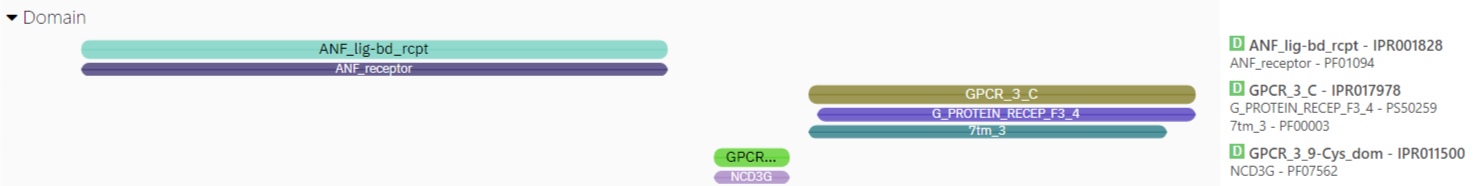
Согласно разметке InterPro (рис.4 и рис.5) эволюционные домены выполняют следующую функцию:
- Лиганд-связывающий домен: 77-487.
- Домен с девятью цистеинами (находится вне мембраны), участвующими в дисульфидных мостиках: 519-571.
- С-концевой домен: 586-834. По данным из InterPro он содержит 7 трансмембранных участков, которые собираются в карман, куда попадают цикламат (используется в качестве подсластителя) и лактизол (маскирует сладкий вкус, действует как ингибитор), связываются с белком; из-за этого появляется ощущение сладкого вкуса.
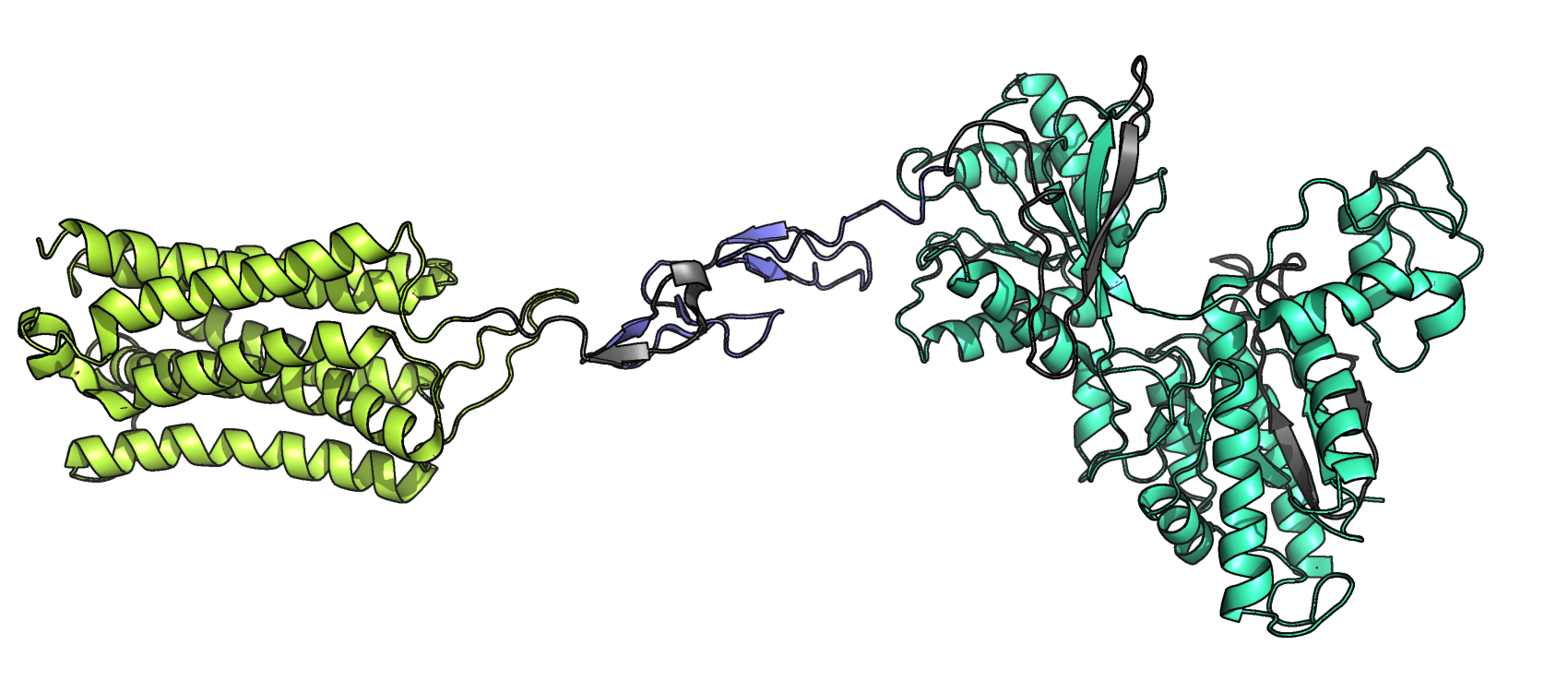
Согласно информации из Uniprot[2], у этого белка действительно есть трансмембранный домен, состоящий из семи участков в пределах предсказаний InterPro, но, что касается двух остальных доменов, то такой информации нет, а также есть информация о разметке доменов лишь для цепи В, но функции их неизвестны. По другим данным[3], данный трансмембранный домен может связываться с аллостерическим модулятором FITM, что приводит к регуляции связывания с глутаматом внеклеточного домена. Есть цистеин-богатый домен[3][4], необходимый для димеризации белка, между трансмембранным и N-концевым доменом (авторы статьи[4] обозначают четыре домена: N-, C-концевой (идёт после трансмембранного, внутриклеточный), трансмембранный и цистеин-богатый) и для стабилизации этих доменов. Внеклеточный N-концевой домен ответственнен за связывание с лигандом - L-глутаматом.
Задание 4
Сгенерируем аннотацию вторичной структуры с помощью 2Struc. В целом, результаты выдачи алгоритмов похожи (рис.6), за исключением мелких различий, однако есть и большие различия.

Рассмотрим три таких участка (ссылка на сессию):
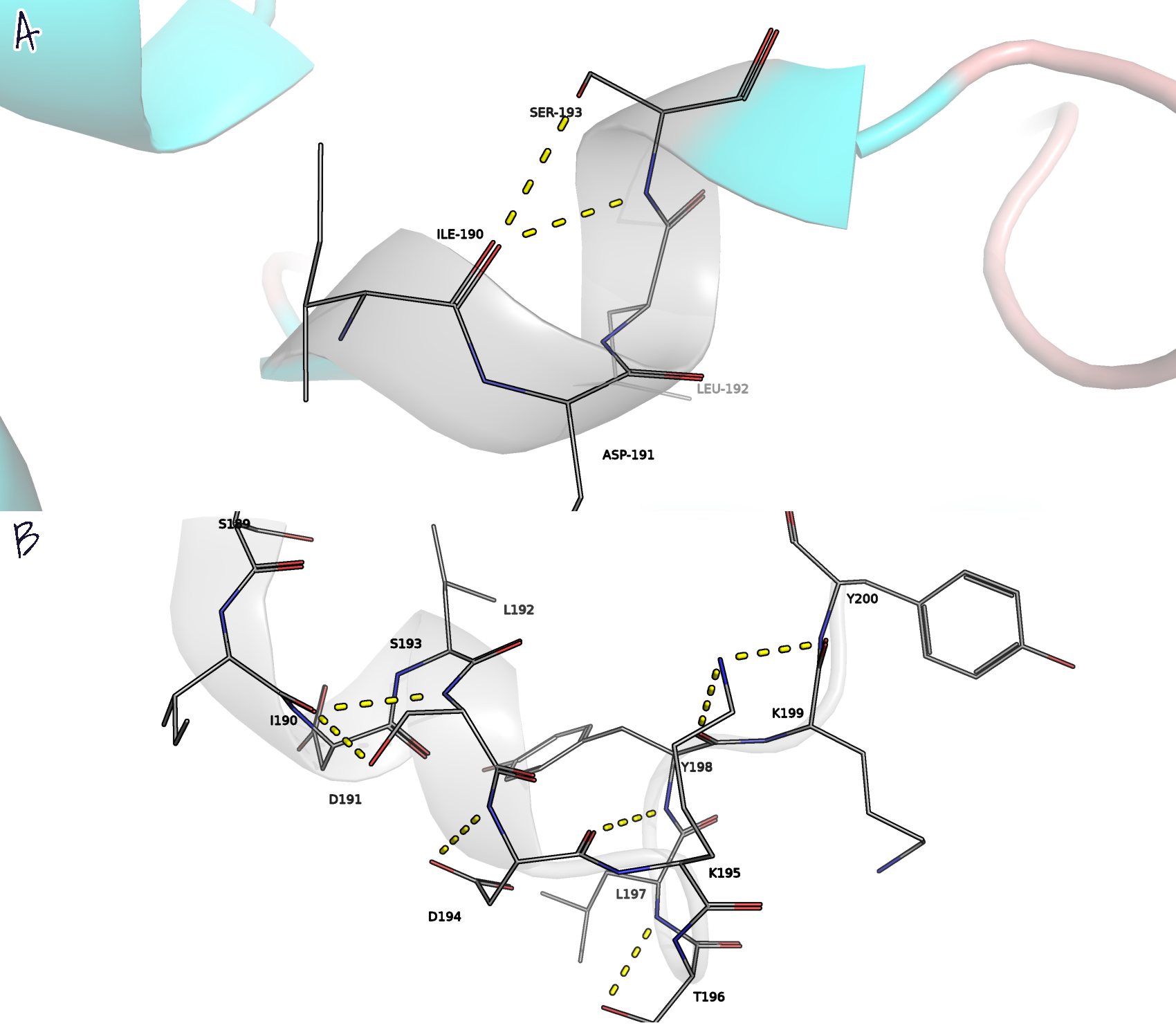
- Участок с координатами 189-200, который, по мнению DSSP, является 310-спиралью (190-193) с n-поворотом (195-197) (рис.7), STRIDE - только n-поворотом (189-200). PyMol тоже нарисовал в этом участке спираль (но на одну аминокислоту по краям больше, то есть с 189 по 194 остатки), причём, здесь всего лишь одна водородная связь между атомами остова 190 и 193 остатков (паттерн i-(i+3) как у 310-спирали); она короткая и поддерживается водородной связью тех же остатков, но кислородом остова и OH-группой бокового радикала серина. Если удлинить N- и C-концы спирали, то найдётся ещё одна водородная связь между азотом остова и кислородом в карбоксильной группе Asp-194.
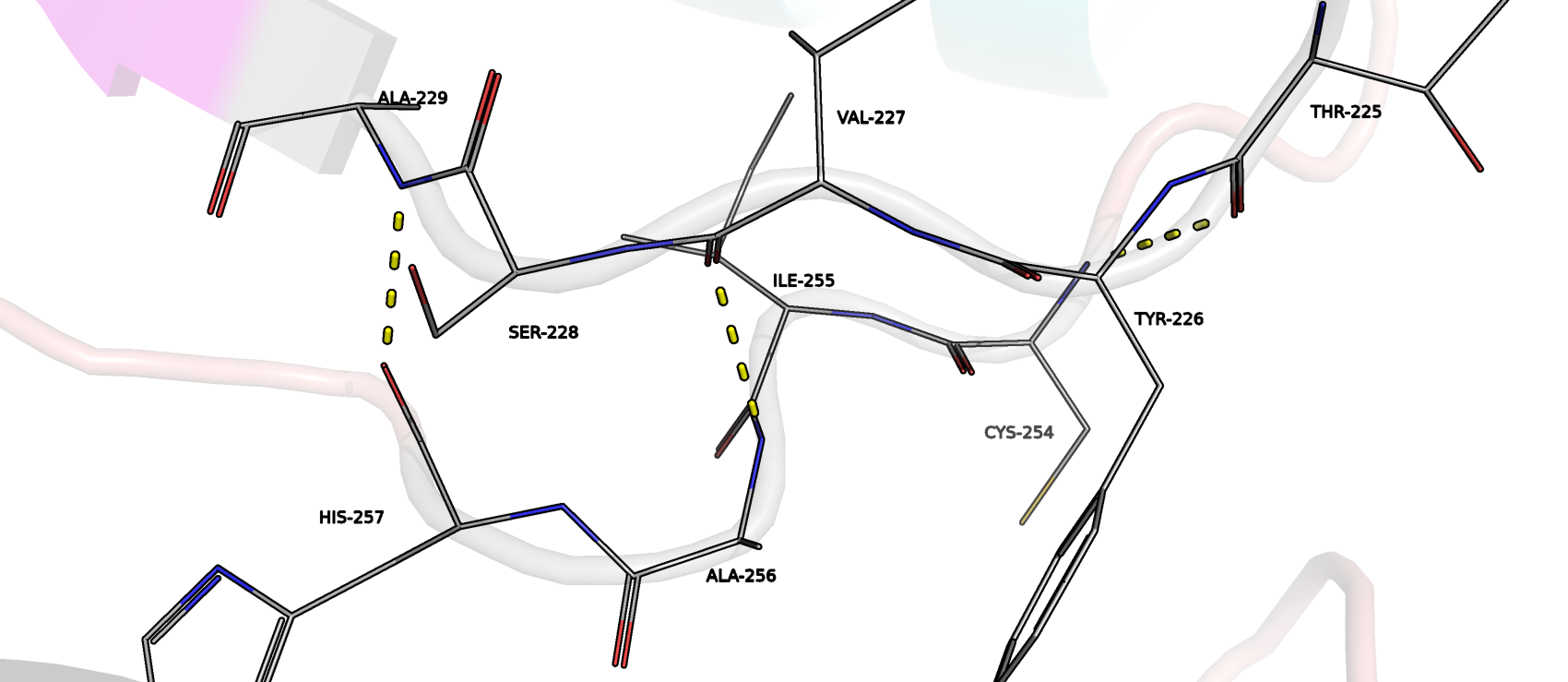
Водородные связи: 190-193, 194-194, 194-198, 196-197, 198-199, 199-200. За исключением 190-193 остатков, остальные связи строгому для всех паттерну не подчиняются; лишь только начиная со 196 остатка наблюдается паттерн i-(i+1). У алгоритма STRIDE все остатки в данном интервале обозначаются как "Т", то есть участки 3-, 4- или 5-поворотов, не составляющие спираль достаточной длины, однако даже они не подчиняются паттерну этих n-поворотов, поэтому он ошибся в аннотации данного участка. В случае DSSP: он правильно отметил 310-спираль, но с поворотом несколько промахнулся: 194-198 подходит под 4-поворот, но 194 остаток не входит в границы n-поворота. PyMol его отнёс к 310-спирали (точнее, авторы структуры). - Участок с координатами 254-257, который, по мнению STRIDE, является β-листом; по мнению DSSP, изломом (256) и остатком в изолированном β-мостике (257) (рис.8). PyMol отрисовывает его как обычное выпетливание, хотя есть намёк на присутствие β-листа (связи между остатками 229-257, 227-256, 225-254). Ala-229 уже находится в составе β-листа (в самом его начале), но водородные связи в нём не образует, лишь только с His-257. Однако, судя по визуализации структуры, укладка β-листа нарушена из-за конформации атомов остатков, поэтому STRIDE ошибся. DSSP не совсем верно определил β-мостик (257), поскольку наблюдается лишь одна водородная связь между 229 и 257 остатками, но с изломом в 256 остатке прав.
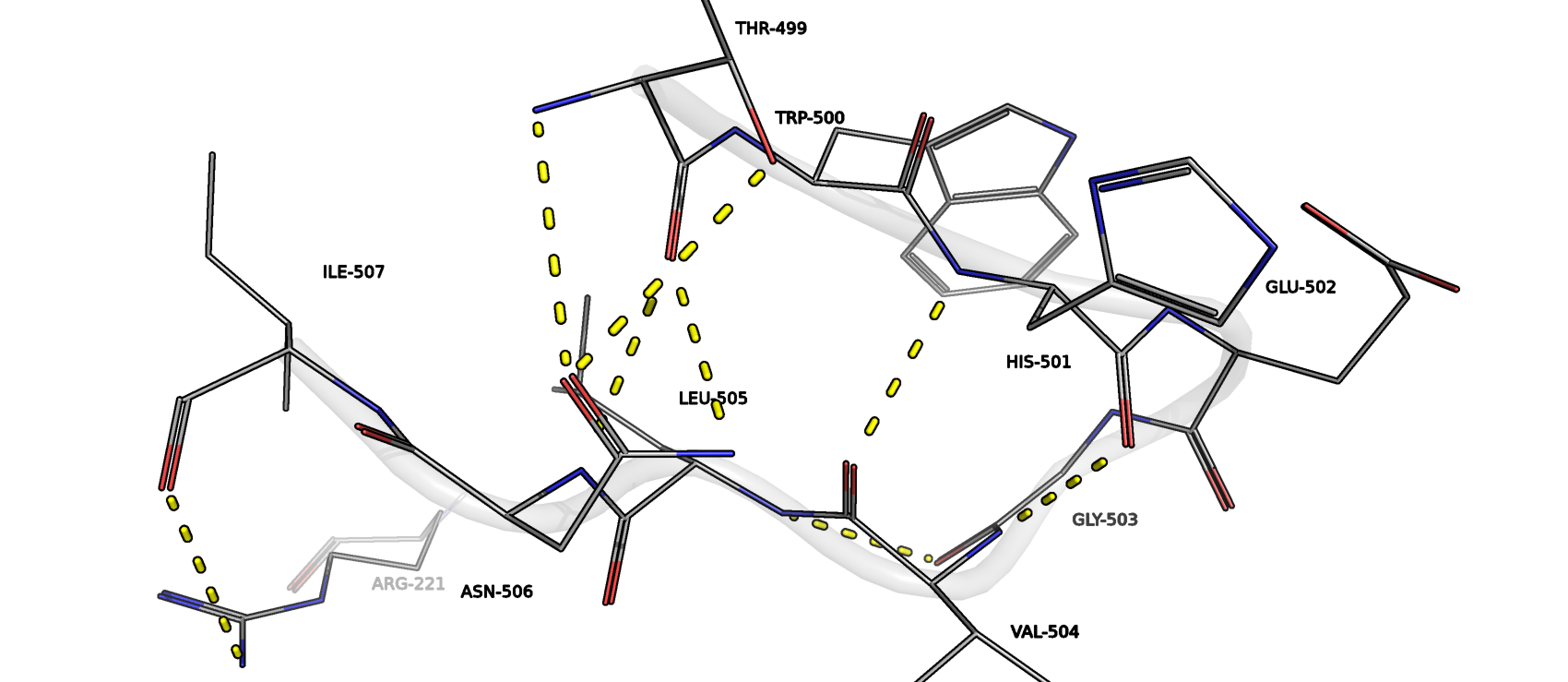
- Участок с координатами 504-507, который, по мнению STRIDE, является β-листом (рис.9). Водородные связи между следующими остатками: 499-506, 501-504, 503-505, 507-221. По идее, STRIDE не так уж и не прав: есть водородные связи между остовными атомами 499-506, 501-504, но они не могут восприниматься как β-листы, потому что есть водородные связи между атомами остова и боковых радикалов, между боковыми радикалами и связь между атомами остова 504 и 505 остатков. Скорее всего, 499-506 и 501-504 лучше отнести к β-мостикам, нежели чем к β-листам, но с натяжкой из-за дополнительных связей. DSSP отнёс участок к другим структурам, PyMol отрисовал его как обчную петлю, что даже более правдоподобно.
STRIDE основан на использовании статистики экспертных аннотаций, то есть на основе уже имеющейся базы данных со вторичными структурами, которых довольно мало, следовательно, алгоритм больше нацелен на подгон к известной вторичной структуре и не распознаёт нетипичные структуры. DSSP в этом плане более разнообразен и точен.
Список литературы
- Zhang J, Qu L, Wu L, Tang X, Luo F, Xu W, Xu Y, Liu ZJ, Hua T. Structural insights into the activation initiation of full-length mGlu1. Protein Cell. 2021 Aug;12(8):662-667. doi: 10.1007/s13238-020-00808-5. Epub 2020 Dec 5. PMID: 33278019; PMCID: PMC8310541.
- Uniprot.
- Wu H, Wang C, Gregory KJ, Han GW, Cho HP, Xia Y, Niswender CM, Katritch V, Meiler J, Cherezov V, Conn PJ, Stevens RC. Structure of a class C GPCR metabotropic glutamate receptor 1 bound to an allosteric modulator. Science. 2014 Apr 4;344(6179):58-64. doi: 10.1126/science.1249489. Epub 2014 Mar 6. PMID: 24603153; PMCID: PMC3991565.
- Watson LM, Bamber E, Schnekenberg RP, Williams J, Bettencourt C, Lickiss J, Jayawant S, Fawcett K, Clokie S, Wallis Y, Clouston P, Sims D, Houlden H, Becker EBE, Németh AH. Dominant Mutations in GRM1 Cause Spinocerebellar Ataxia Type 44. Am J Hum Genet. 2017 Sep 7;101(3):451-458. doi: 10.1016/j.ajhg.2017.08.005. Erratum in: Am J Hum Genet. 2017 Oct 5;101(4):638. Erratum in: Am J Hum Genet. 2017 Nov 2;101(5):866. PMID: 28886343; PMCID: PMC5591020.