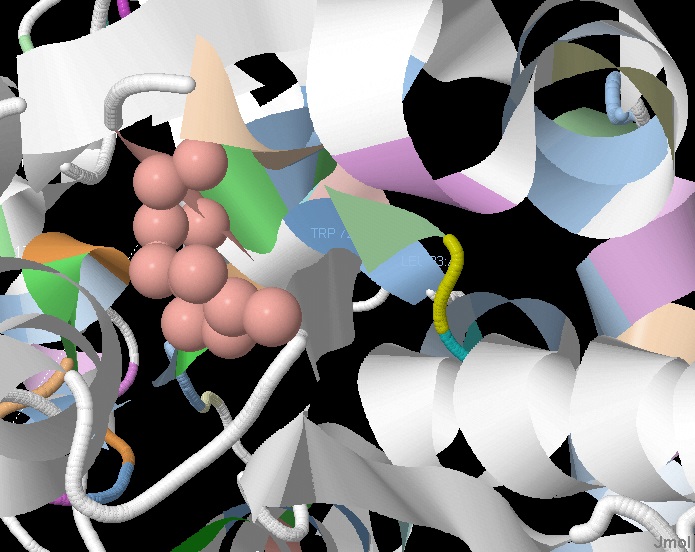
|
Поиск |
Алгоритм BLAST |
Название базы данных |
Ограничения по таксонам |
Порог e-value |
Максимальное количество хитов |
|
По прокариотам |
BLASTP |
Refseq |
Firmicutes(исключены) Eukaryota(исключены) |
6e-09 |
1835 |
|
По эукариотам |
BLASTP |
Refseq |
Eukaryota* |
6e-09 |
3 |
|
Домен |
Филум/царство |
Название организма |
Количество белков |
|
Archaea |
Euryarchaeotes |
Methanoculleus_bourgensis |
23 |
|
Methanoculleus_vannielii |
|||
|
Bacteria |
Proteobacteria |
Pseudomonas |
326 |
|
Desulfovibrio desulfuricons sub.sp. |
|||
|
Pseudomonas nitroreducens |
|||
|
Pseudomonas aeruginosa |
|||
|
Pseudomonas aeruginosa NGGM 2.81 |
|||
|
Pseudomonas viridiflava |
|||
|
Thioalkalivibrio |
|||
|
Thioalkalivibrio |
|||
|
Halomonas elongata DSM 2581 |
|||
|
Ralstonia solanacearum GMI 1000 |
|||
|
Cyanobacteria |
Prochlorococcus marinus str. |
6 |
|
|
Actinobacteria |
Streptomyces sp. |
76 |
|
|
Streptomyces sp |
|||
|
Streptomyces sp |
|||
|
CFB |
Bacteroides |
60 |
|
|
Bacteroides |
|||
|
Bacteroides fragilis |
|||
|
Parabacteroides distasonis |
|||
|
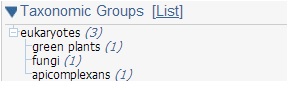
Eukaryotes |
Apicomplexans |
Plasmodium knowlesi strain H |
1 |
|
Green plants |
Cucumis sativus |
1 |
|
|
Fungi |
Moniliophthora perniciosa FA553 |
1 |