Доменная структура белка OPUAC_BACSU по данным Pfam

Cхема из Pfam: |
|||||
| Пояснения к схеме |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов |
Положение в последовательности белка OPUAC_BACSU | Клан |
| 1. | PF04069 | OpuAC | Семейство доменов названо по виду белка, свойство которого связывать глицин-бетаин, за счет характерных липидов. | 18-172 | Клан PBP (CL0177), содержит 23 семейства, у одного неизвестна функция (PFAM ID начинается с DUF): DUF3834, HisG, Lig_chan-Glu_bd, Lipoprotein_8, Lipoprotein_9, LysR_substrate, Mycoplasma_p37, NMT1 NMT1_2, OpuAC, PBP_like, PBP_like_2, Phosphonate-bd, SBP_bac_1, SBP_bac_11, SBP_bac_3, SBP_bac_5, SBP_bac_6, SBP_bac_7, SBP_bac_8, TctC, Transferrin, VitK2_biosynth |
Данные о домене OpuAC по данным Pfam

| Во сколько разных архитектур входит домен? | Данный домен входит в 15 архитектур. |
| Для какого числа белков, содержащих домен, известна последовательность? | Последоваетльность известна для 5220-и белков. |
| Для какого числа разных белков, содержащих домен, определена пространственная структура (домена или всего белка)? | Пространственная структура определена для 55 уникальных белков. |
| Выравнивание "seed" фрагментов белков, соответствующих домену | Доступно для скачивания в формате MSF |
| Эукариоты | Зеленые растения | 39 |
| Грибы | 0 | |
| Животные | 0 | |
| Остальные эукариоты | 12 | |
| Археи | 179 | |
| Бактерии | 4647 | |
| Вирусы | 0 | |
|
Таксон
|
Количество белков с доменом PF04069
|
|
| Эукариоты | Зеленые растения | 0 |
| Грибы | 0 | |
| Животные | 0 | |
| Остальные эукариоты | 8 | |
| Археи | 34 | |
| Бактерии | 2988 | |
| Вирусы | 0 | |
Сравнение описания мотивов в разных банках семейств, по данным InterPro
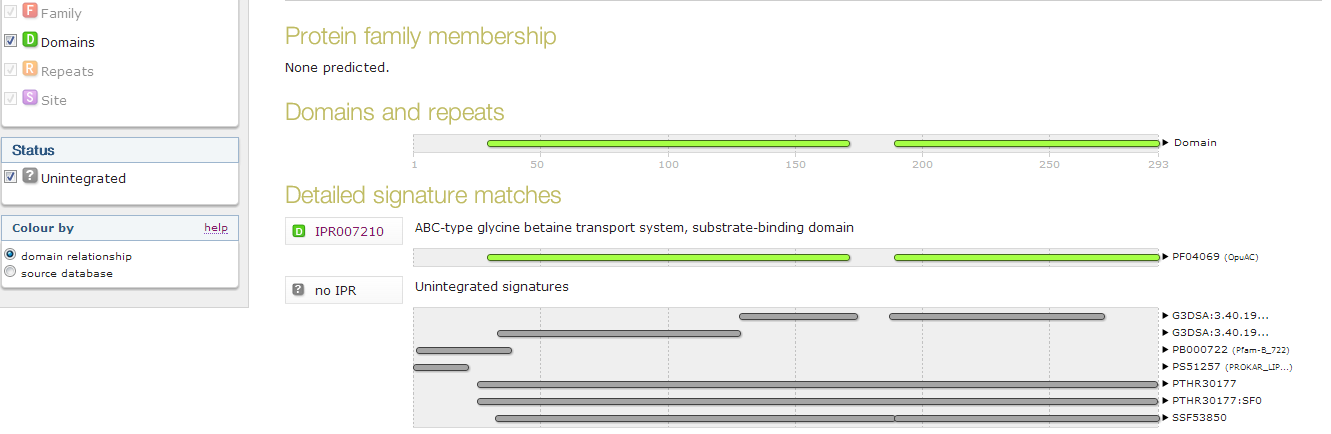
1) Самый короткий мотив – PS51257(PROKAR_LIPOPROTEIN) Он начинается 1-ым аминокислотным остатком и заканчивается 21-ым. Описан в банке PROSITE.
2) Самый длинный мотив – PTHR30177(GLYCINE BETAINE/L-PROLINE TRANSPORT SYSTEM PERMEASE PROTEIN PROW) Он начинается 26-ым аминокислотным остатком и заканчивается 292-ым. Описан в банке PANTHER (довольно новый банк, лично для меня).
3) Интегрирована известная структура белка. Границы структурных доменов не отличаются от границ доменов Pfam.