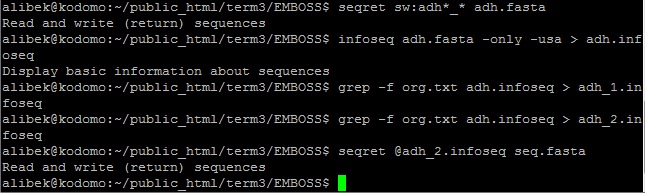
 Ссылка на файл, содержащий 5 открытых рамок считывания
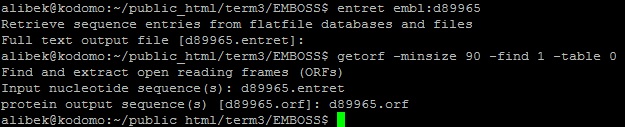
Сравнивая последовательности, легко заметить, что 3-ья открытая рамка считывания с координатами (163 - 432) полностью соответствует кодирующей последовательности с координатами (163 - 435), несмотря на разницу в длине на 3 нуклеотида.
Эта запись Embl ссылается на запись SwissProt P0A7B8
Ссылка на файл, содержащий 5 открытых рамок считывания
Сравнивая последовательности, легко заметить, что 3-ья открытая рамка считывания с координатами (163 - 432) полностью соответствует кодирующей последовательности с координатами (163 - 435), несмотря на разницу в длине на 3 нуклеотида.
Эта запись Embl ссылается на запись SwissProt P0A7B8FT /db_xref="UniProtKB/Swiss-Prot:P0A7B8"Вытащил эту запись: seqret sw:P0A7B8.fasta ссылка ниже: