Используя программу megablast, определил организм, которому принадлежит данный фрагмент. В качестве Database, я выбрал refseq_genomic:
✧ Организм - Nanoarchaeum equitans Kin4-M
✧ ACCESSION - NC_005213
✧ Координаты фрагмента - 1145-1444
✧ Не является кодирующим ("hypothetical protein")
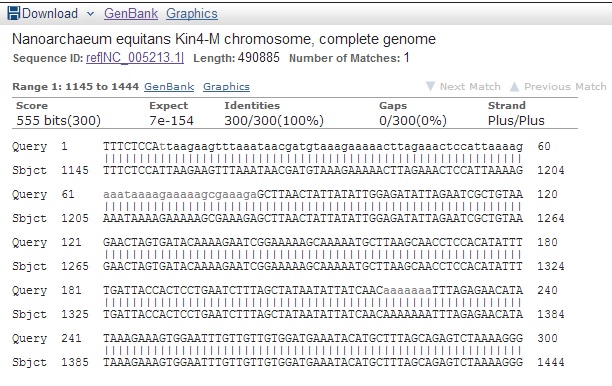
Рис.1. Данные полученные после работы с megablast.