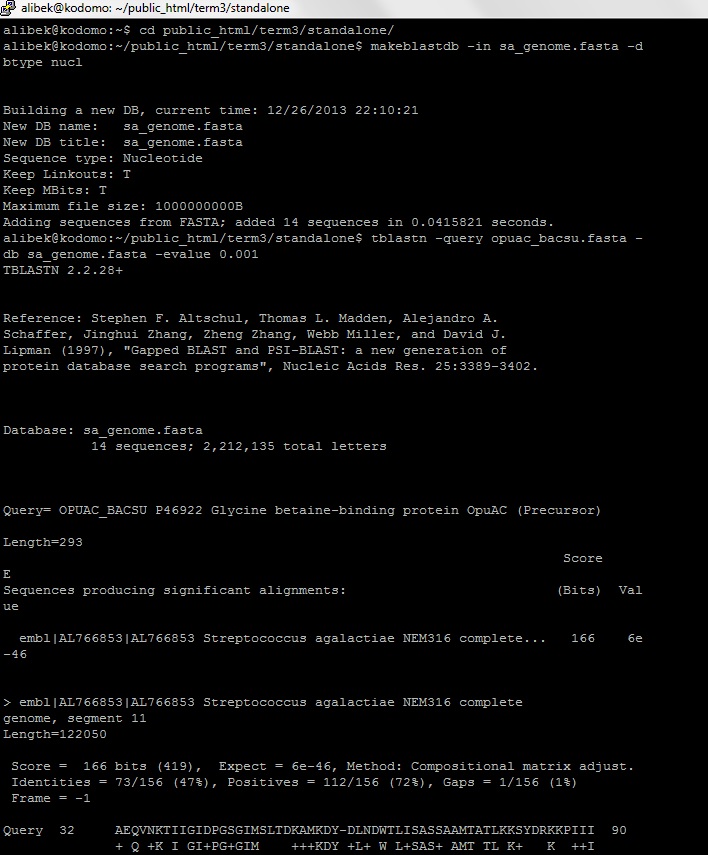
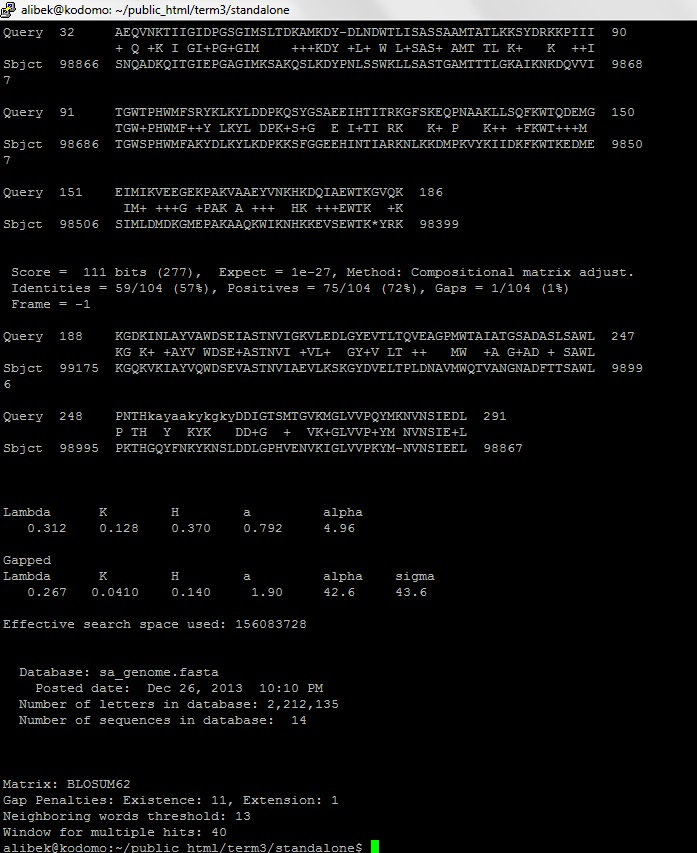
| Число находок с E-value < 0,001 | 2 |
| E-value лучшей находки | 6e-46 |
| Название последовательности с лучшей находкой | Streptococcus agalactiae NEM316 complete genome, segment 11 |
| Координаты лучшей находки (от-до) | 98866..98399 |
| Доля последовательности белка, вошедшая в выравнивание с лучшей находкой | 72% |
 Затем командой grep (а именно grep ">" trna_bacsu.fasta) была создана колонка названий входных последовательностей тРНК. Файл Exell я прикрепил ниже, с результатами задания №3 (для экономии время и места).
Затем командой grep (а именно grep ">" trna_bacsu.fasta) была создана колонка названий входных последовательностей тРНК. Файл Exell я прикрепил ниже, с результатами задания №3 (для экономии время и места). Далее было сделано выравнивание этой последователтности с различными тРНК Bacillus subtilis. Cледующей командой:
needle al766843.fasta trna_bacsu.fasta
В результате было получено много выравниваний, ниже представлены самые лучшие из них.
Далее было сделано выравнивание этой последователтности с различными тРНК Bacillus subtilis. Cледующей командой:
needle al766843.fasta trna_bacsu.fasta
В результате было получено много выравниваний, ниже представлены самые лучшие из них.
# Aligned_sequences: 2
# 1: AL766843
# 2: BSn5_t20930
# Matrix: EDNAFULL
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 77
# Identity: 72/77 (93.5%)
# Similarity: 72/77 (93.5%)
# Gaps: 3/77 ( 3.9%)
# Score: 352.0
#
#
#=======================================
AL766843 1 ggcggtgtagctcagctggctagagcgtccggttcatacccgggaggtcg 50
||||||||||||||||||||||||||||.|||||||||||||.|||||||
BSn5_t20930 1 ggcggtgtagctcagctggctagagcgtacggttcatacccgtgaggtcg 50
AL766843 51 ggggttcgatcccctccgccgcta--- 74
||||||||||||||||||||||||
BSn5_t20930 51 ggggttcgatcccctccgccgctacca 77
Aligned_sequences: 2
# 1: AL766843
# 2: BSn5_t20976
# Matrix: EDNAFULL
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 77
# Identity: 72/77 (93.5%)
# Similarity: 72/77 (93.5%)
# Gaps: 3/77 ( 3.9%)
# Score: 352.0
#
#
#=======================================
AL766843 1 ggcggtgtagctcagctggctagagcgtccggttcatacccgggaggtcg 50
||||||||||||||||||||||||||||.|||||||||||||.|||||||
BSn5_t20976 1 ggcggtgtagctcagctggctagagcgtacggttcatacccgtgaggtcg 50
AL766843 51 ggggttcgatcccctccgccgcta--- 74
||||||||||||||||||||||||
BSn5_t20976 51 ggggttcgatcccctccgccgctacca 77
#
# Aligned_sequences: 2
# 1: AL766843
# 2: BSn5_t20952
# Matrix: EDNAFULL
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 78
# Identity: 58/78 (74.4%)
# Similarity: 58/78 (74.4%)
# Gaps: 5/78 ( 6.4%)
# Score: 199.5
#
#
#=======================================
AL766843 1 -ggcggtgtagctcagctggctagagcgtccggttcatacccgggaggtc 49
||| .||||||||||||||.|||||||..||..|.|||..||.||||||
BSn5_t20952 1 gggc-ctgtagctcagctggttagagcgcacgcctgataagcgtgaggtc 49
AL766843 50 gggggttcga-tcccctccg--ccgcta 74
||.||||||| |||.|||.| ||.|.|
BSn5_t20952 50 ggtggttcgagtccactcaggcccacca 77
#=======================================
#
# Aligned_sequences: 2
# 1: AL766843
# 2: BSn5_t20904
# Matrix: EDNAFULL
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 78
# Identity: 56/78 (71.8%)
# Similarity: 56/78 (71.8%)
# Gaps: 5/78 ( 6.4%)
# Score: 181.5
#
#
#=======================================
AL766843 1 -ggcggtgtagctcagctggctagagcgtccggttcatacccgggaggtc 49
||| .||||||||||||||.|||||||..||..|.|||..||.||||||
BSn5_t20904 1 gggc-ctgtagctcagctggttagagcgcacgcctgataagcgtgaggtc 49
AL766843 50 gggggttcga-tcccctccg--ccgcta 74
|..||||||| |||..||.| ||.|.|
BSn5_t20904 50 gatggttcgagtccattcaggcccacca 77
Это выравнивания с BSn5_t20930 (из этого выравнивания была найдена анализируемая последовательность), BSn5_t20976 (tRNA-Met), BSn5_t20952 (tRNA-Met) и BSn5_t20904 (tRNA-Ser). Как видно, гены, кодирующие тРНК, очень похожи друг на друга. Здесь последоательности BSn5_t20930 и BSn5_t20976 совпадают, хотя утверждается, что они кодируют тРНК к разным аминокислотам. Возможно экспресиия одного из них в клетке подавлена. В данном случае была выбрана последовательность, выравнивание которой скорее всего оказалось хорошим в обоих случаях (разная длина слова), однако другие треониноые тРНК не нашлись при стандартной длине слова. При уменьшении длины слова сильно возрастает чувствительность алгоритма, а значит он принимает за подходящие не самые хорошие выравнивания. Так как гены тРНК похожи, при уменьшении длины слова резко возрастает количество находок.