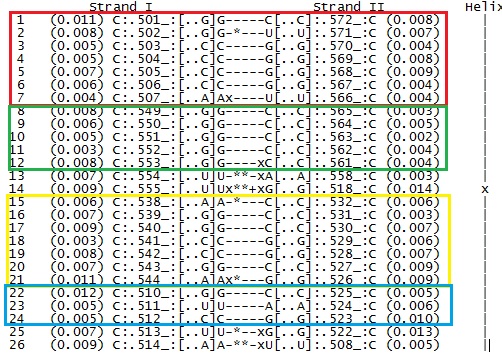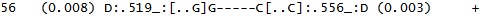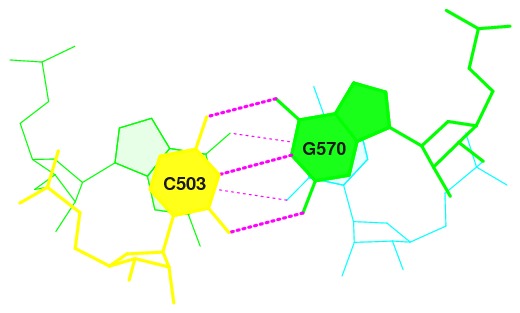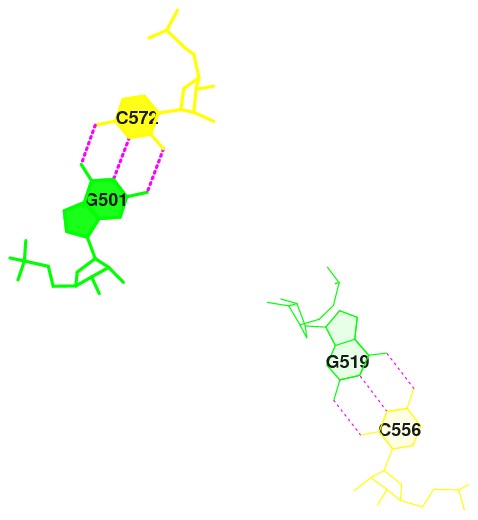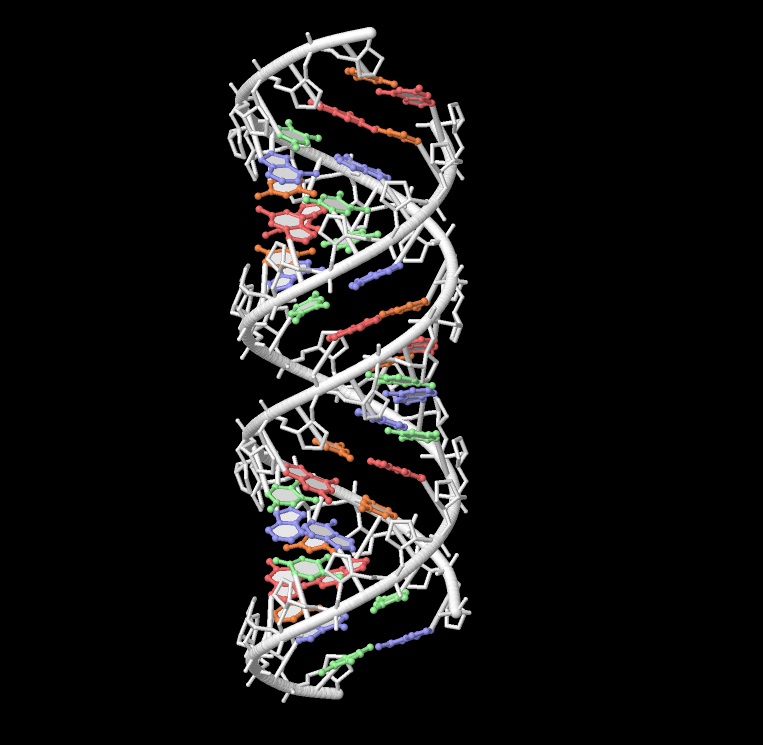



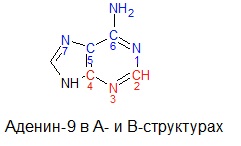
| А-форма | B-форма | Z-форма | |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
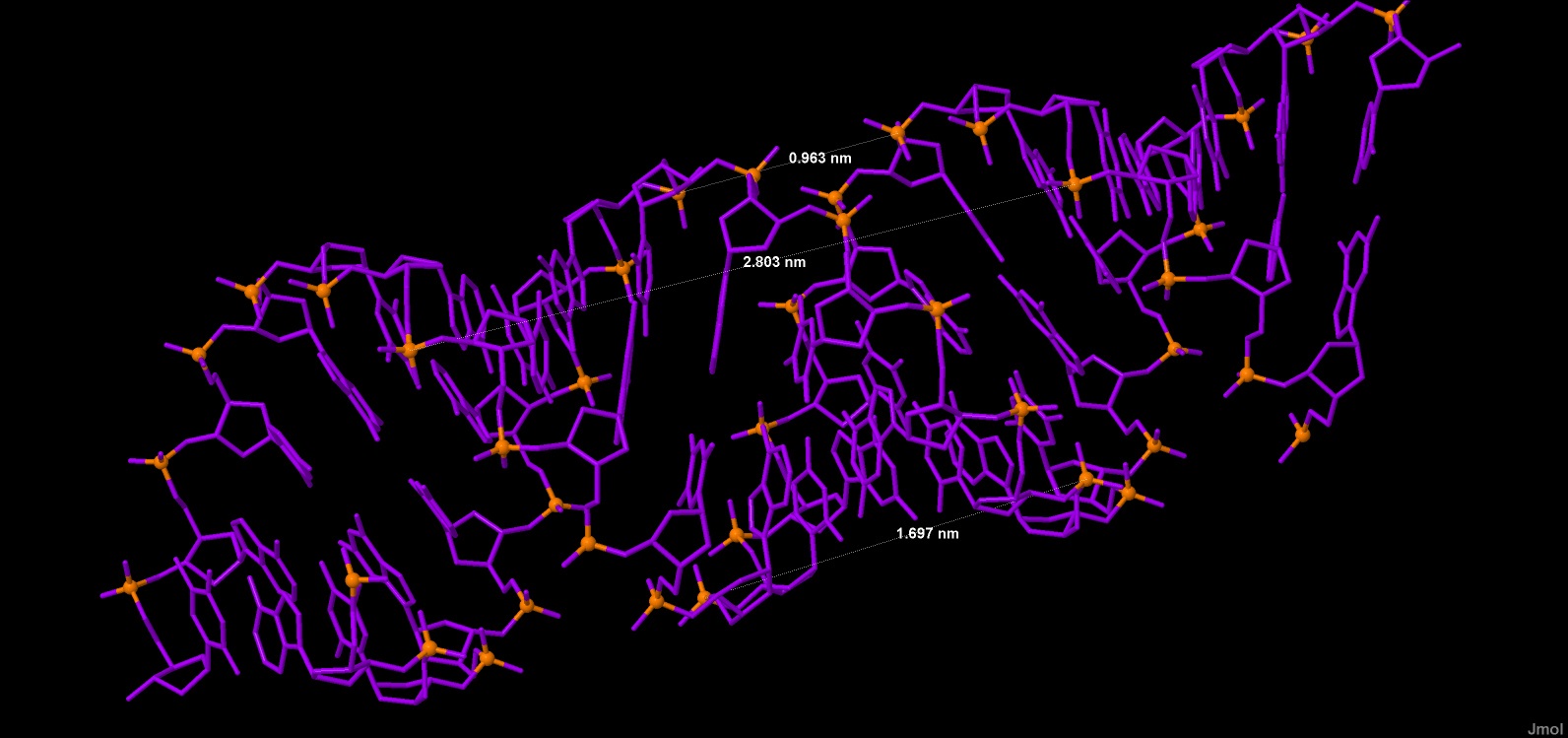
| Шаг спирали (A) | 28,03 [T]24:B.P #473 - [A]35:B.P #698 | 33,75 [G]25:B.P #493 - [A]35:B.P #698 | 43,5 [G]1:A.P #1 - [G]11:B.P #206 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (A) | 16,97 A.P #247 - [A]31:B.P #616 | 17,91 C]34:B.P #679 - [G]3:A.P #42 | 18.3 [G]1:A.P #1 - [C]6:A.P #105 |
| Ширина малой бороздки (A) | 9.63 C]26:B.P #515 - [C]8:A.P #146 | 13,20 [G]7:A.P #124 - [A]39:B.P #780 | 9,00 [C]2:A.P #23 - [G]7:A.O1P #125 |
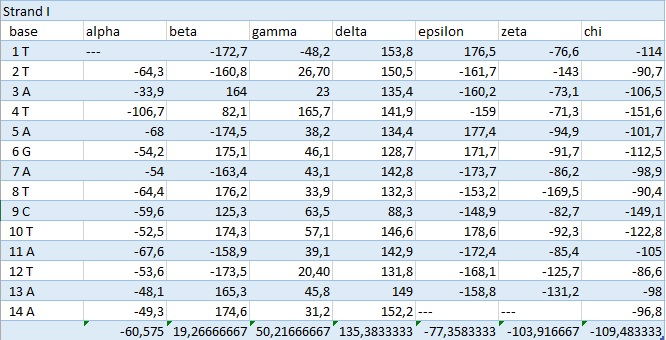
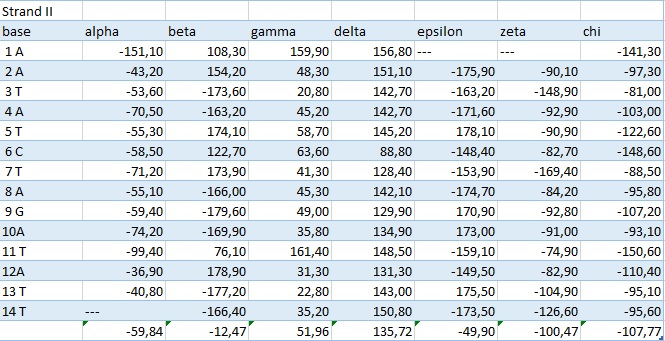
| α | β | γ | σ | ε | ζ | χ | |
| A-форма | -51,70 | 174,80 | 41,67 | 79,14 | -147,78 | -75,12 | -157,23 |
| А-форма из презентации | 62 | 173 | 52 | 88,3 | -178 | -50 | 160 |
| B-форма | -29,87 | 136,38 | 31,10 | 143,42 | -140,77 | -160,52 | -97,99 |
| B-форма из презентации | 63 | 171 | 54 | 123 | 155 | -90 | -117 |