| PDB код | Тип
(спираль, баррель) |
Какая мембрана (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 1I78 | Beta-barrel | Bact. Gram-neg outer, Escherichia coli | 26.5 ± 1.6 | 9.4 |
| 4FUV | Beta-barrel | Bact. Gram-neg outer, Acinetobacter baumannii | 25.0 ± 2.3 | 6.5 |
| 1XKW | Beta-barrel | Bact. Gram-neg outer, Pseudomonas aeruginosa | 24.8 ± 1.0 | 8.3 |
| 1YQ3 | Alpha-helical | Mitochon. inner, Gallus gallus | 29.6 ± 1.8 | 22.8 |
| 3RVY | Alpha-helical | Bact. Gram-neg inner, Arcobacter butzleri | 29.6 ± 0.4 | 21 |
| 2MGY | Alpha-helical | Mitochon. outer, Mus musculus | 28.6 ± 1.6 | 19 |
| PDB ID | Организм | Тип мембраны | TC-код | Угол наклона спиралей к нормали | Количество трансмембранных спиралей |
| 2JAF | Halobacterium salinarum | Плазматическая мембрана | 3.E.1.2.1* | 0 ± 1° | 7 |
Cl¯ (out) + hν -> Cl¯ (in)3.* - первичные транспортеры
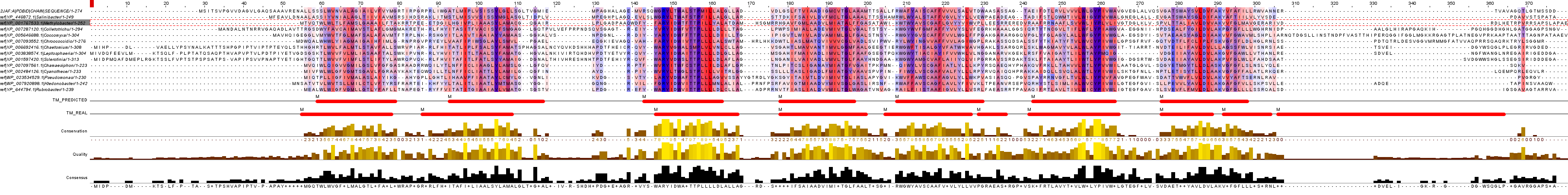 Были добавлены 2 аннотации:
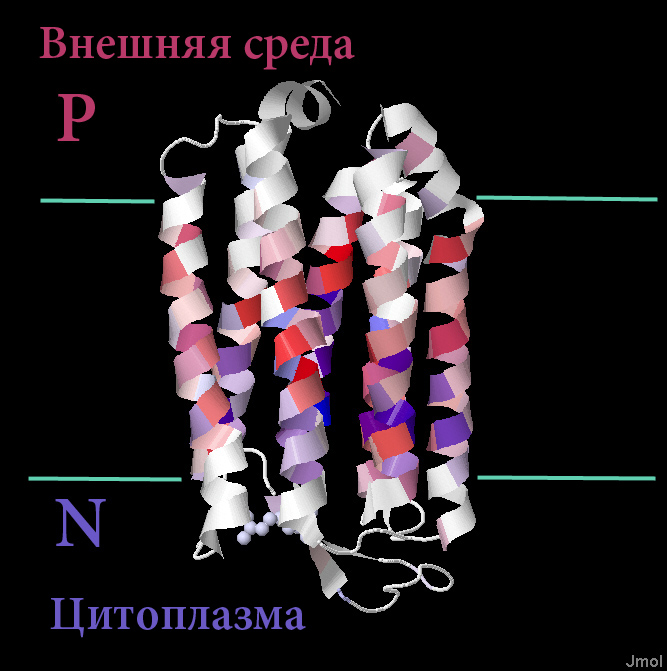
1)"TM_REAL" - показывает α-cпирали моего белка 2JAF, ниже представлено изображение структуры, откуда брались координаты α-спиралей.
Были добавлены 2 аннотации:
1)"TM_REAL" - показывает α-cпирали моего белка 2JAF, ниже представлено изображение структуры, откуда брались координаты α-спиралей.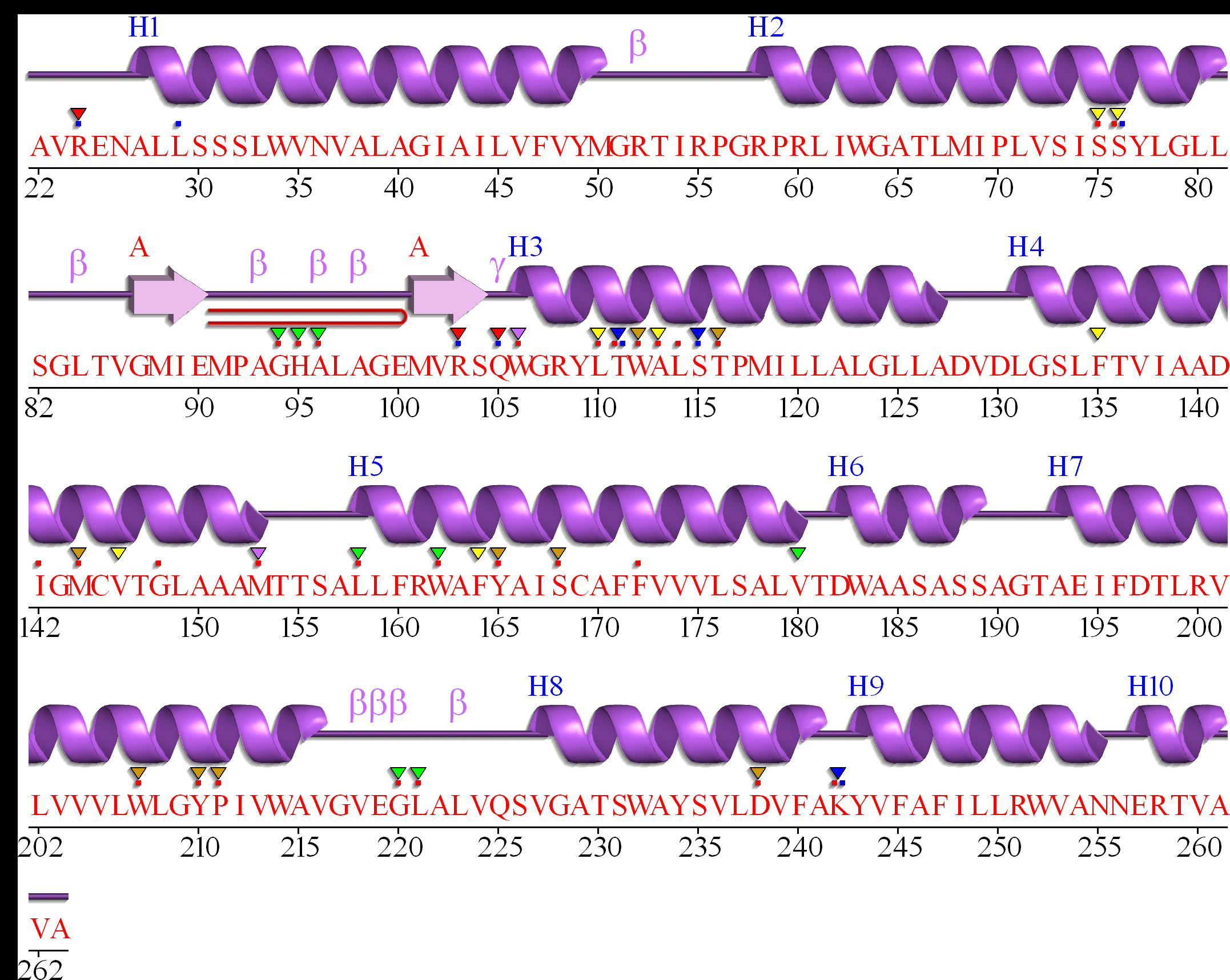 2) "TM_PREDICTED", выданная программой TMHMM для гомолога, а именно для YP_001767533.1, структура которого нeизвестна. Ниже изображение.
2) "TM_PREDICTED", выданная программой TMHMM для гомолога, а именно для YP_001767533.1, структура которого нeизвестна. Ниже изображение. 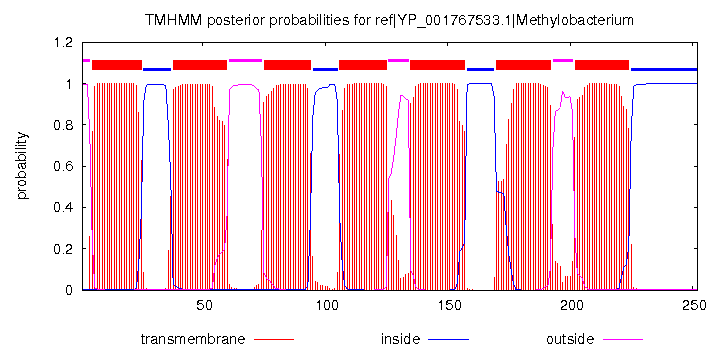
 Ответы на вопросы. Ниже снова представил изображение с выравниванием, на этот раз пронумеровав спирали, для удобства.
Ответы на вопросы. Ниже снова представил изображение с выравниванием, на этот раз пронумеровав спирали, для удобства.
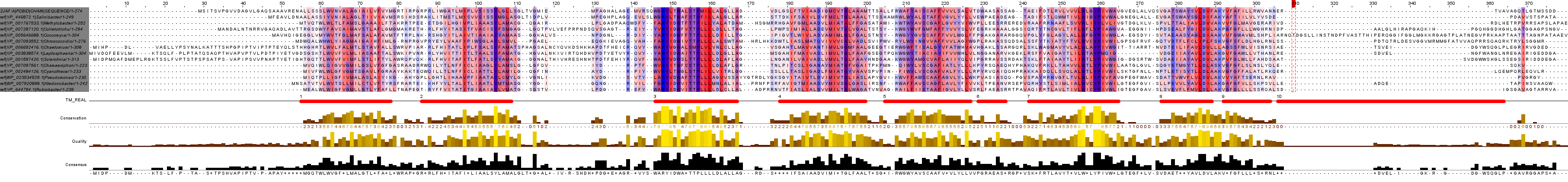 •1) Как видно из выравнивания, участки с трансмембранными спиралями образуют консервативные блоки. Наиболее часто встречаются следующие аминокислотные остатки: Лейцин,Изолейцин, Тирозин, Пролин, Аспартат, Треонин, Аланин/Глицин, Метионин
•1) Как видно из выравнивания, участки с трансмембранными спиралями образуют консервативные блоки. Наиболее часто встречаются следующие аминокислотные остатки: Лейцин,Изолейцин, Тирозин, Пролин, Аспартат, Треонин, Аланин/Глицин, Метионин