| Тип | Класс | Порядок | Семейство | Род | Вид |
| Firmicutes | Clostridia | Clostridiales | Clostridiaceae | Clostridium | Clostridium botulinum |
| Firmicutes | Clostridia | Clostridiales | Clostridiaceae | Clostridium | Clostridium tetani |
| Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae | Lactobacillus | Lactobacillus acidophilus |
| Firmicutes | Bacilli | Lactobacillales | Streptococcaceae | Streptococcus | Streptococcus pneumoniae |
| Firmicutes | Bacilli | Lactobacillales | Streptococcaceae | Streptococcus | Streptococcus pyogenes |
| Firmicutes | Bacilli | Bacillales | Bacillaceae | Bacillus | Bacillus anthracis |
| Firmicutes | Bacilli | Bacillales | Bacillaceae | Bacillus | Bacillus subtilis |



 •Филогенетическое дерево построенное алгоритмом Average distance using blosum62. Аналогично рассуждению выше.
•Филогенетическое дерево построенное алгоритмом Average distance using blosum62. Аналогично рассуждению выше.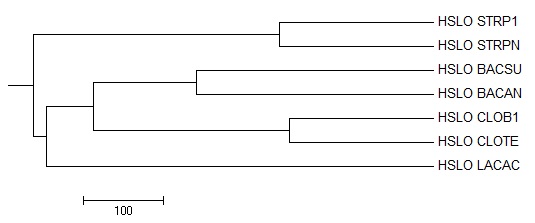 •Филогенетическое дерево построенное алгоритмом Neighbour joining tree using % identity.
•Филогенетическое дерево построенное алгоритмом Neighbour joining tree using % identity. 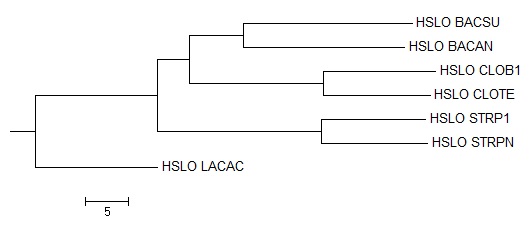 •Филогенетическое дерево построенное алгоритмом Neighbour joining tree using blosum62. Аналогично рассуждению выше.
•Филогенетическое дерево построенное алгоритмом Neighbour joining tree using blosum62. Аналогично рассуждению выше.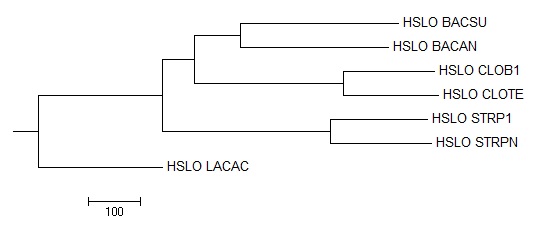
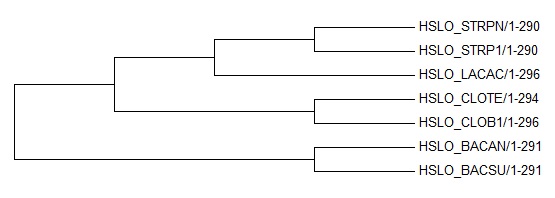 Укоренил дерево в наиболее правильную, с моей точки зрения, ветвь. Отделив, таким образом, класс Clostridia от класса Bacilli.
Укоренил дерево в наиболее правильную, с моей точки зрения, ветвь. Отделив, таким образом, класс Clostridia от класса Bacilli.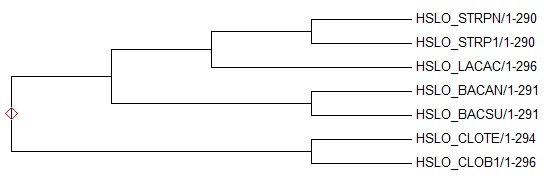 • Вывод: после укоренения, дерево, построенное программой MEGA полностью совпадает в правильным.
• Вывод: после укоренения, дерево, построенное программой MEGA полностью совпадает в правильным.