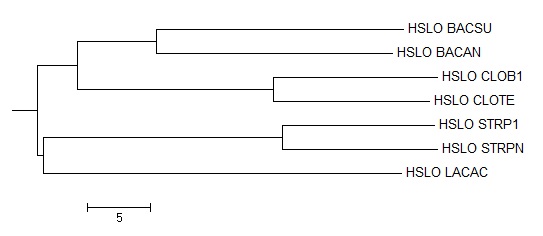
 C данным укоренением я не согласен, и считаю его неверным. И, хотя дерево топологически правильное и не появились новые ветви, укоренение произошло на уровне порядков, а не классов, видно что порядок Lactobacillales отделился.
C данным укоренением я не согласен, и считаю его неверным. И, хотя дерево топологически правильное и не появились новые ветви, укоренение произошло на уровне порядков, а не классов, видно что порядок Lactobacillales отделился. 
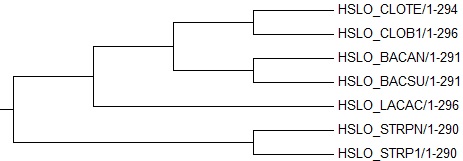 Как можно заметить, полученное дерево отличается от правильного, все листья одинаково отдалены от корня, посколько метод максимальной экономии не может подсчитать расстояние между листьями и корнем. Однако топологически дерево является правильным, поскольку новых ветвей не появилось. Наблюдения: представители семейства Streptococcaceae отделились от остальных бактерий. С одной стороны дерево неправильное, но с другой, как было замечено еще в 4 задании 2 практикума, данное дерево соответствует дереву, полученному алгоритмом Average distance using % identity. Таким образом можно предположить, что эволюция семейства Streptococcaceae происходит все же быстрее, чем целого класса Clostridia.
Вывод, так сказать обобщающий практикумы 2 и 3: построение филогенетических деревьев по одному белку дает достаточно противоречивые данные. Такие же построения с использованием внешней группы методом максимальной экономии и программой RETRE дают топологически правильные деревья без появления новых, нетривиальных ветвей, однако укоренения в обоих случаях происходит на более низких таксономических рангах, чем хотелось бы.
Как можно заметить, полученное дерево отличается от правильного, все листья одинаково отдалены от корня, посколько метод максимальной экономии не может подсчитать расстояние между листьями и корнем. Однако топологически дерево является правильным, поскольку новых ветвей не появилось. Наблюдения: представители семейства Streptococcaceae отделились от остальных бактерий. С одной стороны дерево неправильное, но с другой, как было замечено еще в 4 задании 2 практикума, данное дерево соответствует дереву, полученному алгоритмом Average distance using % identity. Таким образом можно предположить, что эволюция семейства Streptococcaceae происходит все же быстрее, чем целого класса Clostridia.
Вывод, так сказать обобщающий практикумы 2 и 3: построение филогенетических деревьев по одному белку дает достаточно противоречивые данные. Такие же построения с использованием внешней группы методом максимальной экономии и программой RETRE дают топологически правильные деревья без появления новых, нетривиальных ветвей, однако укоренения в обоих случаях происходит на более низких таксономических рангах, чем хотелось бы.  "Bootstrap consensus tree", метод Neighbour Joining
"Bootstrap consensus tree", метод Neighbour Joining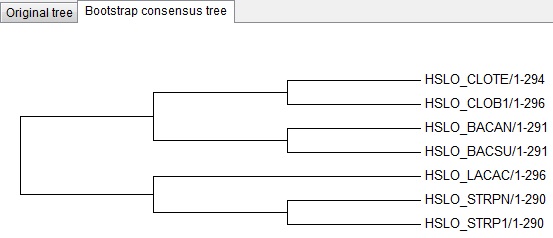 Далее укоренил дерево по классу Clostridia, получилось дерево, полностью совпадающее с правильным, изображено ниже.
Далее укоренил дерево по классу Clostridia, получилось дерево, полностью совпадающее с правильным, изображено ниже.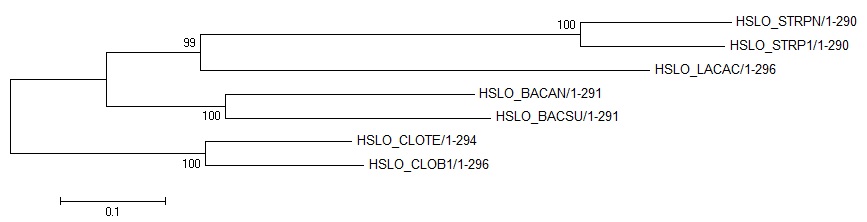 Микровывод: полученные деревья "Original tree" и "Bootstrap consensus tree" не отличаются. Также замечено, что в предыдущем практикуме метод "Neighbour Joining" дал не вовсем верные деревья, а построенные тем же методом, но с бутстрэп-анализом филогении деревья дают правильное построение. Как видно, подтвердился тот факт, что семейство Streptococcaceae эволюционирует быстрее.
Микровывод: полученные деревья "Original tree" и "Bootstrap consensus tree" не отличаются. Также замечено, что в предыдущем практикуме метод "Neighbour Joining" дал не вовсем верные деревья, а построенные тем же методом, но с бутстрэп-анализом филогении деревья дают правильное построение. Как видно, подтвердился тот факт, что семейство Streptococcaceae эволюционирует быстрее.