| Название бактерии | Мнемоника | AC с EMBL | Координаты | Цепь |
| Clostridium botulinum | CLOB1 | CP000726 | 9282..10783 | + |
| Clostridium tetani | CLOTE | AE015927 | 176113..177621 | + |
| Lactobacillus acidophilus | LACAC | CP000033 | 59255..60826 | + |
| Streptococcus pneumoniae | STRPN* -> STRP4 | AE005672* -> CP001015 | 15360..16817 | + |
| Streptococcus pyogenes | STRP1 | AE004092 | 23171..24505 | + |
| Bacillus anthracis | BACAN | AE016879 | 9335..10841 | + |
| Bacillus subtilis | BACSU | AL009126 | 90536..92089 | + |
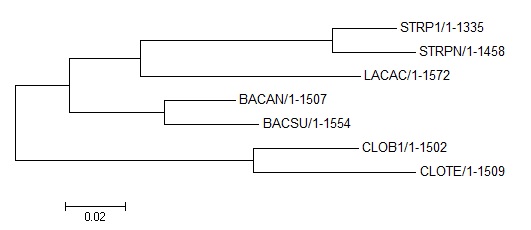 Наконец, после всех неудач с деревьями, построенными по белкам, получилось дерево максимально совпадающее с правильным. В нем класс Clostridia отделен от класса Bacilli, а класс Bacilli, в свою очередь, разделен на 2 порядка: Lactobacillales и Bacillales.
Наконец, после всех неудач с деревьями, построенными по белкам, получилось дерево максимально совпадающее с правильным. В нем класс Clostridia отделен от класса Bacilli, а класс Bacilli, в свою очередь, разделен на 2 порядка: Lactobacillales и Bacillales. 
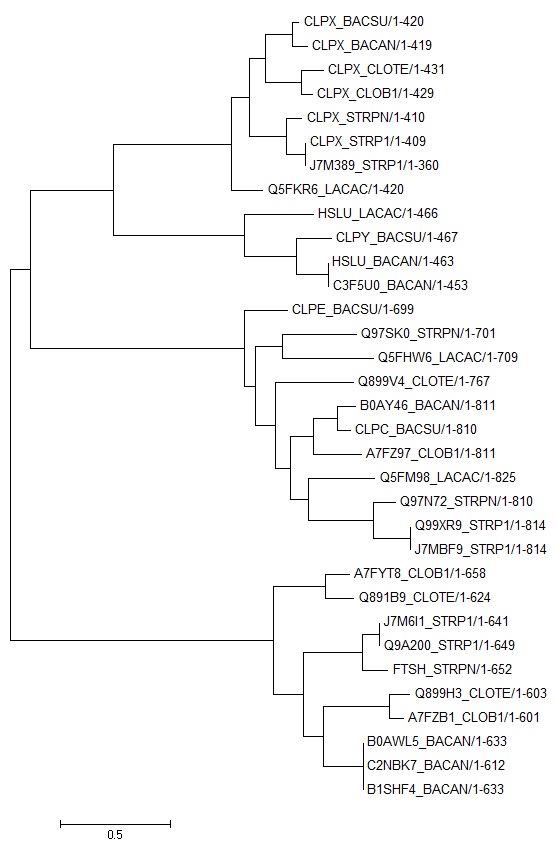 Обсуждение:
Обсуждение: