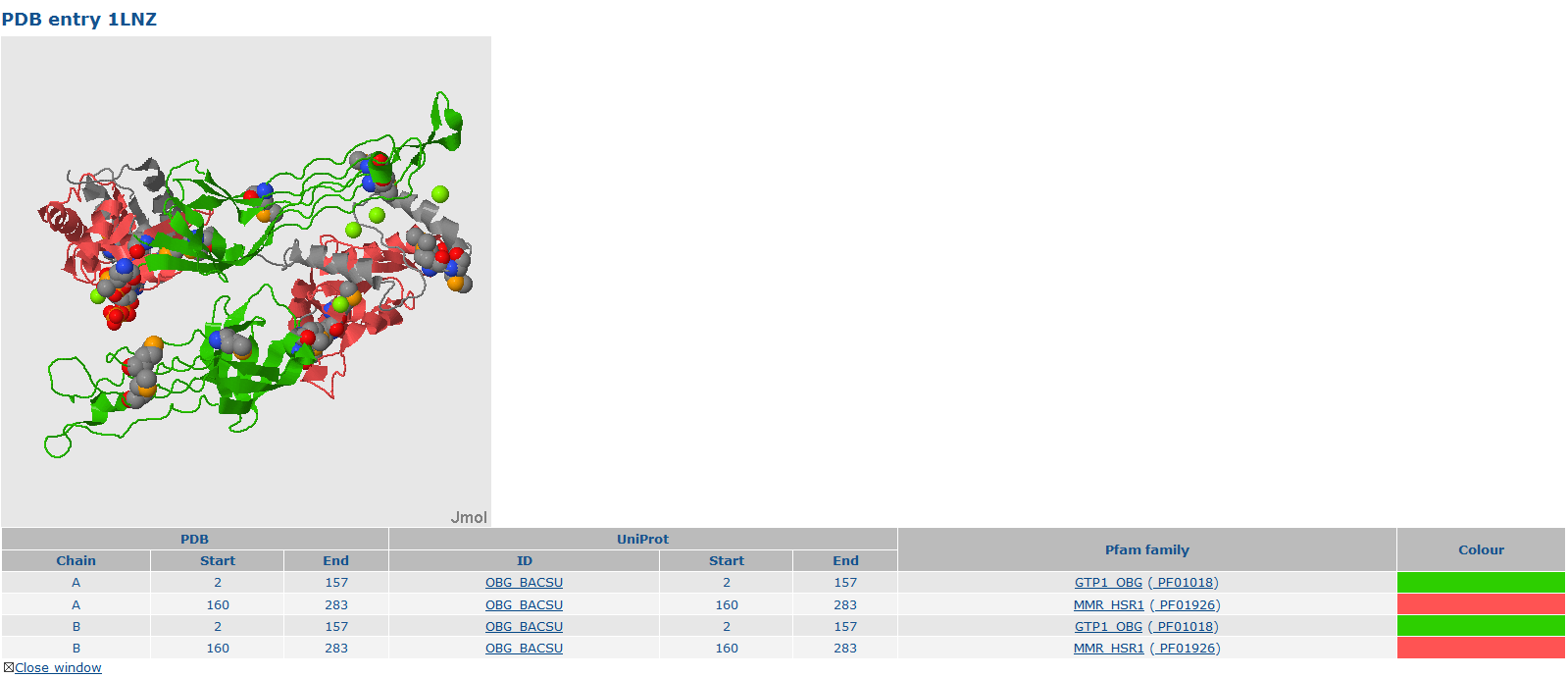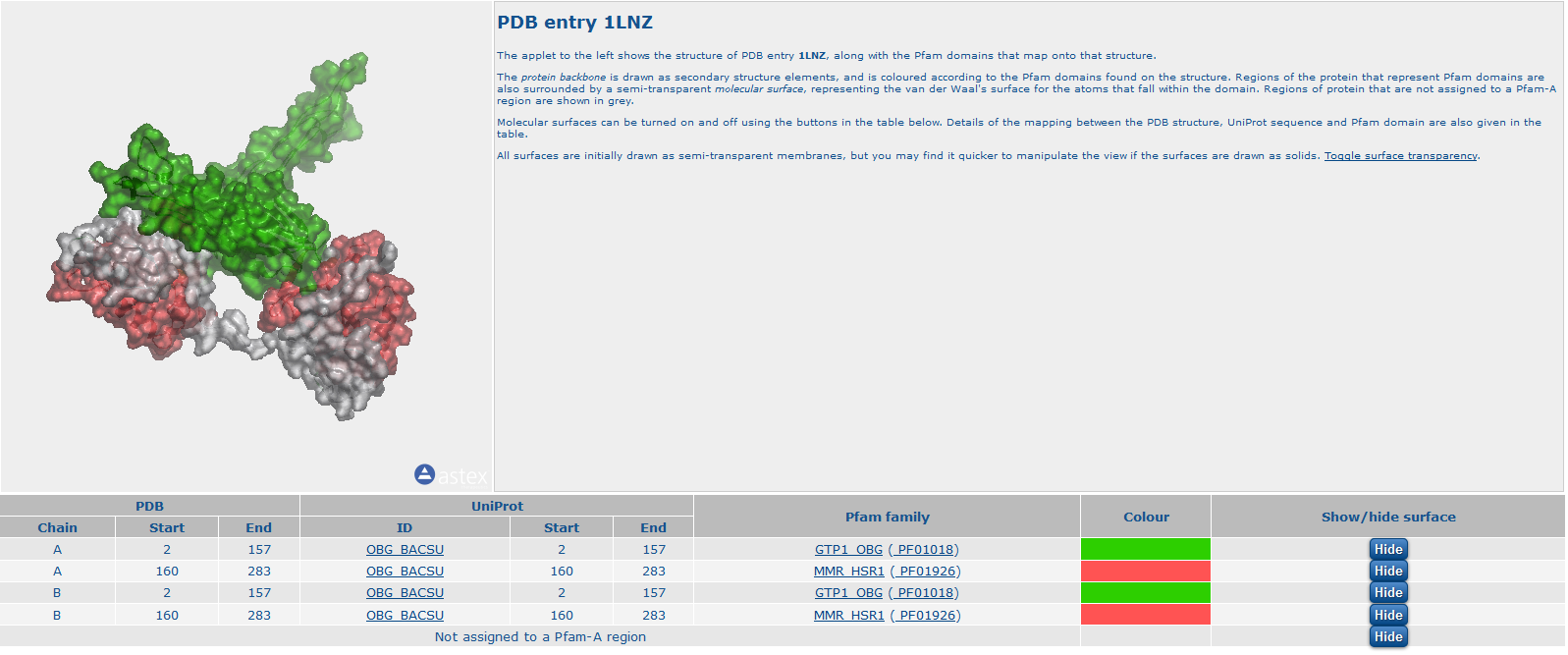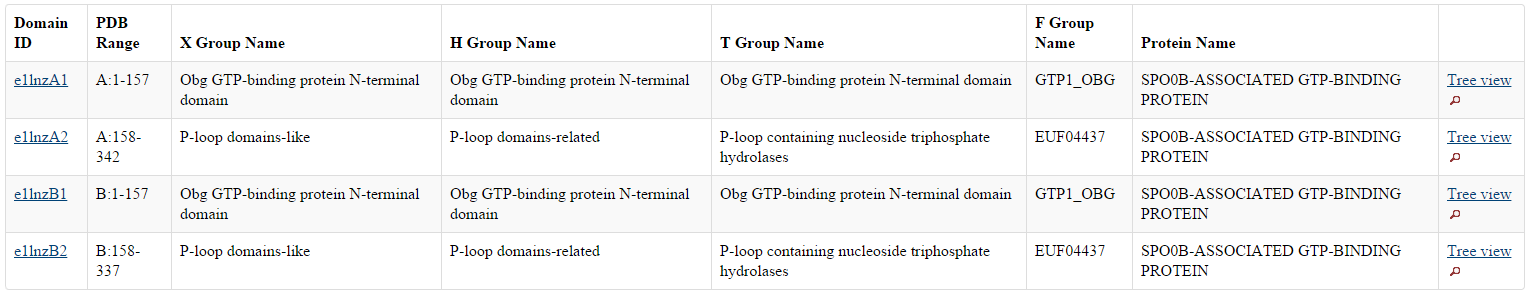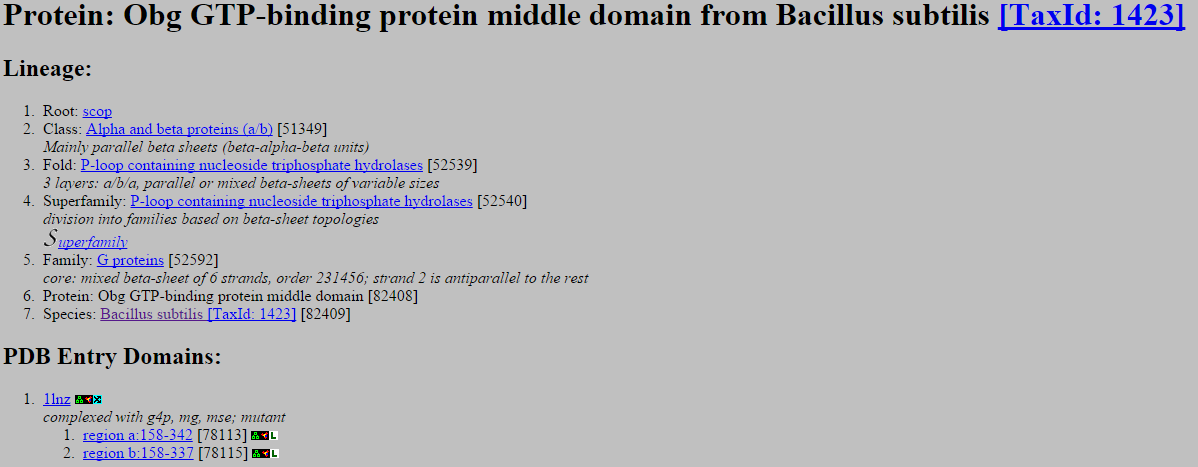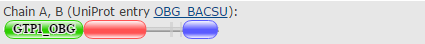
| Домен | ID домена (по Pfam) | Координаты | В какой цепи | Описание домена |
| GTP1_OBG | PF01018 | 2-157 | A, B | N-концевой домен, создающий OBG-фолд, сформированный тремя богатыми глицинами регионами, которые образуют коллаген-подобные левозакрученные спирали, соединенные водородными связями |
| MMR_HSR1 | PF01926 | 160-283 | A, B | 50S ribosome-binding GTPase |
| DUF1967 | PF09269 | 358-426 | A, B | Члены этого семейства содержат бэта-лист, состоящий из 4 тяжей, а также 3 альфа-спирали, фланкирующие этот лист. Этот домен обнаружен у GTP-связывающих белков, однако до сих пор остается неохарактеризованным функционально доменом |