Данный мини-обзор представляет собой поверхностное изучение генома и протеома бактерии Mucilaginibacter robiniae. В исследовании использовались данные белков и нуклеотидных последовательностей бактерии. В результате работы были выявлены особенности генома и протеома Mucilaginibacter robiniae.
Ключевые слова: бактерия, геном, протеом, длины белков, GC–состав, кодоны.
Род Mucilaginibacter, относящийся к семейству Sphingobacteriaceae в типе Bacteroidetes, был впервые описан Т. А. Панкратовым [1]. Около 50 видов Mucilaginibacter были выделены из различных сред, включая торфяные болота, водно-болотные угодья, разнообразные виды растений, гнилую древесину, пресную и сточную воду, различные типы почв [2]. Mucilaginibacter играют ключевую роль в расщеплении различных биополимеров, таких как целлюлозу, пектин, ксилан и несколько других полисахаридов [3]. В штаммах бактерий Mucilaginibacter были обнаружены белки, относящиеся к семейству GH9 белков, состоящее из эндо-β-1,4 глюканаз, которые гидролизуют полисахариды. Две каталитические аминокислоты были идентифицированы как остатки аспарата и глутамата [4].
Бактерия вида Mucilaginibacter robiniae принадлежит роду Mucilaginibacter, семейству Sphingobacteriaceae порядка Sphingobacteriales, классу Sphingobacteriia в типе Bacteroidota домена Bacteria [5].
Геном и протеом бактерии для анализа взяты из источника [A].
В электронной таблице “СDS from genome of Mucilaginibacter robiniae” [B], в листах “prot_len_hist” и “gc_hist” построены гистограммы длин белков и GC-состава соответственно. При построении гистограмм использовалась функция СЧЁТЕСЛИМН.
С помощью собственной программы 1 [C], написанной на языке Python, найдены частоты нуклеотидов A, T, G, C в репликонах бактерии. С помощью программы 2 [C] найдены частоты кодонов, кодирующих аминокислоты аспарагин и глутамин.
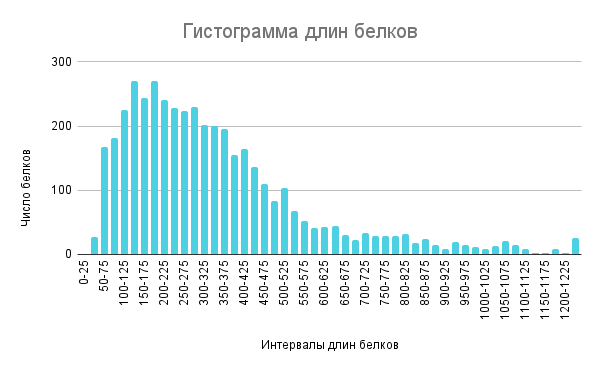
1. Длины белков, закодированных в геноме бактерии Mucilaginibacter robiniae

Большая часть белков бактерии имеют длину от 50 до 525. Наибольшее количество белков имеют длину 150-175.
2. Гистограмма GC-состава по CDS
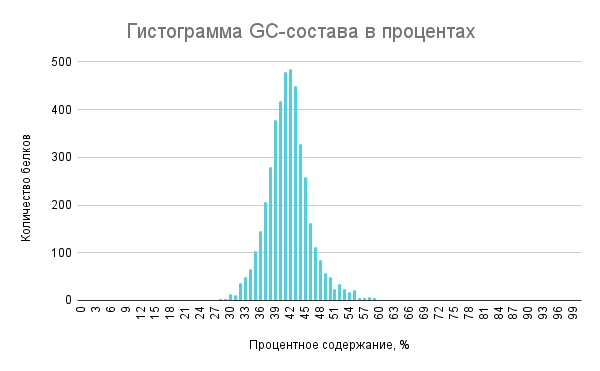
GC-состав показывает процентное содержание гуанина и цитозина среди всех остальных нуклеотидов в цепи [6]. У бактерии Mucilaginibacter robiniae содержание GC у большинства белков колеблется от 39% до 45%.
3. Исследование состава нуклеотидов A, T, G, C по репликонам
| Процентное содержание нуклеотидов в репликонах, % | ||||
|---|---|---|---|---|
| Репликон | Аденин | Гуанин | Цитозин | Тимин |
| Хромосома | 29.327 | 20.731 | 20.647 | 29.291 |
| Плазмида | 28.498 | 20.002 | 21.295 | 30.205 |
По таблице можно увидеть, что состав нуклеотидов в репликонах бактерии примерно одинаков и составляет по 30% содержания аденина и 30% тимина, и по 20% гуанина и 20% цитозина.
4. Содержание кодонов, кодирующих аспарагин и глутамин
| Кол-во триплетов | % содержание | |
|---|---|---|
| Аспарагин | ||
| TTA | 41854 | 56.846 |
| TTG | 31773 | 43.154 |
| Глутамин | ||
| GTT | 26983 | 85.376 |
| GTC | 4622 | 14.624 |
Mucilaginibacter играют ключевую роль в расщеплении полисахаридов. Среди штаммов бактерий разных видов за расщепление полисахаридов чаще всего отвечают RH9 белки, содержащие две каталитические аминокислоты, идентифицированные как остатки аспарата и глутамата [4]. По полученным данным можно увидеть, что содержание триплетов, соответствующим аминокислотам аспарагин и глутамин, невелико в геноме Mucilaginibacter robiniae. Для бактерии аспарагин чаще всего всего кодируется кодоном TTA (56.846%), а глутамин кодируется GTT (85.376%).