Для сравнения множественного выравнивания были выбраны белковые последовательности (seed) из семейства PF00053. Было сделано три выравнивания - muscle, mufft, tcoffee. Сравнение проводилось программ mufft и tcoffee с muscle, выбранным в качестве референсного. Для сравнения использовалась программа MACHO одногруппников.
Результаты сравнения программ Muscle и MAFFT соответсвенно:
Длина первой последовательности: 94
Длина второй последовательности: 115
Процент совпадающих колонок для первой последовательности: 15,96 %
Процент совпадающих колонок для второй последовательности: 13,04 %
| Block | Alignment_1 | Alignment_2 |
|---|---|---|
| 1 | 1-3 | 1-3 |
| 2 | 61-72 | 76-87 |
Результаты сравнения программ Muscle и tcoffeе соответсвенно:
Длина первой последовательности: 94
Длина второй последовательности: 107
Процент совпадающих колонок для первой последовательности: 21,28 %
Процент совпадающих колонок для второй последовательности: 18,69 %
| Block | Alignment_1 | Alignment_2 |
|---|---|---|
| 1 | 1-3 | 1-3 |
| 2 | 55-55 | 68-68 |
| 3 | 58-72 | 71-85 |
| 4 | 94-94 | 107-107 |
Исходя из результатов, полученных с помощью программы MACHO, выравнивания оказались неодинаковыми. Выравнивание с помощью tcoffee оказалось более схожим с референсным, сделанным с помощью программы muscle.
Ссылка на проектДля сравнения пространственных структур были взяты белки из семейства PF00053 c PDB ID: 2y38 (LAMININ ALPHA5 CHAIN N-TERMINAL FRAGMENT), 4ove (X-ray Crystal Structure of Mouse Netrin-1), 8edk (Structure of C. elegans UNC-6 LamN and EGF domains). В начале было проведено совмещение сруктур с помощью сайта PDB, на его основе сделано множественное выравнивание и после множественное выравнивание с помощью программы Muscle. После два множественных выравнивания сравнивались с помощью программы MAHCO.
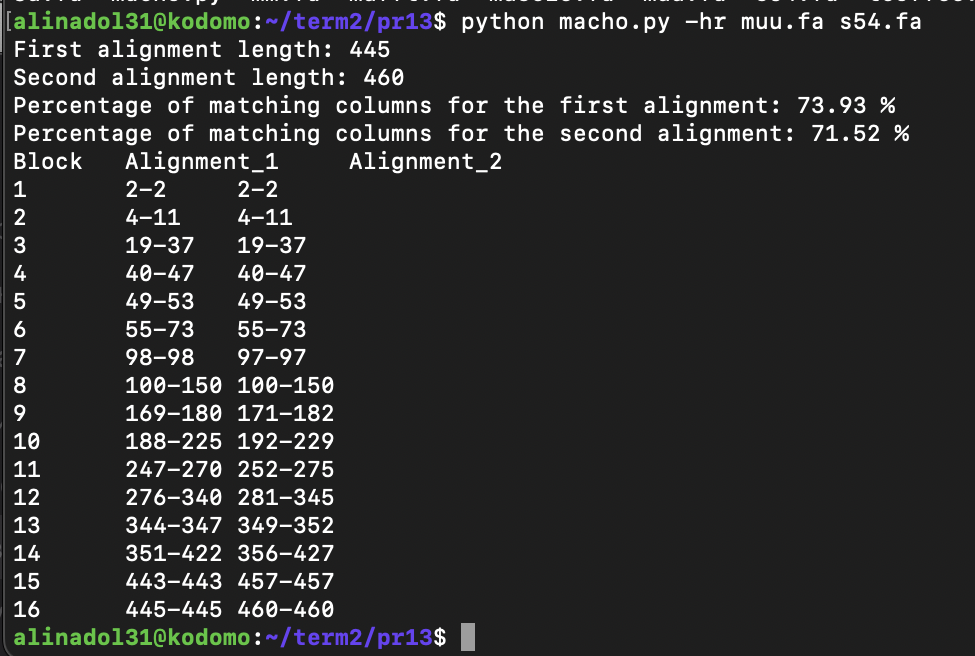
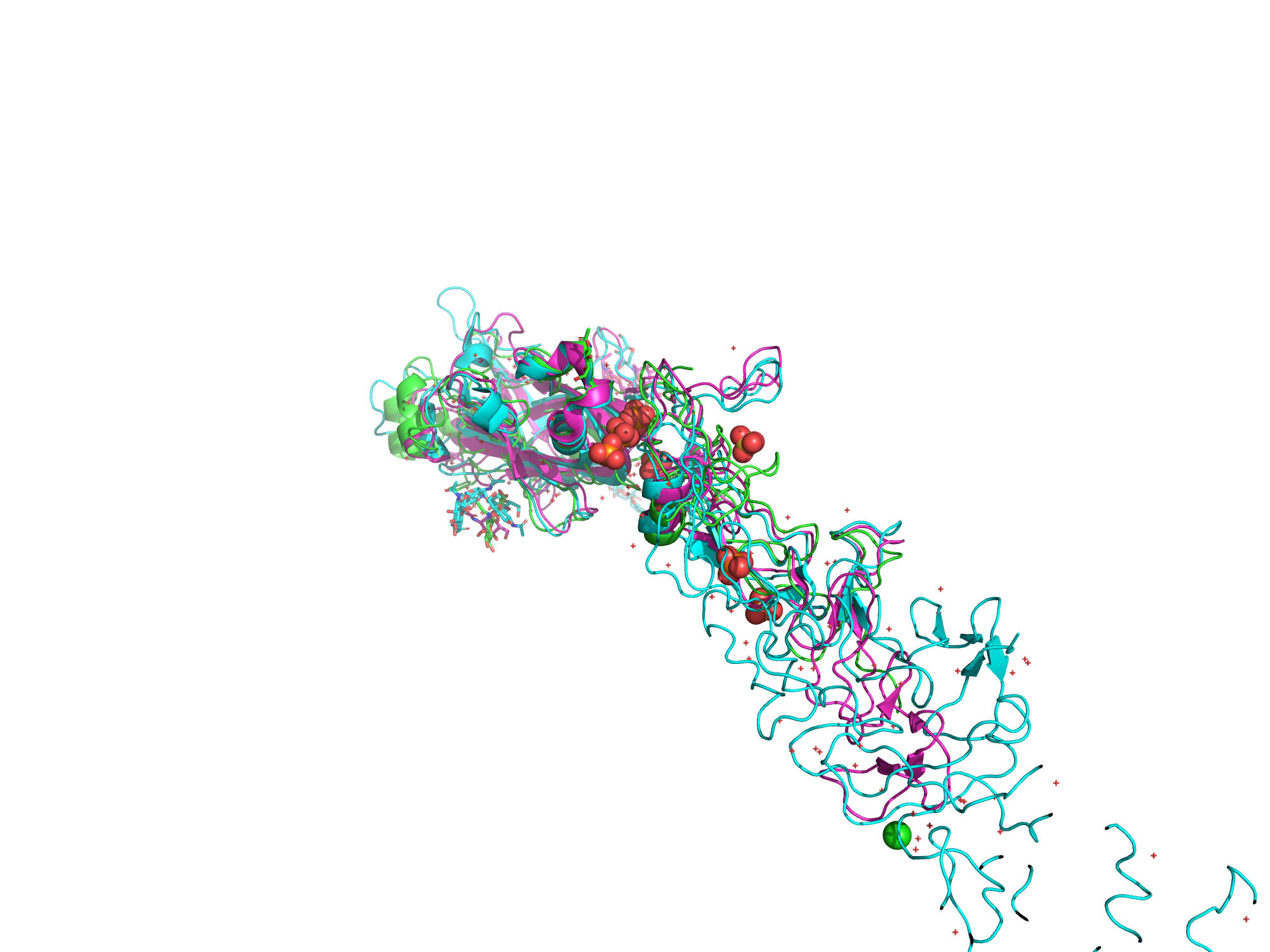
Выравнивания оказались похожими (73% схожести). Самое крупное различие находится в промежутке 427-460/423-445 в выравниваниях структурном и с помощью muscle соответсвенно. Данные различия могут связаны с тем, что белок с ID 2y38 имеет всего 2 структурных домена (Laminin-type EGF domain; Laminin, N-terminal), совпадающих и с другими двумя белками, в то время как 4ove имеет третий домен Galactose-binding-like domain, 8edk - EGF-like domain.

MAFFT (multiple alignment using fast Fourier transform) - программа для создания множественного выравнивания последовательностей аминокислоттных, либо же нуклеотидных.
В данный момент программа имеет несколько алгоритмов и режимов работы, изначально при выпуске в 2002 году она имела только алгоритм прогессивного выравнивания с группировкой последовательностей с помощью преобразования Фурье.
Попарное выравнивание - попарное выравнивание по всем последовательностям для определения схожих участков.
Матрица расстояний - вычисление матрицы расстояний для оценки различий между выравниваниями с помощью их показателей выравниваний.
Направляющее дерево - расположение кластеров в иерархической форме в направляемом дереве на основе матрицы расстояний, где ветви являются расстояниями между кластерами.
Прогрессивное выравнивание - выполняется прогрессивное выравнивание с помощью направояющего дерева от листьев к корню.
Итеративное выравнивание - повторение всего процесса с корректировкой позиций пропусков и вставок для повышения точности выравнивания.
Программа MAFFT имеет множество различных методов, адаптированных под конкретные задачи. Методы, ориентированные на Точность - L-INS-i (вероятно, наиболее точный метод; рекомендуется для последовательностей длиной менее 200), G-INS-i (подходит для последовательностей одинаковой длины; рекомендуется для последовательностей длиной менее 200), E-INS-i (подходит для последовательностей, содержащих большие невыравниваемые участки; рекомендуется для последовательностей длиной менее 200). Методы, ориентированные на скорость - FFT-NS-i (метод итеративной доработки; только два цикла), FFT-NS-2 , NW-NS-i, NW-NS-PartTree-1 (рекомендуется для последовательностей от ~10 000 до ~50 000).