Практикум 1. Филогенетическое дерево
Отобранные бактерии
| Название | Мнемоника |
|---|---|
| Rhizobium meliloti | RHIME |
| Saccharophagus degradans | SACD2 |
| Shewanella denitrificans | SHEDO |
| Yersinia pestis | YERPE |
| Burkholderia mallei | BURMA |
| Paracoccus denitrificans | PARDP |
| Proteus mirabilis | PROMH |
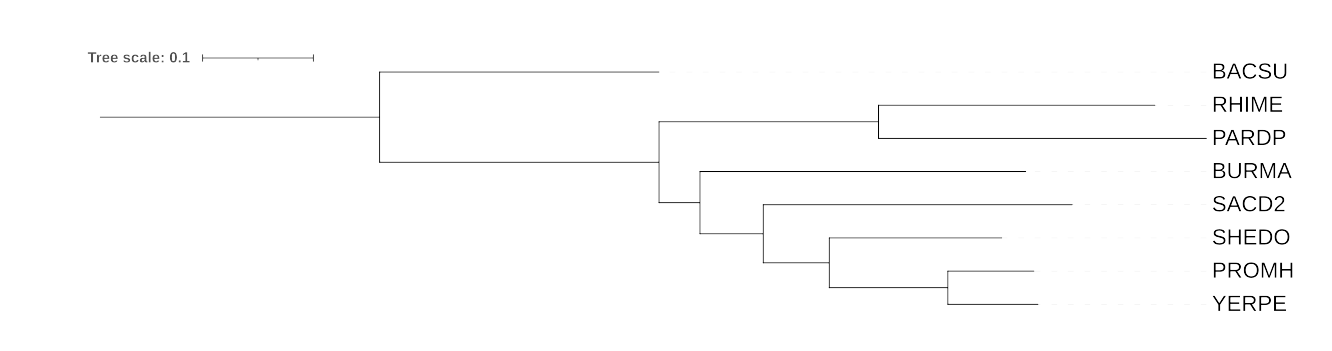
Скобочная формула дерева: ((PARDP,RHIME),(BURMA,(SACD2,(SHEDO,(PROMH,YERPE)))))
Изображение дерева
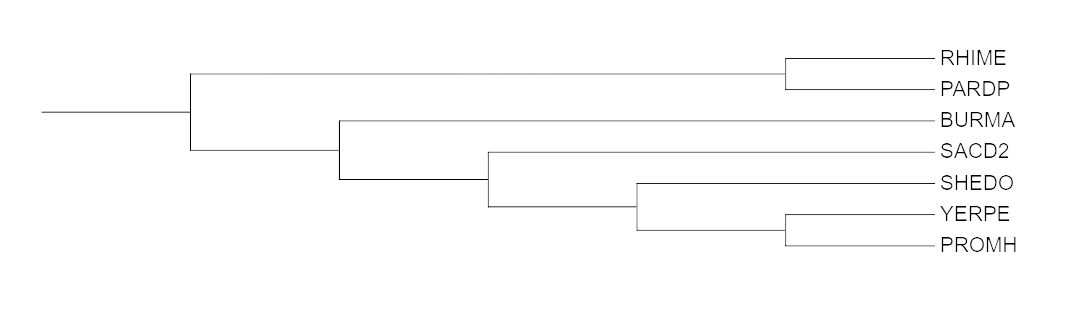
Нетривиальные ветви дерева
(PROMH,YERPE) vs. (RHIME,BURMA,SACD2,SHEDO,PARDP)- отделяют Enterobacterales
(PARDP,RHIME) vs. (BURMA,SACD2,SHEDO,PROMH,YERPE)- отделяют Alphaproteobacteria
(SHEDO,SACD2,YERPE,PROMH) vs. (PARDP,RHIME,BURMA)- отделяют Gammaproteobacteria
(PROMH,YERPE,SHEDO) vs. (RHIME,BURMA,SACD2,PARDP)
(SACD2,SHEDO,YERPE,PROMH,BURMA) vs. (PARDP,RHIME)
Практикум 2. Реконструкция филогении
Таксономические взаимоотношения среди отобранных мною бактерий:
| Название | Мнемоника | Таксономия |
|---|---|---|
| Burkholderia mallei | BURMA | Bacteria; Pseudomonadota; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Burkholderia; Burkholderia mallei |
| Saccharophagus degradans | SACD2 | Bacteria; Pseudomonadota; Gammaproteobacteria; Cellvibrionales; Cellvibrionaceae; Saccharophagus; Saccharophagus degradans |
| Paracoccus denitrificans | PARDP | Bacteria; Pseudomonadota; Alphaproteobacteria; Rhodobacterales; Paracoccaceae; Paracoccus; Paracoccus denitrificans |
| Proteus mirabilis | PROMH | Bacteria; Pseudomonadota; Gammaproteobacteria; Enterobacterales; Morganellaceae; Proteus |
| Sinorhizobium (bas. Rhizobium) meliloti | RHIME | Bacteria; Pseudomonadota; Alphaproteobacteria; Hyphomicrobiales; Rhizobiaceae; Sinorhizobium/Ensifer group; Sinorhizobium |
| Shewanella denitrificans | SHEDO | Bacteria; Pseudomonadota; Gammaproteobacteria; Alteromonadales; Shewanellaceae; Shewanella |
| Yersinia pestis | YERPE | Bacteria; Pseudomonadota; Gammaproteobacteria; Enterobacterales; Yersiniaceae; Yersinia; Yersinia pseudotuberculosis complex |
Реконструкцию филогенетического дерева я решила производить по семейству белков, выполняющих функцию SECA(АТФаза, ответственная за перенос препротеина через цитоплазматическую мембрану). Из базы данных Uni-Prot были получены последовательности белков с функцией SECA для отобранных мною бактерий в формате Fasta-файла .
На сайте NGPhylogeny.fr была проведена реконструкция деревьем тремя алгоритмами: FastME (минимальная эволюция), TNT (максимальная экономия), PhyML (максимальное правдоподобие) (рис 2-6).

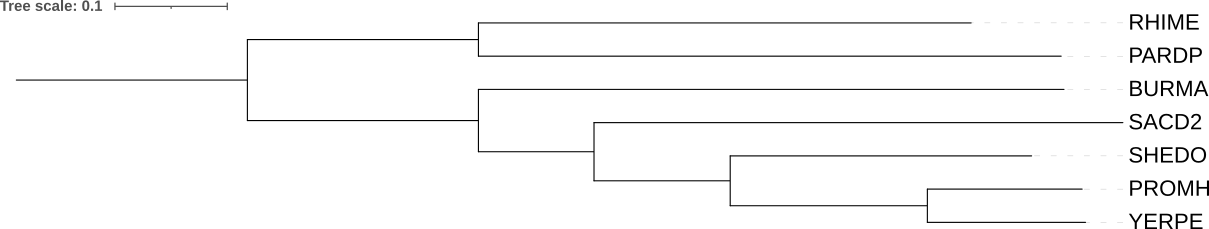
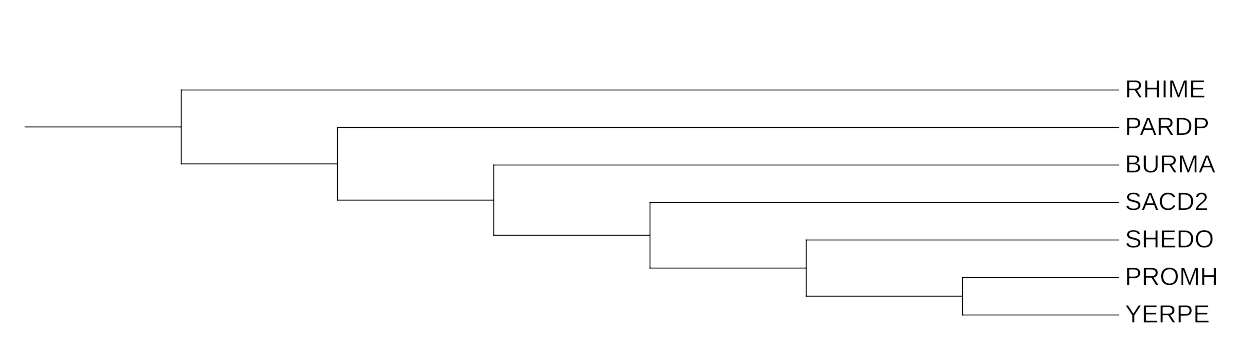

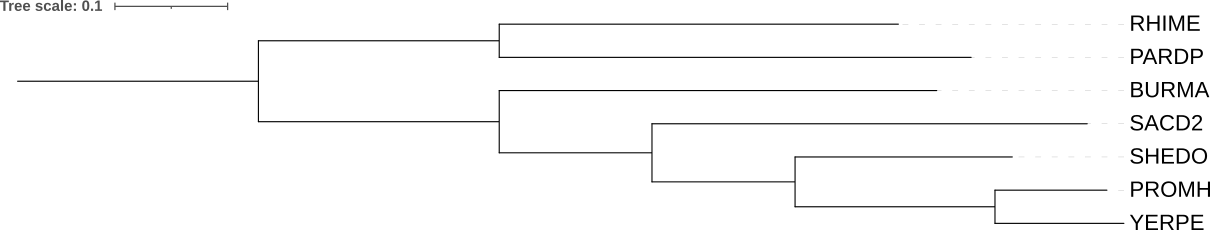
Практикум 3. Укоренение и бутстреп
Бутстреп анализ используется для оценки устойчивости и надежности топологии дерева, путем создания множества псевдовыборок из исходных данных и повторного анализа.
- Как это работает:
- Из исходного набора данных создаются множества псевдовыборок путем случайной выборки с возвращением.
- Для каждой псевдовыборки строится филогенетическое дерево.
- Путем анализа множества построенных деревьев определяется, насколько часто конкретная ветвь (или узел) встречается в этих деревьях.
- Мутации в нуклеотидах могут привести к изменениям в аминокислотной последовательности. Некоторые мутации могут быть нейтральными с точки зрения аминокислот, но изменять нуклеотидную последовательность, что может повлиять на реконструкцию филогенетических деревьев.
- Селекция на уровне аминокислот, когда эволюционные процессы могут приводить к изменениям в аминокислотной последовательности, которые не всегда отражаются в нуклеотидной последовательности. Это может отражаться при построении филогенетических деревьях.
Значение бутстрепа выражается в процентах (обычно от 0 до 100) и указывает на частоту, с которой конкретная ветвь или узел встречается в построенных деревьях. Чем выше значение бутстрепа, тем более надежной считается эта ветвь или узел.

Для построения дерева использовался конвейр MAFT-FastME, при этом топология деревьев не изменилась при изменении способа укоренения. Само по себе укоренение с использованием аутгруппы это процесс, при котором к дереву добавляется дополнительная ветвь (внешняя группа), которая не имеет общих предков с остальными таксонами в дереве. Укоренение внешней группой помогает определить, какие из двух направлений эволюции более вероятно для рассматриваемых таксонов. Путем сравнения последовательностей ДНК между внешней группой и внутренними таксонами можно определить, какие таксоны более близки к общему предку, а какие более далеки. Таким образом, укоренение внешней группой помогает строить более точные филогенетические деревья.
Построение дерева по нуклеотидным последовательностям
Из базы данных NCBI был получен файл с нуклеотидными последовательностями для 8 организмов
После применения алгоритмов MAFT-FastME и бутстрепа с 1000 репликами было получено дерево (рис.9). Из него видно, что достаточным индексом достоверности обладают только внутренние узлы. И топлогия отличается у деревьев, построенных на основе аминокислотных или нуклеотидных последовательностей.
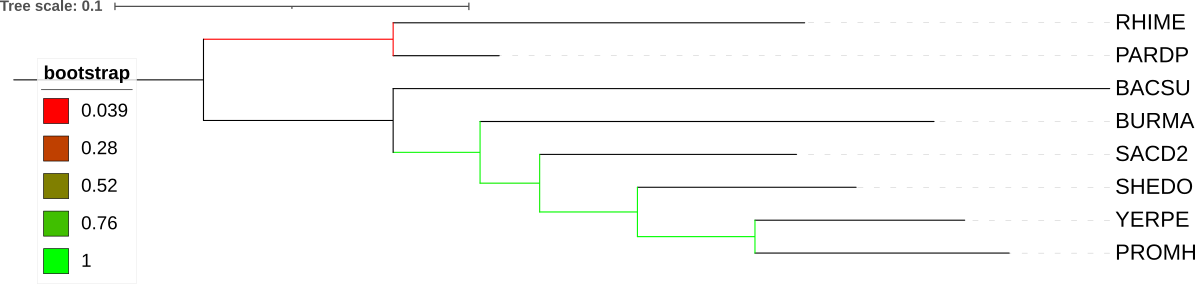
Newick formula: ((RHIME,PARDP),(BACSU,(BURMA,(SACD2,(SHEDO,(YERPE,PROMH))))))
Возможные причины изменения топологии дерева: