Составление списка гомологичных белков, включающих паралоги
Отобранные бактерии
| Название бактерии | Мнемоника |
|---|---|
| Rhizobium meliloti | RHIME |
| Saccharophagus degradans | SACD2 |
| Shewanella denitrificans | SHEDO |
| Yersinia pestis | YERPE |
| Burkholderia mallei | BURMA |
| Paracoccus denitrificans | PARDP |
| Proteus mirabilis | PROMH |
Составление списка гомологичных белков, включающих паралоги.
Результаты работы blasp представлены ниже или посмотреть полный файл можно по ссылке. После получения файла с гомологами белков командой egrep 'sp | tr' homologi_pr4_sem4.txt > result были отобраны записи, содержащие названия и ID.
sp|Q8ZC66|CLPX_YERPE ATP-dependent Clp protease ATP-binding subun... 805 0.0
sp|B4EU54|CLPX_PROMH ATP-dependent Clp protease ATP-binding subun... 769 0.0
sp|Q12LA2|CLPX_SHEDO ATP-dependent Clp protease ATP-binding subun... 712 0.0
sp|Q21KA8|CLPX_SACD2 ATP-dependent Clp protease ATP-binding subun... 645 0.0
sp|Q62JK8|CLPX_BURMA ATP-dependent Clp protease ATP-binding subun... 617 0.0
sp|Q92QQ2|CLPX_RHIME ATP-dependent Clp protease ATP-binding subun... 596 0.0
sp|A1B1H7|CLPX_PARDP ATP-dependent Clp protease ATP-binding subun... 580 0.0
sp|A1B5T0|HSLU_PARDP ATP-dependent protease ATPase subunit HslU O... 103 3e-24
sp|Q21H71|HSLU_SACD2 ATP-dependent protease ATPase subunit HslU O... 99.8 7e-23
sp|B4F171|HSLU_PROMH ATP-dependent protease ATPase subunit HslU O... 96.7 9e-22
sp|Q8ZJJ5|HSLU_YERPE ATP-dependent protease ATPase subunit HslU O... 95.1 2e-21
sp|Q12IT8|HSLU_SHEDO ATP-dependent protease ATPase subunit HslU O... 94.0 6e-21
sp|Q92TA7|HSLU_RHIME ATP-dependent protease ATPase subunit HslU O... 92.8 1e-20
sp|Q62F00|HSLU_BURMA ATP-dependent protease ATPase subunit HslU O... 82.4 5e-17
tr|A0A5P8YGZ0|A0A5P8YGZ0_YERPE ATP-dependent Clp protease ATP-bin... 51.2 6e-07
tr|B4EV83|B4EV83_PROMH ATP-dependent Clp protease ATP-binding sub... 50.1 1e-06
tr|A1B8N4|A1B8N4_PARDP ATP-dependent Clp protease, ATP-binding su... 50.1 1e-06
tr|B4F2B3|B4F2B3_PROMH ATP-dependent zinc metalloprotease FtsH OS... 46.6 2e-05
tr|A0A5P8YB42|A0A5P8YB42_YERPE ATP-dependent protease OS=Yersinia... 46.2 2e-05
tr|A0A5P8YCE6|A0A5P8YCE6_YERPE ATP-dependent zinc metalloprotease... 45.8 3e-05
tr|Q92M98|Q92M98_RHIME ATP-dependent zinc metalloprotease FtsH OS... 45.4 4e-05
tr|A1AZV8|A1AZV8_PARDP ATP-dependent zinc metalloprotease FtsH OS... 44.3 9e-05
tr|A1AZV8|A0A0H2WJ72_BURMA ATP-dependent zinc metalloprotease... 43.9 1e-04
sp|A1AZW1|RUVB_PARDP Holliday junction branch migration complex s... 43.1 2e-04
tr|A1BBJ2|A1BBJ2_PARDP ATP-dependent zinc metalloprotease FtsH OS... 41.6 7e-04
tr|A1AY35|A1AY35_PARDP Chaperone protein ClpB OS=Paracoccus denit... 41.6 8e-04
tr|Q12QI8|Q12QI8_SHEDO ATP-dependent zinc metalloprotease FtsH OS... 41.2 0.001
Итого 26 находок с порогом E-value 0,001 и меньше.
Реконструкция и визуализация
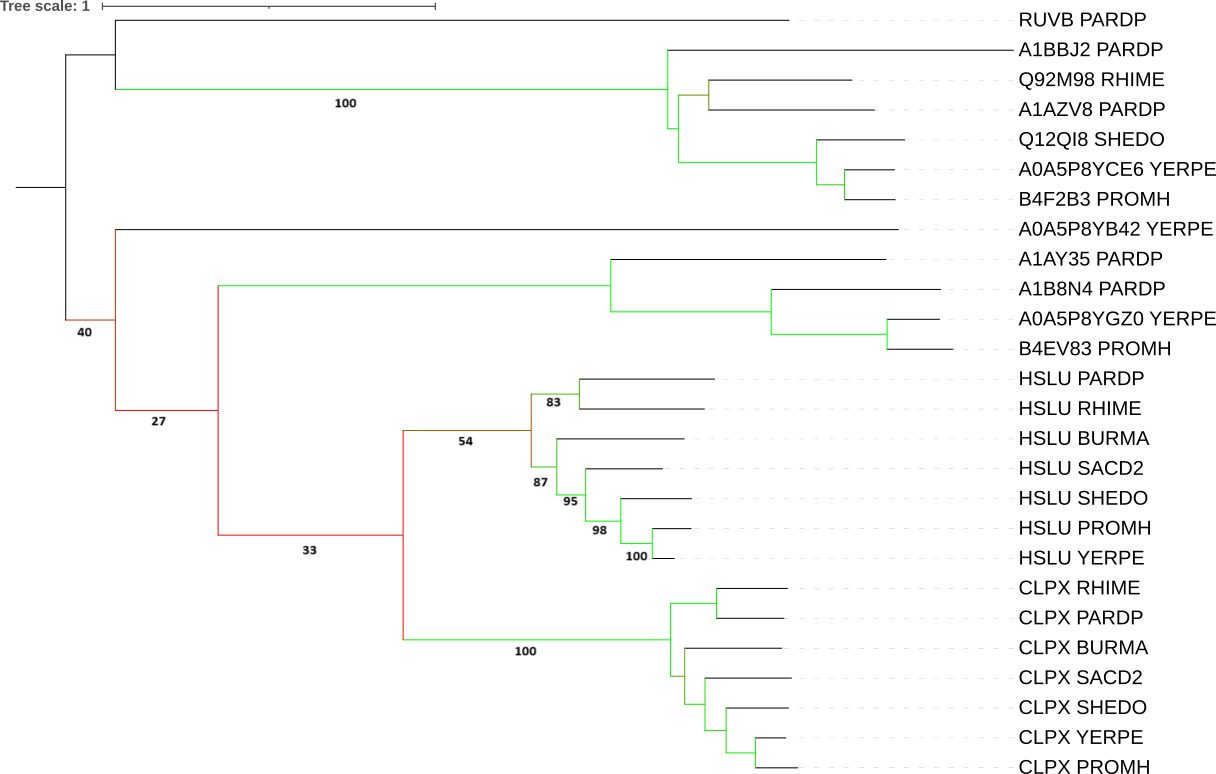
Сначала был составлен fasta-файл с последовательностями 26 находки. Построено филогенетическое дерево на сайте NGPhylogeny.fr, с помощью конвейра FastME-MAFFT.
Для программы FastME были изменены параметры:
| Gamma distributed rates across sites | No |
|---|---|
| Starting tree | BIONJ, No refinement |
| Bootstrap branch supports | Yes,100 replicates |
Дерево укорено в middlepoint для лучшей визуализации ортологов.
Ортологи: HSLU-PROMH и HSLU-YERPE, HSLU-PARDP HSLU-RHIME,A0A5P8YGZ0_YERPE И B4EV83_PROMH
Паралоги: A0A5P8YB42_YERPE и A0A5P8YCE6_YERPE,A1BBJ2_PARDP и A1AY35_PARDP,CLPX_BURMA и HSLU_BURMA.
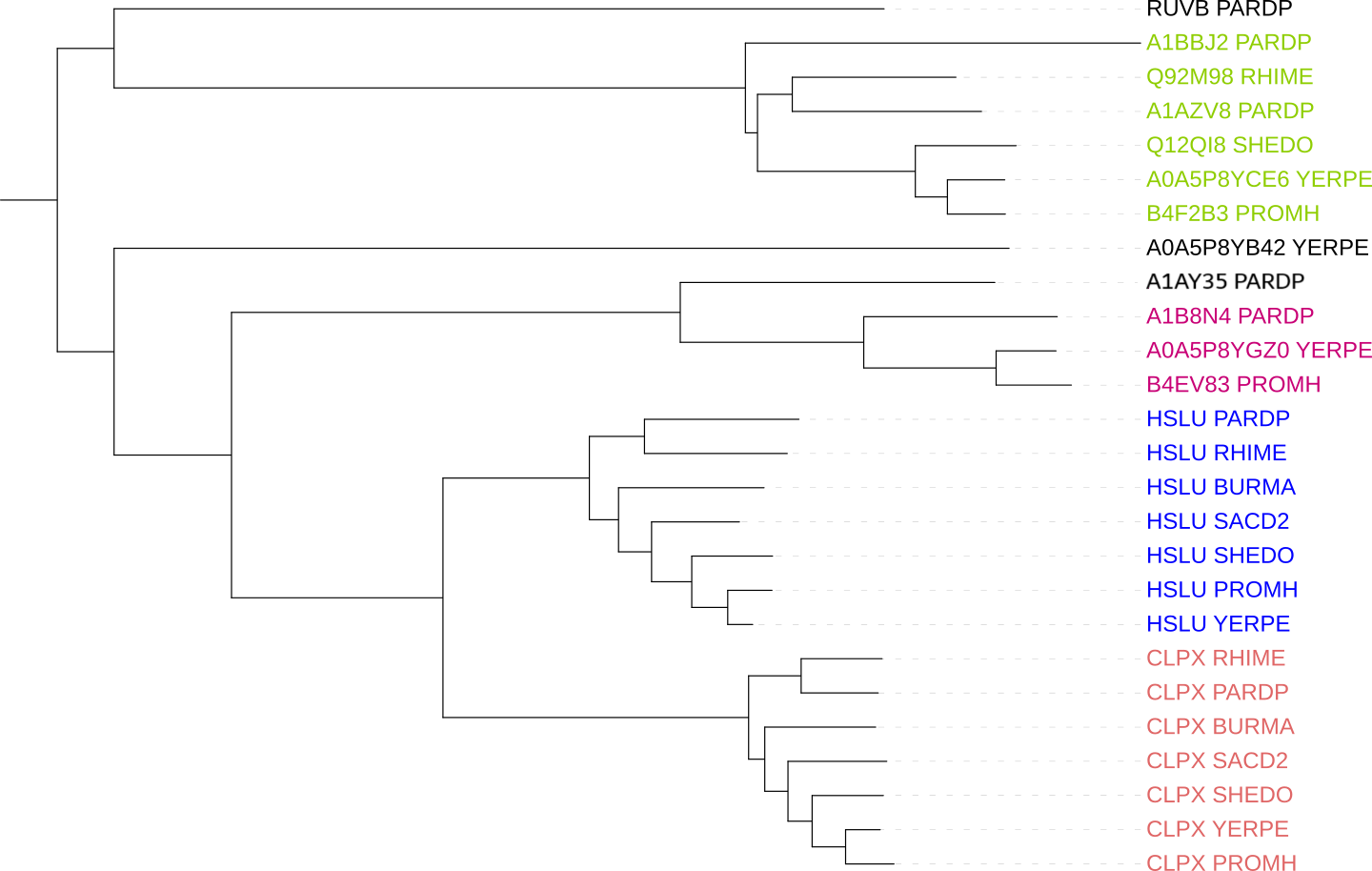

Описание семейств белков:
FtsH- АТФ-зависимая цинковая металлопротеаза, участвующая в деградации неправильно собранных белков. Гомологи этого белка были найдены только для 7 из 8 бактерий (не найдено для RURMA). Топология дерева не отличается.
Clp protease- АТФ-связывающая субъединица ClpA протеазы Clp, расщепляющая, например, казеин и альбумин. Белки найдены только для 3 из 7 бактерий (не найдено для RHIME,SACD2,SHEDO,BURMA). Топология дерева соостветсвует реконструированной филогении бактерий.
HSLU- АТФ-связывающая субъединица комплекса деградации, также обладает шаперонной активностью. Гомологи данного белка найдены для всех отобранных бактерий. Топология дерева не отличается от реконструированной филогении бактерий.
CLPX- АТФ-связывающая субъединица протеазы Clp. Гомологи данного белка найдены для всех отобранных бактерий. Топология дерева не отличается от реконструированной филогении бактерий.