Сравнение предсказаний трансмембранных участков в бета-листовом белке
Выбранный белок:- Type
- - transmembrane
- Class
- - beta-barrel transmembrane
- Superfamily
- - mitochondrial and plastid porins
- Family
- - Voltage-dependent anion channel (VDAC) porin
- Superfamily
- - mitochondrial and plastid porins
- Protein name
- - VDAC-1 channel
- PDB
- - 3emn, 4c69
- UniProt
- - VDAC1_MOUSE, Q60932
- Функции
- - позволяет АТФ диффундировать из митохондрий в цитоплазму, а также участвует в апоптозе, вызванным окислительным стрессом.
| Tilt- X | TM Segments | Coordinates | Coordinated predicted DeepTMHMM |
|---|---|---|---|
| 1 | 26-34 | - | 2 | 38-47 | - |
| 3 | 55-64 | 54-60 | 4 | 69-76 | 68-74 |
| 5 | 80-88 | 82-88 | |
| 6 | 69-76 | 94-100 | |
| 7 | 94-103 | 109-117 | |
| 8 | 122-131 | 125-131 | |
| 9 | 137-146 | 137-144 | |
| 10 | 149-156 | 149-157 | |
| 11 | 166-174 | 163-170 | |
| 12 | 178-185 | 179-186 | |
| 13 | 190-197 | 193-197 | |
| 14 | 202-210 | 203-208 | |
| 15 | 218-226 | 218-222 | |
| 16 | 231-238 | 231-237 | |
| 17 | 242-250 | 245-250 | |
| 18 | 255-263 | 256-262 | |
| 19 | 274-282 | 270-275 | |
| 20 | - | 287-294 | |
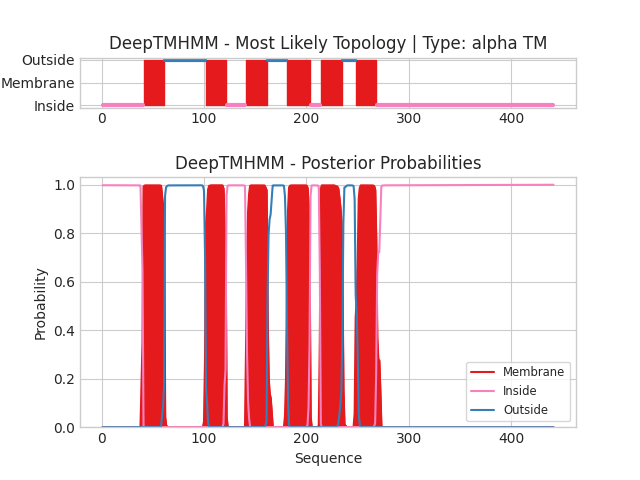
Предсказание структуры белка программой DeepTMHMM.
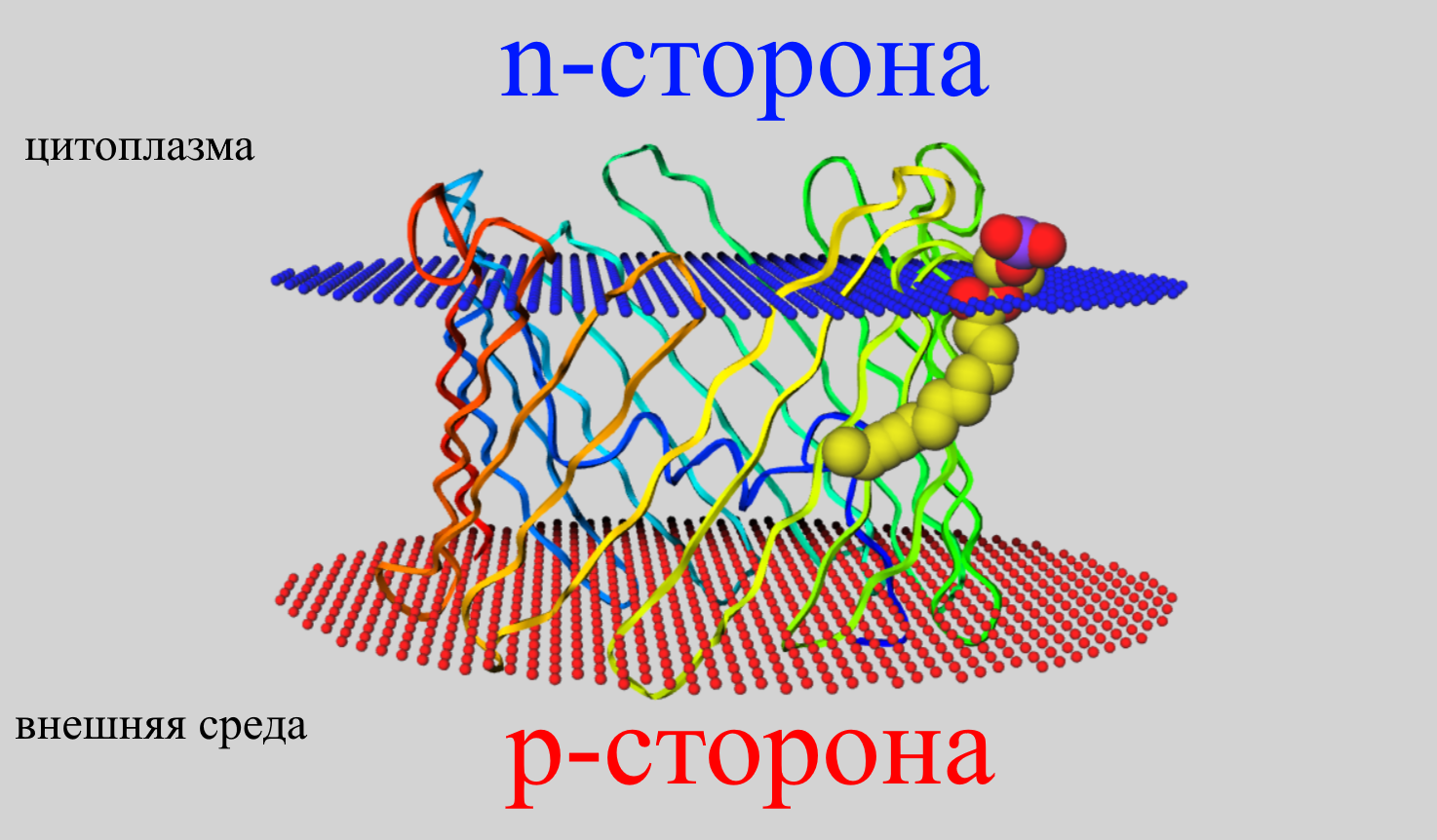
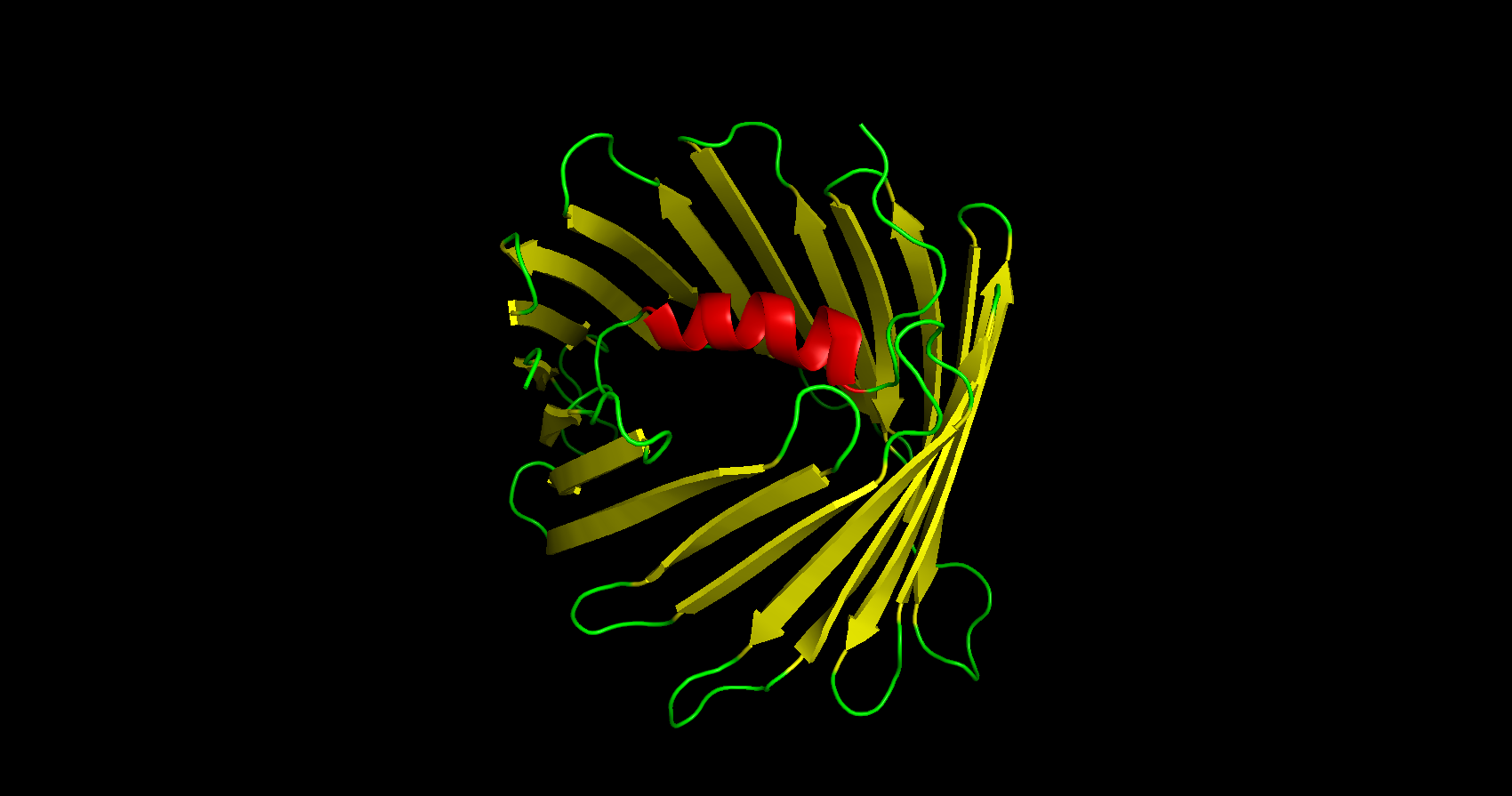
Для предсказания топологии мембраны трансмембранного белка была использована последовательность из базы данных UniProt (Q60932). По результатм работы программы, было найдено 18 бета-листов. Но по сведениям ЯМР анализа и X-Ray кристаллографии [1] VDAC-1 channel представляет собой β-бочонок, состоящий из 19 бета-листов, 18 петлеобразных соединений и N-концевой альфа-спирали, расположенной горизонтально посередине поры (рис.2.). Координаты, предсказанные DeepTMHMM сильно отличаются от коориднат, представленных в OPM. Такую разницу в координатах можно объяснить различными входными данными для OPM и DeppTMHMM: OPM обычно требует трехмерную структуру белка в качестве входных данных, в то время как DeepTMHMM требуется только аминокислотная последовательность белка. Доступность и качество этих входных данных могут повлиять на прогнозы обоих методов. Попробовав соотнести коориднаты, выяснилось, что координаты DeepTMHMM начинаются с 3 бета-листа и далее отличаются на 1-4 аминокислоты от таковых в OPM. Как видно из графика (рис.3.) первая петля была проигнорирована алгоритмом, а в положении 38-50, что соответсвует повялению второго бета-листа, алгоритм не предсказал его наличие, из-за низкой вероятности и шума. Текстовый результат выдачи программы доступен по ссылке.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке.
Мне был выдан белок Y1559_RALSO
| Название на русском | Название на английском языке | Идентификатор SwissProt | Название организма | Функции белка |
|---|---|---|---|---|
| Мембранный белок UPF0761 | UPF0761 membrane protein RSc1559 | Y1559_RALSO | Ralstonia nicotianae (strain GMI1000) | Транспорт веществ (?) |
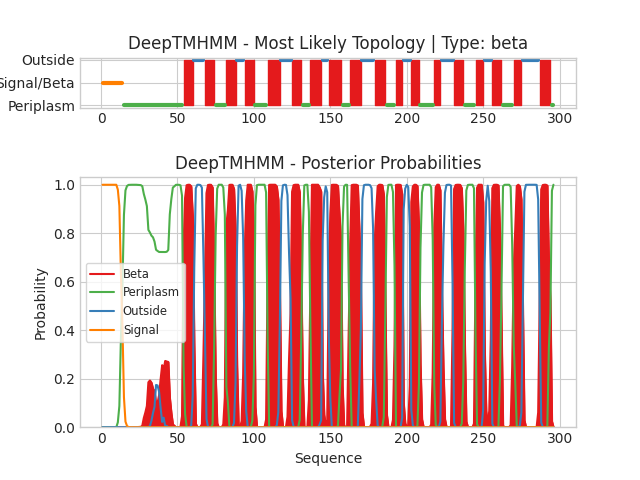
Последовательность белка была скачана в fasta-формате из базы данных UniProt и проанализирована DeepTMHMM. Было предсказано 6 альфа-спиралей. Результаты представлены в таблице 2. Предсказанные координаты практически полностью совпадают с координатами, указанными на старнице UniProt.
| Segments | Coordinated predicted DeepTMHMM | UniProt coordinates |
| 1 | 41-61 | 44-64 |
| 2 | 102-121 | 101-121 |
| 3 | 141-161 | 141-161 |
| 4 | 181-203 | 182-202 |
| 5 | 214-235 | 207-227 |
| 6 | 248-268 | 248-268 |
Далее стркутура, предсказанную при помощи AlphaFold была скачана и подана в алгоритм PPM со следующими параметрами: Type of membrane — Gram-negative bacteria inner membrane, Allow curvature — no, Topology (N-ter) — in ( DeepTMHMM предсказал положение N-конца внутри). Результаты представлены в табл. 3, получившийся на выходе pdb файл можно скачать по ссылке. Как можно заметить все 6 альфа-спиралей были предсказаны, но координаты некоторых из них отличаются иногда до 10 аминокислот. Качество предсказания в разных участках полученной модели может влиять на результаты прогнозирования PPM. Регионы с высоким качеством предсказания, вероятно, будут более точными и надежными, в то время как участки с низким качеством предсказания могут иметь погрешности. Если модель низкого качества или содержит области неопределенности, основанные на ней прогнозы могут быть ненадежными.
| Subunits | Tilt | Segments |
|---|---|---|
| Embedded residues: | ||
| A | 13 | 3,5,8,11,13,16-17,19-21,23-24,27-28,30-64,67,95,99,101-126,128-129,135-165,176-204,208-235,238,241,245,248-272,275 |
| Transmembrane secondary structure segments: | ||
| A | 3 | 1( 30- 62), 2( 101- 129), 3( 137- 162), 4( 177- 203), 5( 210- 235), 6( 250- 272) |