Выбор и скачивание протеомов.
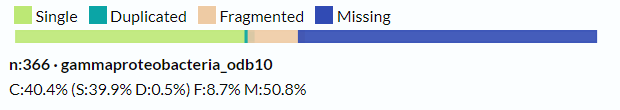
В прошлом практикуме был анализирован белок Chaperone protein DnaK- с протеомом ID UP000886100. Его CPD- Outlier (low value) , то есть размер протеома ниже среднего по таксономической группе. BUSCO показывает, что в протеоме 40.4% покрытия ортологичных генов, а значение Single(процент генов с единственной копией) имеют 39.9% генов (рис.1).
В качестве протеома сравнения был выбран протеом с ID UP000537130, принадлежащий Litorivivens lipolytica, который как и Thiolapillus brandeum относится к Gammaproteobacteria (рис.2).
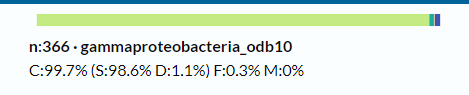
CPD протеома- Outlier (low value) , а статусы BUSCO имеют значение 99.7% для покрытия ортологичных генов и 98.6% Single (рис.3).
Скачивание протеомов на kodomo.
Получение протеома бактерии Litorivivens lipolytica. Количество белков в запросе на UniProtKB совпадает с количеством белков в протеоме на Proteoms.
wget 'https://rest.uniprot.org/uniprotkb/stream?compressed=true&format=txt&query=proteome:UP000537130' -O UP000537130.swiss.gz
Получение протеома бактерии Thiolapillus brandeum.
wget 'https://rest.uniprot.org/uniprotkb/stream?compressed=true&format=txt&query=proteome:UP000886100' -O UP000886100.swiss.gz
У обоих бактерий для предсказания через CPD слишком мало близкородственных протеомов. Важно заметить, что у Thiolapillus brandeum нехорошие показатели BUSCO: C:40.4% (S:39.9% D:0.5%) F:8.7% M:50.8%, что может влиять на результаты сравнения.
Сравнение протеомов.
Размер протеома Thiolapillus brandeum- 1,482 белковых записей;Размер протеома Litorivivens lipolytica- 3,241.
Thiolapillus brandeum:
Запрос для подсчета трансмембранных белков (и белков, образующих трансмембранные комплексы):
(proteome:UP000886100) AND (ft_transmem:*))
298 результатов из 1482, 20% протеома>
Запрос для подсчета ферментов. Названия (хотя бы одно из) подавляющего большинства ферментов заканчиваются на *ase:
(proteome:UP000886100) AND (protein_name:*ase)
769 результатов, 52% протеома.Использование не слишком специфичных фильтров конечно не даст максимально точных результатов, но позволит покрыть максимальное число белков с соответствующими функциями, т.к. такие свойства как внутриклеточное положение или EC систематика могут быть не указаны (недостаточно аннотированные белки протеома).Не стоит забывать, что эти две функциональные группы - пересекающиеся множества: ферменты иногда могут быть трансмембранными белками или хотя бы состоять в их комплексах.
В качестве третьей группы взяты рибосомальные белки, которых в протеоме 60 штук (4% протеома), эта информация взята из моего мини-обзора.
Litorivivens lipolytica:
Запрос для подсчета трансмембранных белков:
(proteome:UP000537130) AND (ft_transmem:*)
565 результатов, 16% протеома
Запрос для подсчета ферментов:
(proteome:UP000537130) AND (protein_name:*ase)
1736 результатов, 54% протеома. В целом видно, что трансмембранные белки и ферменты занимают почти одинаковые доли протеома у обеих бактерий, если конечно запросы, использованные для подсчета белков, достаточно эффективны. Количество же рибосомальных белков остается почти постоянным для всех гаммапротеобактерий и мало связано с размером протеома.
Сравнение протеомов по...
Оценка количества белков теплового шока у Thiolapillus brandeum выдало 1 результата с белкoм Protein GrpE, HSP-70 cofactor.
Поиск производился по:(proteome:UP000886100) AND (protein_name:"hsp*")
Аналогичный поиск для Litorivivens lipolytica выдал 1 результата.(proteome:UP000537130) AND (protein_name:"hsp*")
Получается, что бактерии имеют равное число белков теплового шока.