Выравнивание лизоцима форели с известной структурой 1lmp с данным мне лизоцимом LYS_BPN15 из Enterobacteria phage N15 (Bacteriophage N15).
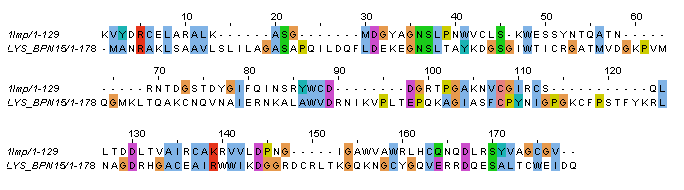
По-моему, белки не очень похожи, хоть оба и лизоцимы.
Выравнивание alig.pir
Модифицируем координаты 1lmp_now.ent
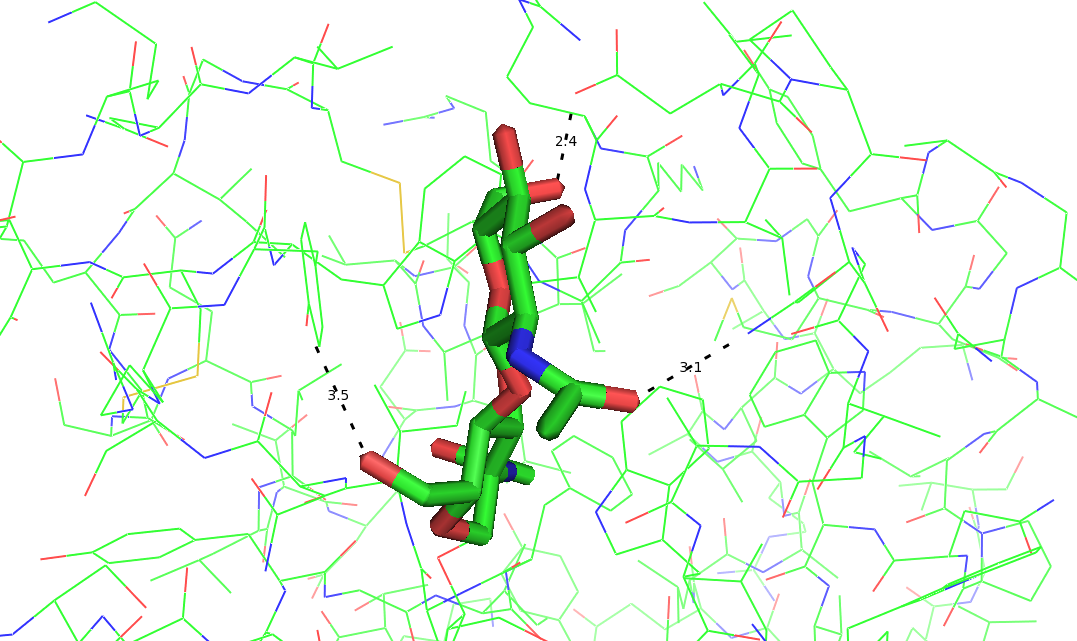
1. ('OD2:101:A', 'O6:130:A')
2. ('ND2:103:A', 'O7:130:A')
3. ('OH:62:A', 'O6:131:A')
или в варианте форматированных координат:
1. ('OD2:101:A', 'O6:130:A')
2. ('ND2:103:A', 'O7:130:A')
3. ('OH:62:A', 'O6:130:B')
1. ('OD2:143:A', 'O6:179:A')
2. ('ND2:145:A', 'O7:179:A')
3. ('OH:85:A', 'O6:179:B')
скрипт
модель 1
модель 2
модель 3
модель 4
модель 5
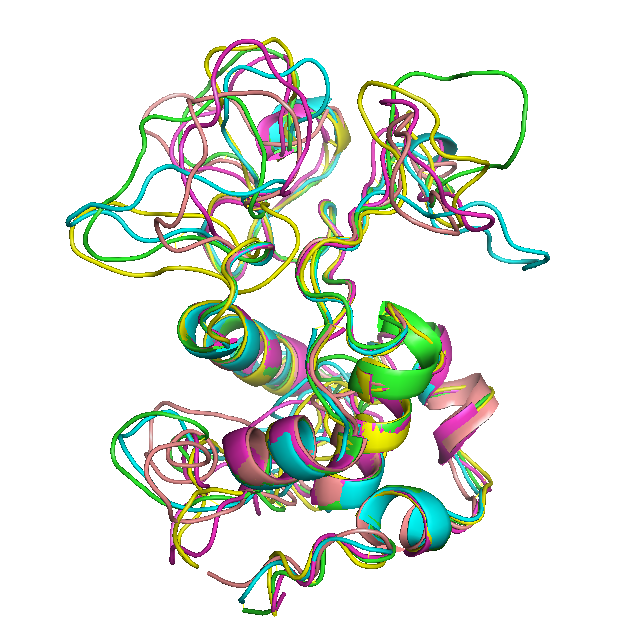
Спирали во всех моделях хорошо наложились друг на друга.
Модель Ramachandran Z-score Z-score for bond angles 1lmp -0.325 1.752 seq1 -1.144 1.454 seq2 -1.255 1.490 seq3 -0.994 1.448 seq4 -1.397 1.552 seq5 -0.791 1.409
Лучшая модель - seq5.