
|
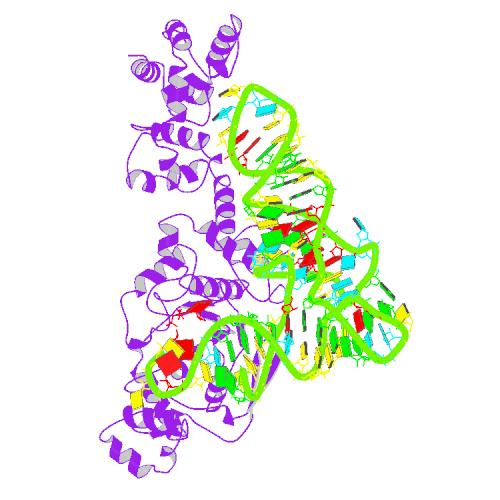
Организм: THERMUS THERMOPHILUS
В файле приведены координаты атомов следующих молекул:
PROTEIN ATOMS : 7628
NUCLEIC ACID ATOMS : 3194
HETEROGEN ATOMS : 64
SOLVENT ATOMS : 505
Для исследования была выбрана цепь С, представляющая тРНК со следующей последовательностью:
[501]GGCCCCAUCGUCUAGCGGUUAGGACGCGGCCCUCUCAAGGCCGAAACGGGGGUUCGAUUCCCCCUGGGGUCACCA[576]
где 501 и 576 - номера первого и последнего нуклеотидов.
Последовательность завершает триплет CCA, к которому может присоединиться аминокислота,
и в документе pdb приведены координаты всех его атомов.
Для С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями.
( trna.out).
В соответствии с полученными данными:
Акцепторный стебель:501-507 и 576-566
D-стебель:509-512 и 525-522
T-стебель:548-553 и 565-560
Антикодоновый стебель:533-527 и 543-538
|
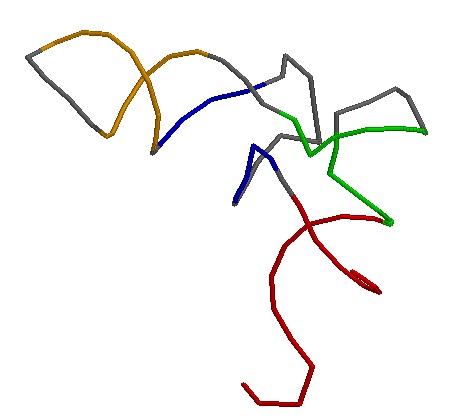
Вторичная структура тРНК из THERMUS THERMOPHILUS.
Акцепторный стебель - красный
D-стебель - синий
Т-стебель - зелёный
Антикодоновый - оранжевый
скрипт для RasMol
Структуру стеблевых дуплексов поддерживают 15 канонических и 5 неканонических пар оснований.
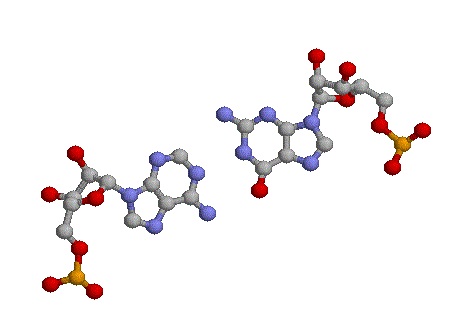
Пример неканонической пары G526-A544 |
Bозможность стекинг-взаимодействия между основаниями конца акцепторно стебля и начала Т-стебля.
На рисункe видно, что все основания лежат в одной плоскости и в значительной мере перекрываются. В пользу
стекинг-взаимодействий говорят и данные программы analyze пакета 3DNA.
Bозможность стекинг-взаимодействия между антикодоновым стеблем и D-стеблем аналогична.
Дополнительные водородные связи между основаниями D- и Т-петель я не обнаружила.