скобочная формула
дерево:
| 1 | Архитектура 1 (C3PT80) |
| 2 | Архитектура 2 (C5YZX3) |
| A | Arthropoda |
| CH | Chordata |
| N | Nematoda |
| C | Cnidaria |
| P | Placozoa |
| V | Viridiplantae |
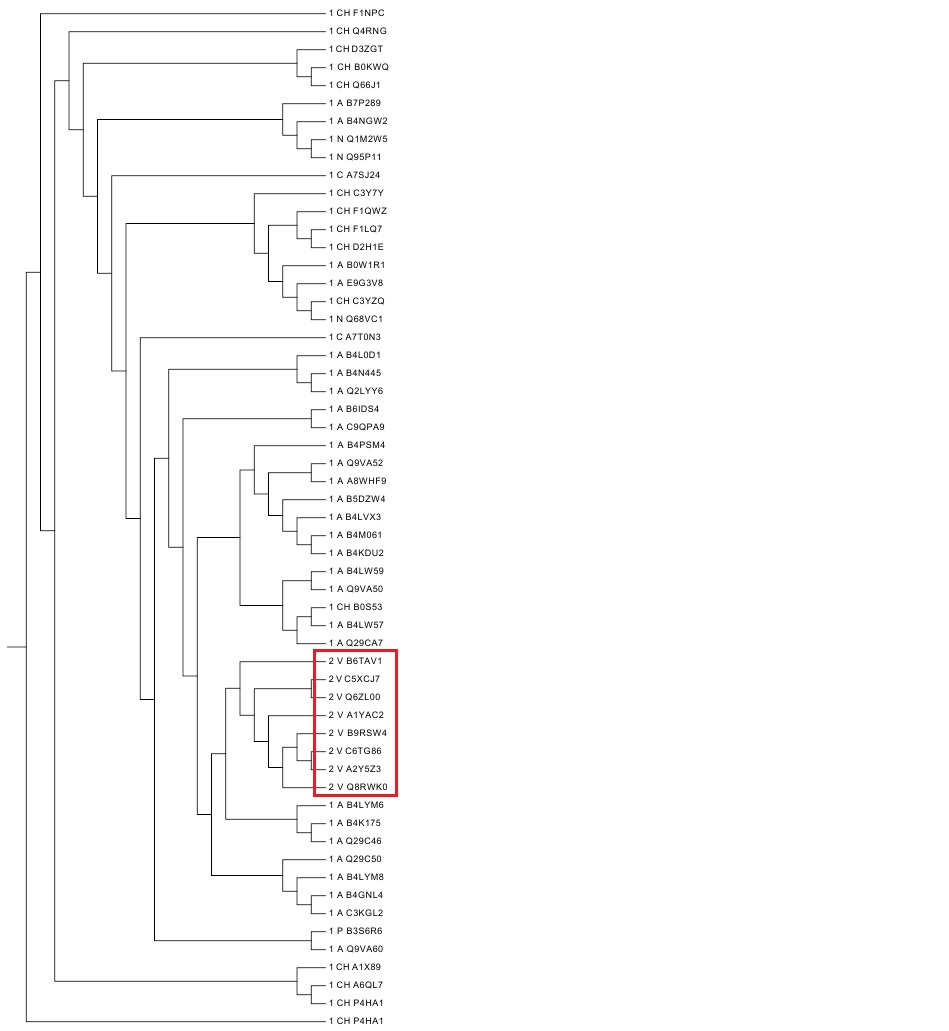
Филогенетическое дерево построено программой fprotpars на основе полученного выравнивания.
Молекулярные часы не учитывались, так как таксономия сильно различается. В результате программа выдала очень
много вариантов деревьев, меня устроил такой:
скобочная формула
дерево:

Итак, по построенному дереву мы явно видим, архитектура 2 в ходе эволюции компактно отделилась от
архитектуры 1, причем архитектура 2 присутствует только у одного таксона V, который в общем принадлежит к царству растений.
Выравнивание можно разделить по доменной архитектуре белков (1 и 2), так как доменные архитектуры образуют клады на дереве.
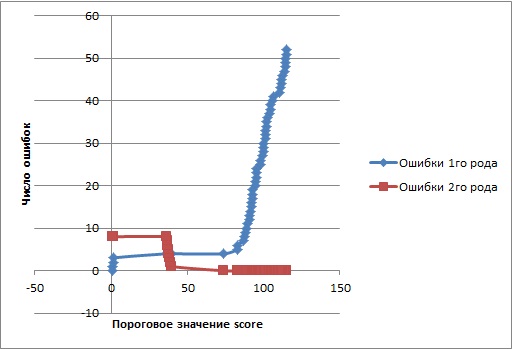
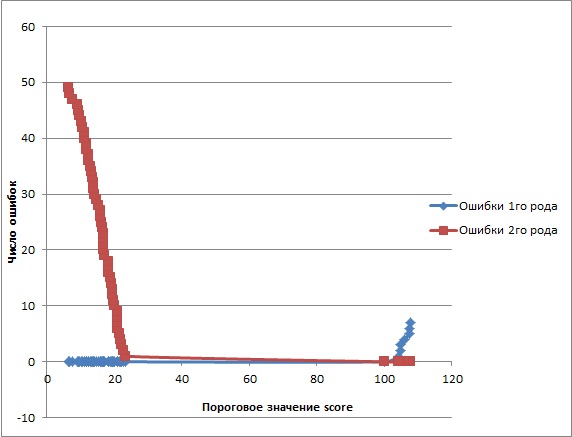
По построенным профилям для архитектур 1 и 2, сделаны следующие выводы:
| Архитектура 1 | Не Архитектура 1 | Всего | |
| Предсказание Архитектуры 1 | 49 | 0 | 49 |
| Предсказание не Архитектуры 1 | 4 | 8 | 12 |
| Всего | 53 | 8 | 61 |
| Архитектура 2 | Не Архитектура 2 | Всего | |
| Предсказание Архитектуры 2 | 8 | 0 | 8 |
| Предсказание не Архитектуры 2 | 0 | 49 | 49 |
| Всего | 8 | 49 | 57 |
Поиск по всему банку последовательностей SwissProt с помощью профиля для архитектуры 1 выдал 16 находок (порог = 74,0),
причем найдено только 2 последовательности принадлежащие к архитектуре 1, по которой строился профиль. Остальные
14 последовательностей имеют очень похожую доменную архитектуру: везде присутствует домен P4Ha_N (PF08336), а домен, который выбирался изначально
(2OG-FeII_Oxy (PF03171)) везде есть в модифицированном виде (на него навешаны ионы железа).
Все 16 последовательностей являются альфа субъединицами
Prolyl 4-hydroxylase, отсюда и их однообразие доменов.
Если изменить порог веса до 40 (число, которое следует из графиков ошибок), то в результате
находится уже 348 последовательностей.
Таким образом, для архитектуры 1 удалось создать достаточно неплохой профиль,
по крайней мере он находит последовательности из одного семейства, архитектуры которых очень похожи.
А поиск с помощью профиля для архитектуры 2, к сожалению, прошел не так хорошо. Так проведя поиск по установленному порогу = 100, не
нашлось ни одной последовательности вообще. Поиск с порогом 50 тоже ничего не нашел, с порого 30 - 9 последовательностей, ни одна из которых
не принадлежит к последовательности с архитектурой 2, по которым строился профиль. Зато 2 последовательности из
найденных 9ти принадлежат к архитектуре 1, по которой строился профиль. Это странно. И вообще все найденные последовательности
имеют архитектуру 1 и являются альфа субъединицами Prolyl 4-hydroxylase. Таким образом, построенный профиль плохой, неправильный. А может
последовательностей с архитектурой 2, по которым строился профиль не было в банке swissprot (если такое возможно), тогда
этим можно объяснить, что последовательностей вообще не нашлось и что с порогами 100 и 50 - нет результатов.
Для интереса был проведен еще поиск с порогом 10. Найдено 18 последовательностей - то же самое: 2 - с архитектурой 1 и ни одной с архитектурой 2.
Таким образом, для архитектуры 2 не удалось создать профиля, это может быть связано, что в этом случае для его
построения использовали намного меньше последовательностей в выравнивании, чем для профиля с архитектурой 1.