Реконструкция филогенетических деревьев
Поиск диагностических позиций выравнивания
Сначала я разделила выравнивания по ветвям {CLOTE, CLOB1}, {FINM2, LACAC} и остальные 3 последовательности, т.е.
отделила Clostridium и Bacillaceae от остальных:

Видно, что Clostridium (синие) четко выделяются, поэтому для них диагностическими позициями можно назвать следующие участки:
2-85, 155-233.
Теперь отделим Bacilli от Clostridia, т.е. {LACAC, ENTFA, BACSU, GEOKA} от {CLOTE, CLOB1, FINM2}:
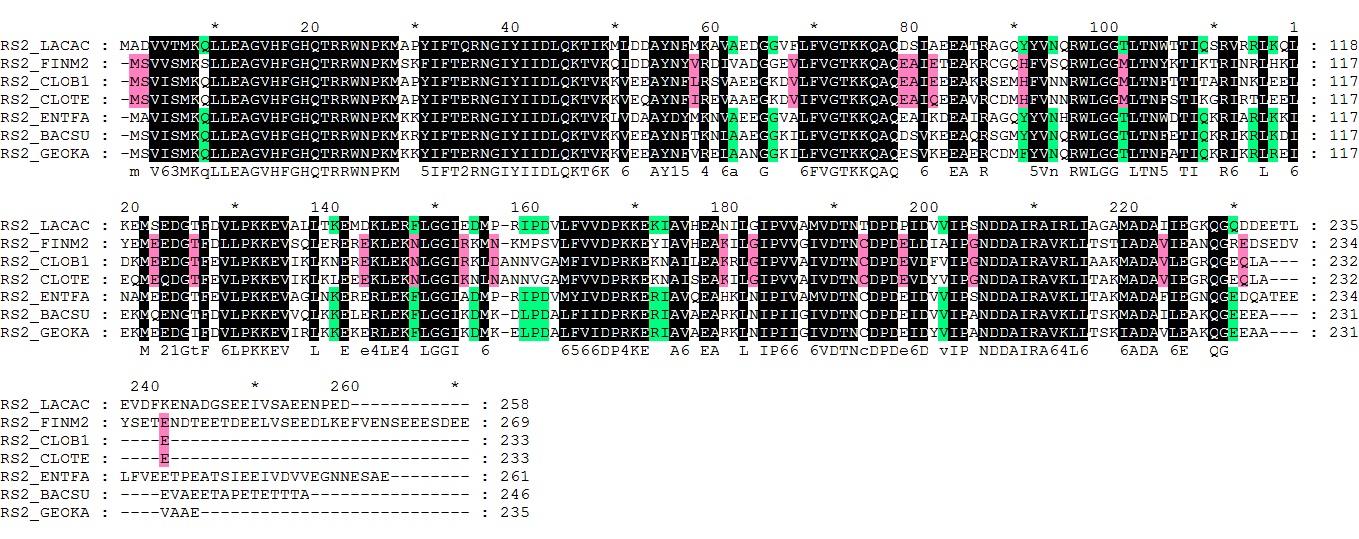
Здесь диагностическими позициями я бы назвала участки:
для Bacilli (зеленый): 4-49, 91-118, 160-179.
для Clostridia (розовый): 2-59, 68-118, 164-231.
Таким образом, построив несколько выравниваний можно предугадать структуру дерева.
Знакомство с сервисом TreeTop
На вход было подано полученное выравнивание. Впечатлило, что сервис выдал результат буквально через 3 секунды, даже не пришлось ждать.
Причем сразу было постороено и дерево и матрица расстояний, так что можно не использовать несколько программ для получения того же результата:
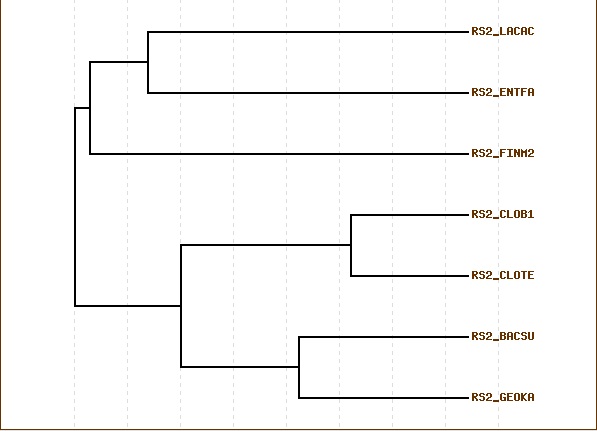
Distance Matrix
1 2 3 4 5 6 7
1 RS2_LACAC 0.000 0.355 0.393 0.399 0.303 0.323 0.366
2 RS2_FINM2 0.355 0.000 0.399 0.393 0.361 0.369 0.386
3 RS2_CLOB1 0.393 0.399 0.000 0.112 0.387 0.302 0.251
4 RS2_CLOTE 0.399 0.393 0.112 0.000 0.392 0.306 0.233
5 RS2_ENTFA 0.303 0.361 0.387 0.392 0.000 0.286 0.331
6 RS2_BACSU 0.323 0.369 0.302 0.306 0.286 0.000 0.161
7 RS2_GEOKA 0.366 0.386 0.251 0.233 0.331 0.161 0.000
Помимо того показывается еще алгоритм построения, в том числе и скобочная формула.
Дерево можно представить и в угловой форме:
Минусом, на мой взгляд является то, что в качестве входного файла используется выравнивание, то есть придется привлекать к работе
программы выравнивания.
В целом, сервис мне понравился: достаточно простой, содержит только самое нужное.
© Alisa Garaeva



