Метод максимальной экономии не использует расстояния, поэтому укоренение невозможно.
Укоренение в среднюю точку дерева не имеет смысла проводить с деревом, построенным методом UPGMA,
т.к. мы получим ту же точку, т.к. алгоритм UPGMA предполагает молекулярные часы (ультраметричность).
Метод максимальной экономии не использует расстояния, поэтому укоренение невозможно.

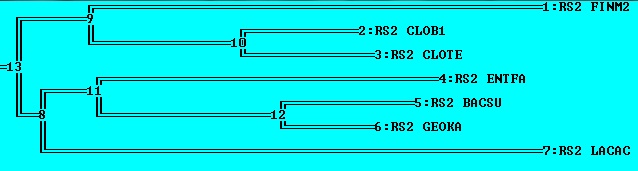
Укоренение является правильным, так как оно совпало с верным деревом. Кроме того, правильно построилась и верхняя ветка {FINM2, CLOB1, CLOTE}.
Нижняя ветка получилась немного неточной: {BACSU,GEOKA} - это верно, но {ENTFA, LACAC} - нет.
Для сравнения правильное дерево:
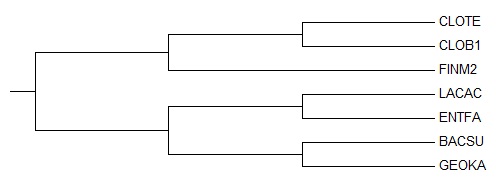
В качестве внешней группы использовался рибосомальный белок RS2 из кишечной палочки (Escherichia coli, ECOLI).
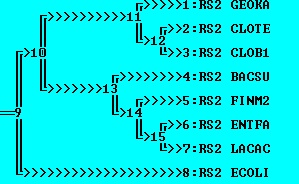
Полученное дерево неправильное. С верным деревом совпали только ветки {CLOB1, CLOTE} и {ENTFA, LACAC}. Очень странно, что BACSU и GEOKA оказались так сильно разнесены друг от друга. Таком образом, алгоритм "внешней группы" сработал очень плохо.
+-----------------------------------------------RS2 FINM2
|
| +-----------------------RS2 GEOKA
| |
| +--61.0-| +-------RS2 CLOB1
| | | +-100.0-|
| | +--90.0-| +-------RS2 CLOTE
| +--82.5-| |
| | | +---------------RS2 LACAC
+-------| |
| +-------------------------------RS2 BACSU
|
+---------------------------------------RS2 ENTFA
Получилось новое дерево, которое опять отличается от верного. Кроме того это дерево сильно отличается от дерева, построенного
fprotpars на исходном выравнивании (см. предыдущий практикум). Ветка {CLOB1, CLOTE} является верной и в сравнении с правильным деревом,
и с точки зрения таксономии, поэтому имеет поддержку 100. Но вот ветка {CLOB1, CLOTE, LACAC} - совершенно неправильная, так как LACAC относится к Bacilli, в отличие
от CLOB1, CLOTE - Clostridia, и эта ветка имеет уже поддержку 90. Совершенно не логично, что FINM2, относящийся так же к Clostridia оказался так далеко. Таким образом, имеем
только одну верную ветвь {CLOB1, CLOTE} с поддержкой 100.
В выходном файле есть данные о ветвях, которые не были включены в консенсусное дерево: правильная ветвь {GEOKA, BACSU} с поддержкой 29, неправильная ветвь {ENTFA, GEOKA, BACSU}
с поддержкой 10,5, неправильная ветвь {GEOKA, BACSU, CLOB1, CLOTE} - 10, неправильная ветвь {GEOKA, CLOB1, CLOTE} - 10, неправильная ветвь {ENTFA, GEOKA, BACSU, CLOB1, CLOTE} - 7.