Реконструкция деревьев по нуклеотидным последовательностям. Анализ деревьев, содержащих паралоги.
Построение дерева по нуклеотидным последовательностям.
| Бактерия
|
AC записи EMBL
|
Координаты рРНК
|
| BACSU
|
AL009126
|
9810..11364
|
| CLOB1
|
CP000726
|
9282..10783
|
| CLOTE
|
AE015927
|
complement(8715..10223)
|
| ENTFA
|
AE016830
|
248466..249987
|
| FINM2
|
AP008971
|
197837..199361
|
| GEOKA
|
BA000043
|
10421..11973
|
| LACAC
|
CP000033
|
59255..60826
|
Для получения указанных последовательностей использовалась команда seqret -sask. У CLOTE не забываем
"обратить" последовательность.
Выравнивание получено программой muscle.
Реконструировала дерево программой fdnaml.
Реконструированное дерево по последовательности РНК малой
субъединицы рибосомы (16S rRNA)
+--GEOKA
+---4
| | +-BACSU
| +--5
| | +--ENTFA
| +--3
| +-----LACAC
|
| +-CLOB1
2---1
| +--CLOTE
|
+------FINM2
|
Правильное дерево
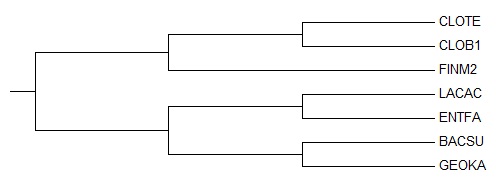
|
Получилось неукорененное дерево, которое очень похоже на правильное.
Нижня ветвь {CLOB1,CLOTE,FINM2} точно правильная, ветвь {LACAC,ENTFA} - тоже.
Отличия с правильным деревом составляют только разнесенные BACSU и GEOKA, но они
находятся не так далеко друг от друга на этом реконструированном дереве.
Таким образом, реконструкция дерева по нуклеотидным последовательностям, как мне показалось, эффективнее и
полученные деревья правильнее по сравнению с деревьями, построенным по белкам (см. предыдущие отчеты).
Построение и анализ дерева, содержащего паралоги.
Сначала нужно найти в выбранных бактериях достоверные гомологи белка CLPX_BACSU.
Для этого воспользуемся файлом proteo.fasta на диске P,
проводим поиск программой BLASTP гомологов (с разумным порогом на E-value, скажем, 0,001) и отбираем по
мнемонике видов только те находки, которые относятся к отобранным мной бактериям.
Получаем последовательности полученных находок, выравниваем программой muscle.
Строим дерево этих гомологов программой fprotpars:
+-----------CLPX_ENTFA
!
+--6 +--CLPX_GEOKA
! ! +-----8
! ! ! +--CLPX_BACSU
! +--7
+-----------------4 ! +--CLPX_CLOB1
! ! +-----5
! ! +--CLPX_CLOTE
! !
! +--------------B0S2N5_FIN
!
+--3 +--HSLU_ENTFA
! ! +----12
! ! ! +--HSLU_LACAC
! ! +----11
! ! ! ! +--CLPY_BACSU
! ! ! +----10
1 +-----------------9 +--HSLU_GEOKA
! !
! ! +--CLPE_BACSU
! +-----------2
! +--CLPC_BACSU
!
+-----------------------------------B0S0E3_FIN
Считая дерево реконструированным верно, можно указать несколько пар ортологов и несколько пар паралогов:
Ортологи:
CLPX_ENTFA, CLPX_BACSU, CLPX_CLOB1, CLPX_CLOTE.
HSLU_ENTFA, HSLU_LACAC, HSLU_GEOKA.
Паралоги:
CLPY_BACSU, CLPE_BACSU, CLPC_BACSU, CLPX_BACSU.
CLPX_GEOKA, HSLU_GEOKA.
CLPX_ENTFA, HSLU_ENTFA.
B0S2N5_FIN, B0S0E3_FIN.
© Alisa Garaeva
